[English] 日本語
 Yorodumi
Yorodumi- PDB-8d3k: Crystal structure of human Apoptosis-Inducing Factor (AIF) comple... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 8d3k | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Title | Crystal structure of human Apoptosis-Inducing Factor (AIF) complexed with 8-fluoro-2-methylquinolin-4-amine | ||||||||||||
 Components Components | (Apoptosis-inducing factor 1, ...) x 2 | ||||||||||||
 Keywords Keywords | OXIDOREDUCTASE / mitochondrial import / oxidative phosphorylation / SAXS | ||||||||||||
| Function / homology |  Function and homology information Function and homology informationOxidoreductases; Acting on NADH or NADPH; With unknown physiological acceptors / mitochondrial disulfide relay system / mitochondrial respiratory chain complex assembly / protein import into mitochondrial intermembrane space / NAD(P)H oxidase H2O2-forming activity / poly-ADP-D-ribose binding / cellular response to aldosterone / positive regulation of necroptotic process / oxidoreductase activity, acting on NAD(P)H / response to L-glutamate ...Oxidoreductases; Acting on NADH or NADPH; With unknown physiological acceptors / mitochondrial disulfide relay system / mitochondrial respiratory chain complex assembly / protein import into mitochondrial intermembrane space / NAD(P)H oxidase H2O2-forming activity / poly-ADP-D-ribose binding / cellular response to aldosterone / positive regulation of necroptotic process / oxidoreductase activity, acting on NAD(P)H / response to L-glutamate / NADH dehydrogenase activity / intrinsic apoptotic signaling pathway in response to endoplasmic reticulum stress / cellular response to nitric oxide / FAD binding / response to ischemia / cellular response to estradiol stimulus / mitochondrial intermembrane space / cellular response to hydrogen peroxide / response to toxic substance / neuron differentiation / positive regulation of neuron apoptotic process / cellular response to hypoxia / mitochondrial inner membrane / protein dimerization activity / positive regulation of apoptotic process / apoptotic process / perinuclear region of cytoplasm / mitochondrion / DNA binding / nucleus / cytosol / cytoplasm Similarity search - Function | ||||||||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | ||||||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2.3 Å MOLECULAR REPLACEMENT / Resolution: 2.3 Å | ||||||||||||
 Authors Authors | Brosey, C.A. / Tainer, J.A. | ||||||||||||
| Funding support |  United States, 3items United States, 3items
| ||||||||||||
 Citation Citation |  Journal: Nat.Chem.Biol. / Year: 2024 Journal: Nat.Chem.Biol. / Year: 2024Title: Chemical screening by time-resolved X-ray scattering to discover allosteric probes. Authors: Brosey, C.A. / Link, T.M. / Shen, R. / Moiani, D. / Burnett, K. / Hura, G.L. / Jones, D.E. / Tainer, J.A. | ||||||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  8d3k.cif.gz 8d3k.cif.gz | 601.2 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb8d3k.ent.gz pdb8d3k.ent.gz | 411.5 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  8d3k.json.gz 8d3k.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Summary document |  8d3k_validation.pdf.gz 8d3k_validation.pdf.gz | 1.5 MB | Display |  wwPDB validaton report wwPDB validaton report |
|---|---|---|---|---|
| Full document |  8d3k_full_validation.pdf.gz 8d3k_full_validation.pdf.gz | 1.5 MB | Display | |
| Data in XML |  8d3k_validation.xml.gz 8d3k_validation.xml.gz | 33.9 KB | Display | |
| Data in CIF |  8d3k_validation.cif.gz 8d3k_validation.cif.gz | 47.3 KB | Display | |
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/d3/8d3k https://data.pdbj.org/pub/pdb/validation_reports/d3/8d3k ftp://data.pdbj.org/pub/pdb/validation_reports/d3/8d3k ftp://data.pdbj.org/pub/pdb/validation_reports/d3/8d3k | HTTPS FTP |
-Related structure data
| Related structure data |  8d3eC  8d3gC  8d3hC  8d3iC  8d3jC  8d3nC  8d3oC  4bv6S S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Unit cell |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Noncrystallographic symmetry (NCS) | NCS domain:
NCS domain segments:
NCS oper: (Code: givenMatrix: (-0.530070383182, -0.845638696056, -0.0626145718284), (-0.847649611898, 0.530410951393, 0.0124240931509), (0.0227051606828, 0.0596608613267, -0.997960448767)Vector: 37. ...NCS oper: (Code: given Matrix: (-0.530070383182, -0.845638696056, -0.0626145718284), Vector: |
- Components
Components
-Apoptosis-inducing factor 1, ... , 2 types, 2 molecules AB
| #1: Protein | Mass: 59502.566 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: AIFM1, AIF, PDCD8 / Production host: Homo sapiens (human) / Gene: AIFM1, AIF, PDCD8 / Production host:  References: UniProt: O95831, Oxidoreductases; Acting on NADH or NADPH; With unknown physiological acceptors |
|---|---|
| #2: Protein | Mass: 59486.566 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: AIFM1, AIF, PDCD8 / Production host: Homo sapiens (human) / Gene: AIFM1, AIF, PDCD8 / Production host:  References: UniProt: O95831, Oxidoreductases; Acting on NADH or NADPH; With unknown physiological acceptors |
-Non-polymers , 5 types, 255 molecules 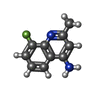








| #3: Chemical | | #4: Chemical | ChemComp-EDO / | #5: Chemical | #6: Chemical | ChemComp-SO4 / #7: Water | ChemComp-HOH / | |
|---|
-Details
| Has ligand of interest | Y |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.66 Å3/Da / Density % sol: 53.9 % |
|---|---|
| Crystal grow | Temperature: 295 K / Method: vapor diffusion, hanging drop / pH: 8.5 / Details: 0.1 M Tris, pH 8.5, 0.36 M Li2SO4, 25-26% PEG3350 |
-Data collection
| Diffraction | Mean temperature: 100 K / Serial crystal experiment: N |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  ALS ALS  / Beamline: 8.3.1 / Wavelength: 1.1159 Å / Beamline: 8.3.1 / Wavelength: 1.1159 Å |
| Detector | Type: DECTRIS PILATUS3 S 6M / Detector: PIXEL / Date: Sep 11, 2019 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1.1159 Å / Relative weight: 1 |
| Reflection | Resolution: 2.3→124.12 Å / Num. obs: 58766 / % possible obs: 99.9 % / Redundancy: 13.2 % / Biso Wilson estimate: 42.22 Å2 / CC1/2: 0.999 / Rmerge(I) obs: 0.076 / Rpim(I) all: 0.022 / Rrim(I) all: 0.079 / Net I/σ(I): 21.6 |
| Reflection shell | Resolution: 2.3→2.36 Å / Redundancy: 13.4 % / Rmerge(I) obs: 0.707 / Mean I/σ(I) obs: 3.5 / Num. unique obs: 4468 / CC1/2: 0.939 / Rpim(I) all: 0.199 / Rrim(I) all: 0.735 / % possible all: 99.8 |
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 4BV6 Resolution: 2.3→71.55 Å / SU ML: 0.2383 / Cross valid method: FREE R-VALUE / σ(F): 1.34 / Phase error: 25.4856 Stereochemistry target values: GeoStd + Monomer Library + CDL v1.2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å / Solvent model: FLAT BULK SOLVENT MODEL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 53.71 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 2.3→71.55 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints NCS | Type: Torsion NCS / Rms dev position: 0.888377043016 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group | Refine-ID: X-RAY DIFFRACTION
|
 Movie
Movie Controller
Controller


 PDBj
PDBj






