+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8bvd | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
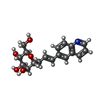
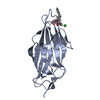
| タイトル | FimH lectin domain in complex with mannose C-linked to quinoline | |||||||||
 要素 要素 | Type 1 fimbrin D-mannose specific adhesin | |||||||||
 キーワード キーワード | CELL ADHESION / Type-1 fimbriae / Escherichia coli / FimH / Adhesin / Lectin / Anti-adhesives / C-linked mannose | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報pilus tip / mechanosensory behavior / cell adhesion involved in single-species biofilm formation / pilus / cell-substrate adhesion / D-mannose binding / host cell membrane / cell adhesion 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.995 Å 分子置換 / 解像度: 2.995 Å | |||||||||
 データ登録者 データ登録者 | Bouckaert, J. / Bridot, C. | |||||||||
| 資金援助 |  フランス, フランス,  カナダ, 2件 カナダ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Pharmaceutics / 年: 2023 ジャーナル: Pharmaceutics / 年: 2023タイトル: Insightful Improvement in the Design of Potent Uropathogenic E. coli FimH Antagonists. 著者: Mousavifar, L. / Sarshar, M. / Bridot, C. / Scribano, D. / Ambrosi, C. / Palamara, A.T. / Vergoten, G. / Roubinet, B. / Landemarre, L. / Bouckaert, J. / Roy, R. #1:  ジャーナル: Molecules / 年: 2017 ジャーナル: Molecules / 年: 2017タイトル: Sites for Dynamic Protein-Carbohydrate Interactions of O- and C-Linked Mannosides on the E. coli FimH Adhesin. 著者: Touaibia, M. / Krammer, E.M. / Shiao, T.C. / Yamakawa, N. / Wang, Q. / Glinschert, A. / Papadopoulos, A. / Mousavifar, L. / Maes, E. / Oscarson, S. / Vergoten, G. / Lensink, M.F. / Roy, R. / Bouckaert, J. #2:  ジャーナル: Molecules / 年: 2018 ジャーナル: Molecules / 年: 2018タイトル: A Novel Integrated Way for Deciphering the Glycan Code for the FimH Lectin. 著者: Dumych, T. / Bridot, C. / Gouin, S.G. / Lensink, M.F. / Paryzhak, S. / Szunerits, S. / Blossey, R. / Bilyy, R. / Bouckaert, J. / Krammer, E.M. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8bvd.cif.gz 8bvd.cif.gz | 144.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8bvd.ent.gz pdb8bvd.ent.gz | 110.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8bvd.json.gz 8bvd.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/bv/8bvd https://data.pdbj.org/pub/pdb/validation_reports/bv/8bvd ftp://data.pdbj.org/pub/pdb/validation_reports/bv/8bvd ftp://data.pdbj.org/pub/pdb/validation_reports/bv/8bvd | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ | |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 単位格子 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Components on special symmetry positions |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域: Beg auth comp-ID: PHE / Beg label comp-ID: PHE / End auth comp-ID: THR / End label comp-ID: THR / Refine code: 1 / Auth asym-ID: A / Label asym-ID: A / Auth seq-ID: 1 - 158 / Label seq-ID: 1 - 158
NCSアンサンブル:
|
- 要素
要素
| #1: タンパク質 | 分子量: 16916.828 Da / 分子数: 4 / 由来タイプ: 組換発現 / 由来: (組換発現)   #2: 化合物 | ChemComp-RLO / ( #3: 水 | ChemComp-HOH / | 研究の焦点であるリガンドがあるか | Y | Has protein modification | Y | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 5.53 Å3/Da / 溶媒含有率: 77.75 % |
|---|---|
| 結晶化 | 温度: 293.15 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 5.5 / 詳細: 3.0M NaCl and 0.1M BIS-TRIS at pH=5.5 / PH範囲: 5.25 - 5.75 |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SOLEIL SOLEIL  / ビームライン: PROXIMA 1 / 波長: 0.978565 Å / ビームライン: PROXIMA 1 / 波長: 0.978565 Å |
| 検出器 | タイプ: DECTRIS EIGER X 16M / 検出器: PIXEL / 日付: 2019年9月22日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.978565 Å / 相対比: 1 |
| 反射 | 解像度: 2.995→49.732 Å / Num. obs: 31057 / % possible obs: 98.6 % / 冗長度: 39.4 % / CC1/2: 0.996 / Rrim(I) all: 0.464 / Net I/σ(I): 12.41 |
| 反射 シェル | 解像度: 3→3.18 Å / 冗長度: 40.2 % / Mean I/σ(I) obs: 1.37 / Num. unique obs: 4900 / CC1/2: 0.615 / Rrim(I) all: 3.422 / % possible all: 98.3 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 / 解像度: 2.995→49.732 Å / Cor.coef. Fo:Fc: 0.881 / Cor.coef. Fo:Fc free: 0.83 / WRfactor Rfree: 0.243 / WRfactor Rwork: 0.206 / SU B: 17.437 / SU ML: 0.302 / Average fsc free: 0.9258 / Average fsc work: 0.9484 / 交差検証法: THROUGHOUT / ESU R: 0.669 / ESU R Free: 0.41 / 詳細: Hydrogens have been added in their riding positions 分子置換 / 解像度: 2.995→49.732 Å / Cor.coef. Fo:Fc: 0.881 / Cor.coef. Fo:Fc free: 0.83 / WRfactor Rfree: 0.243 / WRfactor Rwork: 0.206 / SU B: 17.437 / SU ML: 0.302 / Average fsc free: 0.9258 / Average fsc work: 0.9484 / 交差検証法: THROUGHOUT / ESU R: 0.669 / ESU R Free: 0.41 / 詳細: Hydrogens have been added in their riding positions
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK BULK SOLVENT | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 53.611 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.995→49.732 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj