+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8341 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Architecture of the Yeast Mitochondrial Iron-Sulfur Cluster Assembly Machinery: the Sub-Complex Formed by the Iron Donor, Yfh1, and the Scaffold, Isu1 | |||||||||
 マップデータ マップデータ | Yfh1_Isu1 sub-complex | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Mitochondrial iron-sulfur cluster biogenesis / Mitochondrial protein import / mitochondrial electron transport, succinate to ubiquinone / tRNA wobble uridine modification / iron chaperone activity / iron-sulfur cluster assembly complex / response to iron(II) ion / iron-sulfur cluster assembly / heme biosynthetic process / ferroxidase ...Mitochondrial iron-sulfur cluster biogenesis / Mitochondrial protein import / mitochondrial electron transport, succinate to ubiquinone / tRNA wobble uridine modification / iron chaperone activity / iron-sulfur cluster assembly complex / response to iron(II) ion / iron-sulfur cluster assembly / heme biosynthetic process / ferroxidase / ATPase activator activity / ferroxidase activity / glutathione metabolic process / ferric iron binding / ferrous iron binding / mitochondrial intermembrane space / 2 iron, 2 sulfur cluster binding / iron ion transport / intracellular iron ion homeostasis / response to oxidative stress / mitochondrial inner membrane / mitochondrial matrix / iron ion binding / mitochondrion / zinc ion binding / identical protein binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
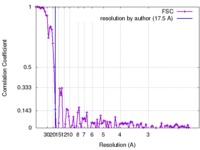
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 17.5 Å | |||||||||
 データ登録者 データ登録者 | Ranatunga W / Gakh O / Galeano BK / Smith IV DY / Soderberg CA / Al-Karadaghi S / Thompson JR / Isaya G | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2016 ジャーナル: J Biol Chem / 年: 2016タイトル: Architecture of the Yeast Mitochondrial Iron-Sulfur Cluster Assembly Machinery: THE SUB-COMPLEX FORMED BY THE IRON DONOR, Yfh1 PROTEIN, AND THE SCAFFOLD, Isu1 PROTEIN. 著者: Wasantha Ranatunga / Oleksandr Gakh / Belinda K Galeano / Douglas Y Smith / Christopher A G Söderberg / Salam Al-Karadaghi / James R Thompson / Grazia Isaya /   要旨: The biosynthesis of Fe-S clusters is a vital process involving the delivery of elemental iron and sulfur to scaffold proteins via molecular interactions that are still poorly defined. We ...The biosynthesis of Fe-S clusters is a vital process involving the delivery of elemental iron and sulfur to scaffold proteins via molecular interactions that are still poorly defined. We reconstituted a stable, functional complex consisting of the iron donor, Yfh1 (yeast frataxin homologue 1), and the Fe-S cluster scaffold, Isu1, with 1:1 stoichiometry, [Yfh1]24·[Isu1]24 Using negative staining transmission EM and single particle analysis, we obtained a three-dimensional reconstruction of this complex at a resolution of ∼17 Å. In addition, via chemical cross-linking, limited proteolysis, and mass spectrometry, we identified protein-protein interaction surfaces within the complex. The data together reveal that [Yfh1]24·[Isu1]24 is a roughly cubic macromolecule consisting of one symmetric Isu1 trimer binding on top of one symmetric Yfh1 trimer at each of its eight vertices. Furthermore, molecular modeling suggests that two subunits of the cysteine desulfurase, Nfs1, may bind symmetrically on top of two adjacent Isu1 trimers in a manner that creates two putative [2Fe-2S] cluster assembly centers. In each center, conserved amino acids known to be involved in sulfur and iron donation by Nfs1 and Yfh1, respectively, are in close proximity to the Fe-S cluster-coordinating residues of Isu1. We suggest that this architecture is suitable to ensure concerted and protected transfer of potentially toxic iron and sulfur atoms to Isu1 during Fe-S cluster assembly. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8341.map.gz emd_8341.map.gz | 11 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8341-v30.xml emd-8341-v30.xml emd-8341.xml emd-8341.xml | 15.7 KB 15.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_8341_fsc.xml emd_8341_fsc.xml | 12 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_8341.png emd_8341.png | 67.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8341 http://ftp.pdbj.org/pub/emdb/structures/EMD-8341 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8341 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8341 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_8341_validation.pdf.gz emd_8341_validation.pdf.gz | 353 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_8341_full_validation.pdf.gz emd_8341_full_validation.pdf.gz | 352.6 KB | 表示 | |
| XML形式データ |  emd_8341_validation.xml.gz emd_8341_validation.xml.gz | 12.2 KB | 表示 | |
| CIF形式データ |  emd_8341_validation.cif.gz emd_8341_validation.cif.gz | 16 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8341 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8341 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8341 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8341 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8341.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8341.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Yfh1_Isu1 sub-complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.034 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Yfh1-Isu1
| 全体 | 名称: Yfh1-Isu1 |
|---|---|
| 要素 |
|
-超分子 #1: Yfh1-Isu1
| 超分子 | 名称: Yfh1-Isu1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: macromolecule comprising 24-mer of Yfh1 and 24-mer of Isu1 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 分子量 | 理論値: 700 KDa |
-分子 #1: Iron sulfur cluster assembly protein 1, mitochondrial
| 分子 | 名称: Iron sulfur cluster assembly protein 1, mitochondrial タイプ: protein_or_peptide / ID: 1 / コピー数: 24 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 15.383872 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSHMSSITKR LYHPKVIEHY THPRNVGSLD KKLPNVGTGL VGAPACGDVM RLQIKVNDST GVIEDVKFKT FGCGSAIASS SYMTELVQG MTLDDAAKIK NTEIAKELSL PPVKLHCSML AEDAIKAAIK DYKSKRNTPT MLS |
-分子 #2: Frataxin homolog, mitochondrial
| 分子 | 名称: Frataxin homolog, mitochondrial / タイプ: protein_or_peptide / ID: 2 / コピー数: 24 / 光学異性体: LEVO / EC番号: ferroxidase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 13.455976 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: VESSTDGQVV PQEVLNLPLE KAHEEADDYL DHLLDSLEEL SEAHPDCIPD VELSHGVMTL EIPAFGTYVI NKQPPNKQIW LASPLSGPN RFDLLNGEWV SLRNGTKLTD ILTEEVEKAI SK |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.12 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.3 構成要素:
| |||||||||
| 染色 | タイプ: NEGATIVE / 材質: uranyl acetate 詳細: Pre-incubated in HN100 buffer, the grid was placed on an 11-microliter drop of protein sample for 1 minute. Excess protein sample was blotted and washed for 3 seconds by placing the grid on a ...詳細: Pre-incubated in HN100 buffer, the grid was placed on an 11-microliter drop of protein sample for 1 minute. Excess protein sample was blotted and washed for 3 seconds by placing the grid on a drop of sterile water. After excess water was blotted, the grid was stained with 1% w/v uranyl acetate for 1 second and 30 seconds by successively placing it on two separate drops of uranyl acetate, with excess stain drawn off after each step. | |||||||||
| グリッド | モデル: carbon-coated, EMS / 材質: COPPER / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE / 詳細: DV-502A instrument, Denton Vacuum Inc. | |||||||||
| 詳細 | The protein complex was prepared by incubating Yfh1 and Isu1 (1:1.5 molar ratio) in HN100 buffer (10 mM HEPES-KOH, pH 7.3, 100 mM NaCl) and purified using Sephacryl S300 gel filtration chromatography. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F30 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 559 / 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 2.8 µm / 最小 デフォーカス(補正後): 0.21 µm / 倍率(補正後): 115000 / 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 2.8 µm / 最小 デフォーカス(公称値): 0.21 µm / 倍率(公称値): 115000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC / ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Tecnai F30 / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-5t0v: |
 ムービー
ムービー コントローラー
コントローラー