+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7bvc | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Mycobacterium smegmatis arabinosyltransferase EmbA-EmbB-AcpM2 in complex with ethambutol | |||||||||||||||
 要素 要素 |
| |||||||||||||||
 キーワード キーワード | TRANSFERASE / Mycobacterium smegmatis / cell wall synthesis / drug target / ethambutol / arabinosyltransferase / EmbA / EmbB / EmbC / acyl carrier protein / arabinogalactan / lipoarabinomannan / drug resistance | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報indolylacetylinositol arabinosyltransferase / indolylacetylinositol arabinosyltransferase activity / arabinosyltransferase activity / Actinobacterium-type cell wall biogenesis / lipid A biosynthetic process / 転移酵素; グリコシル基を移すもの; 五炭糖残基を移すもの / acyl binding / acyl carrier activity / cell wall organization / plasma membrane / cytosol 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) | |||||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||||||||
 データ登録者 データ登録者 | Zhang, L. / Zhao, Y. / Gao, Y. / Wang, Q. / Li, J. / Besra, G.S. / Rao, Z. | |||||||||||||||
| 資金援助 |  中国, 中国,  英国, 4件 英国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Science / 年: 2020 ジャーナル: Science / 年: 2020タイトル: Structures of cell wall arabinosyltransferases with the anti-tuberculosis drug ethambutol. 著者: Lu Zhang / Yao Zhao / Yan Gao / Lijie Wu / Ruogu Gao / Qi Zhang / Yinan Wang / Chengyao Wu / Fangyu Wu / Sudagar S Gurcha / Natacha Veerapen / Sarah M Batt / Wei Zhao / Ling Qin / Xiuna Yang ...著者: Lu Zhang / Yao Zhao / Yan Gao / Lijie Wu / Ruogu Gao / Qi Zhang / Yinan Wang / Chengyao Wu / Fangyu Wu / Sudagar S Gurcha / Natacha Veerapen / Sarah M Batt / Wei Zhao / Ling Qin / Xiuna Yang / Manfu Wang / Yan Zhu / Bing Zhang / Lijun Bi / Xian'en Zhang / Haitao Yang / Luke W Guddat / Wenqing Xu / Quan Wang / Jun Li / Gurdyal S Besra / Zihe Rao /    要旨: The arabinosyltransferases EmbA, EmbB, and EmbC are involved in cell wall synthesis and are recognized as targets for the anti-tuberculosis drug ethambutol. In this study, we determined cryo- ...The arabinosyltransferases EmbA, EmbB, and EmbC are involved in cell wall synthesis and are recognized as targets for the anti-tuberculosis drug ethambutol. In this study, we determined cryo-electron microscopy and x-ray crystal structures of mycobacterial EmbA-EmbB and EmbC-EmbC complexes in the presence of their glycosyl donor and acceptor substrates and with ethambutol. These structures show how the donor and acceptor substrates bind in the active site and how ethambutol inhibits arabinosyltransferases by binding to the same site as both substrates in EmbB and EmbC. Most drug-resistant mutations are located near the ethambutol binding site. Collectively, our work provides a structural basis for understanding the biochemical function and inhibition of arabinosyltransferases and the development of new anti-tuberculosis agents. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7bvc.cif.gz 7bvc.cif.gz | 388.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7bvc.ent.gz pdb7bvc.ent.gz | 318.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7bvc.json.gz 7bvc.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7bvc_validation.pdf.gz 7bvc_validation.pdf.gz | 1.4 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7bvc_full_validation.pdf.gz 7bvc_full_validation.pdf.gz | 1.5 MB | 表示 | |
| XML形式データ |  7bvc_validation.xml.gz 7bvc_validation.xml.gz | 73.7 KB | 表示 | |
| CIF形式データ |  7bvc_validation.cif.gz 7bvc_validation.cif.gz | 107.6 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/bv/7bvc https://data.pdbj.org/pub/pdb/validation_reports/bv/7bvc ftp://data.pdbj.org/pub/pdb/validation_reports/bv/7bvc ftp://data.pdbj.org/pub/pdb/validation_reports/bv/7bvc | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
| 詳細 | The complete assembly should be tetramer. There should be 2 molecules of AcpM, however, only one was modeled due to low density. |
- 要素
要素
-Integral membrane indolylacetylinositol arabinosyltransferase ... , 2種, 2分子 AB
| #1: タンパク質 | 分子量: 116561.898 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア)遺伝子: embA 発現宿主:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア)参照: UniProt: A0R613, 転移酵素; グリコシル基を移すもの; 五炭糖残基を移すもの |
|---|---|
| #2: タンパク質 | 分子量: 119077.672 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア)遺伝子: embB, MSMEI_6221 発現宿主:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア)参照: UniProt: I7GAQ2, UniProt: A0R614*PLUS, indolylacetylinositol arabinosyltransferase |
-タンパク質 , 1種, 1分子 P
| #3: タンパク質 | 分子量: 10743.876 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア)参照: UniProt: A0R0B3 |
|---|
-非ポリマー , 6種, 8分子 
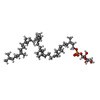

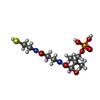

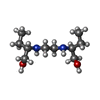





| #4: 化合物 | | #5: 化合物 | ChemComp-F8L / [( | #6: 化合物 | #7: 化合物 | ChemComp-PNS / | #8: 化合物 | ChemComp-PO4 / | #9: 化合物 | ChemComp-95E / | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 由来(天然) |
| ||||||||||||||||||||||||
| 由来(組換発現) | 生物種:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) | ||||||||||||||||||||||||
| 緩衝液 | pH: 7.4 | ||||||||||||||||||||||||
| 緩衝液成分 |
| ||||||||||||||||||||||||
| 試料 | 濃度: 5 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES / 詳細: The sample was monodisperse. | ||||||||||||||||||||||||
| 試料支持 | グリッドの材料: COPPER / グリッドのタイプ: Quantifoil R0.6/1 | ||||||||||||||||||||||||
| 急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 29000 X / 最大 デフォーカス(公称値): 2000 nm / 最小 デフォーカス(公称値): 1000 nm / Calibrated defocus min: 1000 nm / 最大 デフォーカス(補正後): 2000 nm / Cs: 2.7 mm / C2レンズ絞り径: 50 µm / アライメント法: COMA FREE |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER 最高温度: 78.6 K / 最低温度: 78.5 K / Residual tilt: 10 mradians |
| 撮影 | 平均露光時間: 2 sec. / 電子線照射量: 50 e/Å2 / フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 2 / 実像数: 5100 |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 画像処理 | 詳細: The selected images were normalized. | ||||||||||||||||||||||||||||||||||||
| CTF補正 | 詳細: The CTF correction was done by patch CTF correction. タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 1855947 | ||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性: C1 (非対称) | ||||||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 2.9 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 227206 / アルゴリズム: FOURIER SPACE / クラス平均像の数: 1 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | B value: 62.01 / プロトコル: RIGID BODY FIT / Target criteria: correlation coefficient | ||||||||||||||||||||||||||||||||||||
| 精密化 | 立体化学のターゲット値: GeoStd + Monomer Library + CDL v1.2 | ||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 61.93 Å2 | ||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー


















 PDBj
PDBj







