+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7568 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Glutaraldehyde-treated BG505 SOSIP.664 Env in complex with PGV04 Fab | |||||||||||||||
 マップデータ マップデータ | Glutaraldehyde-treated BG505 SOSIP.664 Env in complex with PGV04 Fab | |||||||||||||||
 試料 試料 |
| |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane / viral envelope ...positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity / plasma membrane 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||
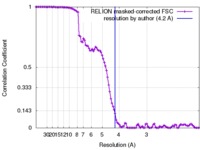
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.2 Å | |||||||||||||||
 データ登録者 データ登録者 | Pallesen J / de Val N / Ward AB | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: PLoS Pathog / 年: 2018 ジャーナル: PLoS Pathog / 年: 2018タイトル: Structural and immunologic correlates of chemically stabilized HIV-1 envelope glycoproteins. 著者: Torben Schiffner / Jesper Pallesen / Rebecca A Russell / Jonathan Dodd / Natalia de Val / Celia C LaBranche / David Montefiori / Georgia D Tomaras / Xiaoying Shen / Scarlett L Harris / Amin E ...著者: Torben Schiffner / Jesper Pallesen / Rebecca A Russell / Jonathan Dodd / Natalia de Val / Celia C LaBranche / David Montefiori / Georgia D Tomaras / Xiaoying Shen / Scarlett L Harris / Amin E Moghaddam / Oleksandr Kalyuzhniy / Rogier W Sanders / Laura E McCoy / John P Moore / Andrew B Ward / Quentin J Sattentau /    要旨: Inducing broad spectrum neutralizing antibodies against challenging pathogens such as HIV-1 is a major vaccine design goal, but may be hindered by conformational instability within viral envelope ...Inducing broad spectrum neutralizing antibodies against challenging pathogens such as HIV-1 is a major vaccine design goal, but may be hindered by conformational instability within viral envelope glycoproteins (Env). Chemical cross-linking is widely used for vaccine antigen stabilization, but how this process affects structure, antigenicity and immunogenicity is poorly understood and its use remains entirely empirical. We have solved the first cryo-EM structure of a cross-linked vaccine antigen. The 4.2 Å structure of HIV-1 BG505 SOSIP soluble recombinant Env in complex with a CD4 binding site-specific broadly neutralizing antibody (bNAb) Fab fragment reveals how cross-linking affects key properties of the trimer. We observed density corresponding to highly specific glutaraldehyde (GLA) cross-links between gp120 monomers at the trimer apex and between gp120 and gp41 at the trimer interface that had strikingly little impact on overall trimer conformation, but critically enhanced trimer stability and improved Env antigenicity. Cross-links were also observed within gp120 at sites associated with the N241/N289 glycan hole that locally modified trimer antigenicity. In immunogenicity studies, the neutralizing antibody response to cross-linked trimers showed modest but significantly greater breadth against a global panel of difficult-to-neutralize Tier-2 heterologous viruses. Moreover, the specificity of autologous Tier-2 neutralization was modified away from the N241/N289 glycan hole, implying a novel specificity. Finally, we have investigated for the first time T helper cell responses to next-generation soluble trimers, and report on vaccine-relevant immunodominant responses to epitopes within BG505 that are modified by cross-linking. Elucidation of the structural correlates of a cross-linked viral glycoprotein will allow more rational use of this methodology for vaccine design, and reveals a strategy with promise for eliciting neutralizing antibodies needed for an effective HIV-1 vaccine. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7568.map.gz emd_7568.map.gz | 96.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7568-v30.xml emd-7568-v30.xml emd-7568.xml emd-7568.xml | 23.1 KB 23.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_7568_fsc.xml emd_7568_fsc.xml | 10.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_7568.png emd_7568.png | 86.2 KB | ||
| その他 |  emd_7568_half_map_1.map.gz emd_7568_half_map_1.map.gz emd_7568_half_map_2.map.gz emd_7568_half_map_2.map.gz | 80.6 MB 80.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7568 http://ftp.pdbj.org/pub/emdb/structures/EMD-7568 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7568 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7568 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_7568_validation.pdf.gz emd_7568_validation.pdf.gz | 904.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_7568_full_validation.pdf.gz emd_7568_full_validation.pdf.gz | 904 KB | 表示 | |
| XML形式データ |  emd_7568_validation.xml.gz emd_7568_validation.xml.gz | 17.5 KB | 表示 | |
| CIF形式データ |  emd_7568_validation.cif.gz emd_7568_validation.cif.gz | 23.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7568 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7568 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7568 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7568 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7568.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7568.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Glutaraldehyde-treated BG505 SOSIP.664 Env in complex with PGV04 Fab | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.02 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: Glutaraldehyde-treated BG505 SOSIP.664 Env in complex with PGV04 Fab
| ファイル | emd_7568_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Glutaraldehyde-treated BG505 SOSIP.664 Env in complex with PGV04 Fab | ||||||||||||
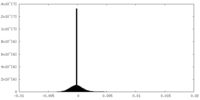
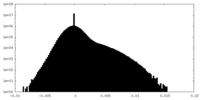
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Glutaraldehyde-treated BG505 SOSIP.664 Env in complex with PGV04 Fab
| ファイル | emd_7568_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Glutaraldehyde-treated BG505 SOSIP.664 Env in complex with PGV04 Fab | ||||||||||||
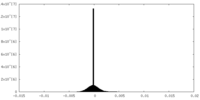
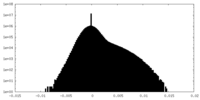
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Glutaraldehyde-treated BG505 SOSIP.664 Env in complex with PGV04 Fab
| 全体 | 名称: Glutaraldehyde-treated BG505 SOSIP.664 Env in complex with PGV04 Fab |
|---|---|
| 要素 |
|
-超分子 #1: Glutaraldehyde-treated BG505 SOSIP.664 Env in complex with PGV04 Fab
| 超分子 | 名称: Glutaraldehyde-treated BG505 SOSIP.664 Env in complex with PGV04 Fab タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 550 KDa |
-分子 #1: Envelope glycoprotein gp160
| 分子 | 名称: Envelope glycoprotein gp160 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 53.950172 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: ENLWVTVYYG VPVWKDAETT LFCASDAKAY ETEKHNVWAT HACVPTDPNP QEIHLENVTE EFNMWKNNMV EQMHTDIISL WDQSLKPCV KLTPLCVTLQ CTNVTNAITD DMRGELKNCS FNMTTELRDK KQKVYSLFYR LDVVQINENQ GNRSNNSNKE Y RLINCNTS ...文字列: ENLWVTVYYG VPVWKDAETT LFCASDAKAY ETEKHNVWAT HACVPTDPNP QEIHLENVTE EFNMWKNNMV EQMHTDIISL WDQSLKPCV KLTPLCVTLQ CTNVTNAITD DMRGELKNCS FNMTTELRDK KQKVYSLFYR LDVVQINENQ GNRSNNSNKE Y RLINCNTS AITQACPKVS FEPIPIHYCA PAGFAILKCK DKKFNGTGPC PSVSTVQCTH GIKPVVSTQL LLNGSLAEEE VM IRSENIT NNAKNILVQF NTPVQINCTR PNNNTRKSIR IGPGQAFYAT GDIIGDIRQA HCNVSKATWN ETLGKVVKQL RKH FGNNTI IRFANSSGGD LEVTTHSFNC GGEFFYCNTS GLFNSTWISN TSVQGSNSTG SNDSITLPCR IKQIINMWQR IGQA MYAPP IQGVIRCVSN ITGLILTRDG GSTNSTTETF RPGGGDMRDN WRSELYKYKV VKIEPLGVAP TRCKRRVVGR RRRRR |
-分子 #2: Envelope glycoprotein gp160
| 分子 | 名称: Envelope glycoprotein gp160 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 17.146482 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AVGIGAVFLG FLGAAGSTMG AASMTLTVQA RNLLSGIVQQ QSNLLRAPEA QQHLLKLTVW GIKQLQARVL AVERYLRDQQ LLGIWGCSG KLICCTNVPW NSSWSNRNLS EIWDNMTWLQ WDKEISNYTQ IIYGLLEESQ NQQEKNEQDL LALD |
-分子 #3: PGV04 VH
| 分子 | 名称: PGV04 VH / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 24.644771 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVQLVQSGSG VKKPGASVRV SCWTSEDIFE RTELIHWVRQ APGQGLEWIG WVKTVTGAVN FGSPDFRQRV SLTRDRDLFT AHMDIRGLT QGDTATYFCA RQKFYTGGQG WYFDLWGRGT LIVVSSASTK GPSVFPLAPS SKSTSGGTAA LGCLVKDYFP E PVTVSWNS ...文字列: QVQLVQSGSG VKKPGASVRV SCWTSEDIFE RTELIHWVRQ APGQGLEWIG WVKTVTGAVN FGSPDFRQRV SLTRDRDLFT AHMDIRGLT QGDTATYFCA RQKFYTGGQG WYFDLWGRGT LIVVSSASTK GPSVFPLAPS SKSTSGGTAA LGCLVKDYFP E PVTVSWNS GALTSGVHTF PAVLQSSGLY SLSSVVTVPS SSLGTQTYIC NVNHKPSNTK VDKKVEPKSC |
-分子 #4: PGV04 VL
| 分子 | 名称: PGV04 VL / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.073822 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: EIVLTQSPGT LSLSPGETAS LSCTAASYGH MTWYQKKPGQ PPKLLIFATS KRASGIPDRF SGSQFGKQYT LTITRMEPED FARYYCQQL EFFGQGTRLE IRRTVAAPSV FIFPPSDEQL KSGTASVVCL LNNFYPREAK VQWKVDNALQ SGNSQESVTE Q DSKDSTYS ...文字列: EIVLTQSPGT LSLSPGETAS LSCTAASYGH MTWYQKKPGQ PPKLLIFATS KRASGIPDRF SGSQFGKQYT LTITRMEPED FARYYCQQL EFFGQGTRLE IRRTVAAPSV FIFPPSDEQL KSGTASVVCL LNNFYPREAK VQWKVDNALQ SGNSQESVTE Q DSKDSTYS LSSTLTLSKA DYEKHKVYAC EVTHQGLSSP VTKSFNRGEC |
-分子 #9: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 9 / コピー数: 18 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.67 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 構成要素 - 名称: TBS / 詳細: 50 mM Tris, 150 mM NaCl, 0.3 mM DDM. |
| グリッド | モデル: C-flat-2/2 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 雰囲気: OTHER |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 297 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3710 pixel / デジタル化 - サイズ - 縦: 3838 pixel / デジタル化 - サンプリング間隔: 5.0 µm / デジタル化 - 画像ごとのフレーム数: 1-35 / 撮影したグリッド数: 1 / 実像数: 1329 / 平均露光時間: 7.0 sec. / 平均電子線量: 66.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 29000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: OTHER |
|---|---|
| 得られたモデル |  PDB-6crq: |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X