+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7446 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the DASH/Dam1 complex shows its role at the yeast kinetochore-microtubule interface (symmetric reconstruction) | |||||||||
 マップデータ マップデータ | DASH/Dam1c symmetric reconstruction | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Chaetomium thermophilum (菌類) Chaetomium thermophilum (菌類) | |||||||||
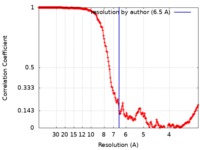
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.5 Å | |||||||||
 データ登録者 データ登録者 | Jenni S / Harrison SC | |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2018 ジャーナル: Science / 年: 2018タイトル: Structure of the DASH/Dam1 complex shows its role at the yeast kinetochore-microtubule interface. 著者: Simon Jenni / Stephen C Harrison /  要旨: Kinetochores connect mitotic-spindle microtubules with chromosomes, allowing microtubule depolymerization to pull chromosomes apart during anaphase while resisting detachment as the microtubule ...Kinetochores connect mitotic-spindle microtubules with chromosomes, allowing microtubule depolymerization to pull chromosomes apart during anaphase while resisting detachment as the microtubule shortens. The heterodecameric DASH/Dam1 complex (DASH/Dam1c), an essential component of yeast kinetochores, assembles into a microtubule-encircling ring. The ring associates with rodlike Ndc80 complexes to organize the kinetochore-microtubule interface. We report the cryo-electron microscopy structure (at ~4.5-angstrom resolution) of a DASH/Dam1c ring and a molecular model of its ordered components, validated by evolutionary direct-coupling analysis. Integrating this structure with that of the Ndc80 complex and with published interaction data yields a molecular picture of kinetochore-microtubule attachment, including how flexible, C-terminal extensions of DASH/Dam1c subunits project and contact widely separated sites on the Ndc80 complex rod and how phosphorylation at previously identified sites might regulate kinetochore assembly. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7446.map.gz emd_7446.map.gz | 464.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7446-v30.xml emd-7446-v30.xml emd-7446.xml emd-7446.xml | 24 KB 24 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_7446_fsc.xml emd_7446_fsc.xml | 21 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_7446.png emd_7446.png | 156.8 KB | ||
| マスクデータ |  emd_7446_msk_1.map emd_7446_msk_1.map | 512 MB |  マスクマップ マスクマップ | |
| その他 |  emd_7446_half_map_1.map.gz emd_7446_half_map_1.map.gz emd_7446_half_map_2.map.gz emd_7446_half_map_2.map.gz | 154.4 MB 154.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7446 http://ftp.pdbj.org/pub/emdb/structures/EMD-7446 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7446 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7446 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7446.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7446.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | DASH/Dam1c symmetric reconstruction | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.64 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
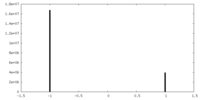
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_7446_msk_1.map emd_7446_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
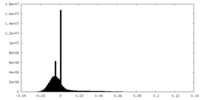
| 密度ヒストグラム |
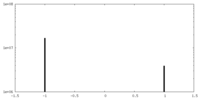
-ハーフマップ: Half-map 1 (not masked, not filtered)
| ファイル | emd_7446_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 1 (not masked, not filtered) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
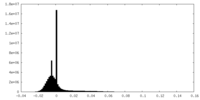
| 密度ヒストグラム |
-ハーフマップ: Half-map 2 (not masked, not filtered)
| ファイル | emd_7446_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 2 (not masked, not filtered) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : DASH/Dam1 complex
+超分子 #1: DASH/Dam1 complex
+分子 #1: Ask1
+分子 #2: Dad1
+分子 #3: Dad2
+分子 #4: Dad3
+分子 #5: Dad4
+分子 #6: Dam1
+分子 #7: Duo1
+分子 #8: Hsk3
+分子 #9: Spc19
+分子 #10: Spc34
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 濃度 | 2.6 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: C-flat-1.2/1.3 4C / 材質: COPPER / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 89 % / チャンバー内温度: 293 K / 装置: GATAN CRYOPLUNGE 3 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 撮影したグリッド数: 1 / 実像数: 2723 / 平均電子線量: 1.115 e/Å2 詳細: Movies collected: 50 frames with 1.115 e/A2/per frame |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 30488 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X