+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6742 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of unstacked C2S2M2-type PSII-LHCII supercomplex from Pisum sativum | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Photosystem II / PSII-LHCII / C2S2M2 / Supercomplex / MEMBRANE PROTEIN | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報photosynthesis, light harvesting in photosystem II / PSII associated light-harvesting complex II / response to high light intensity / plastoglobule / regulation of stomatal movement / response to abscisic acid / photosynthesis, light harvesting / photosynthesis, light harvesting in photosystem I / photosystem II oxygen evolving complex / photosystem II assembly ...photosynthesis, light harvesting in photosystem II / PSII associated light-harvesting complex II / response to high light intensity / plastoglobule / regulation of stomatal movement / response to abscisic acid / photosynthesis, light harvesting / photosynthesis, light harvesting in photosystem I / photosystem II oxygen evolving complex / photosystem II assembly / oxygen evolving activity / photosystem II stabilization / photosystem II reaction center / photosystem II / chloroplast envelope / oxidoreductase activity, acting on diphenols and related substances as donors, oxygen as acceptor / photosystem I / photosynthetic electron transport chain / response to herbicide / photosystem II / photosynthesis, light reaction / chloroplast thylakoid membrane / phosphate ion binding / photosynthetic electron transport in photosystem II / chlorophyll binding / electron transporter, transferring electrons within the cyclic electron transport pathway of photosynthesis activity / response to light stimulus / photosynthesis / electron transfer activity / protein stabilization / iron ion binding / protein domain specific binding / heme binding / metal ion binding 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  | |||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||||||||
 データ登録者 データ登録者 | Su XD / Ma J | |||||||||||||||
| 資金援助 |  中国, 4件 中国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Science / 年: 2017 ジャーナル: Science / 年: 2017タイトル: Structure and assembly mechanism of plant CSM-type PSII-LHCII supercomplex. 著者: Xiaodong Su / Jun Ma / Xuepeng Wei / Peng Cao / Dongjie Zhu / Wenrui Chang / Zhenfeng Liu / Xinzheng Zhang / Mei Li /  要旨: In plants, the photosynthetic machinery photosystem II (PSII) consists of a core complex associated with variable numbers of light-harvesting complexes II (LHCIIs). The supercomplex, comprising a ...In plants, the photosynthetic machinery photosystem II (PSII) consists of a core complex associated with variable numbers of light-harvesting complexes II (LHCIIs). The supercomplex, comprising a dimeric core and two strongly bound and two moderately bound LHCIIs (CSM), is the dominant form in plants acclimated to limited light. Here we report cryo-electron microscopy structures of two forms of CSM (termed stacked and unstacked) from at 2.7- and 3.2-angstrom resolution, respectively. In each CSM, the moderately bound LHCII assembles specifically with a peripheral antenna complex CP24-CP29 heterodimer and the strongly bound LHCII, to establish a pigment network that facilitates light harvesting at the periphery and energy transfer into the core. The high mobility of peripheral antennae, including the moderately bound LHCII and CP24, provides insights into functional regulation of plant PSII. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6742.map.gz emd_6742.map.gz | 95.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6742-v30.xml emd-6742-v30.xml emd-6742.xml emd-6742.xml | 40 KB 40 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_6742.png emd_6742.png | 266 KB | ||
| Filedesc metadata |  emd-6742.cif.gz emd-6742.cif.gz | 9.5 KB | ||
| その他 |  emd_6742_additional.map.gz emd_6742_additional.map.gz | 11.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6742 http://ftp.pdbj.org/pub/emdb/structures/EMD-6742 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6742 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6742 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6742_validation.pdf.gz emd_6742_validation.pdf.gz | 696.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6742_full_validation.pdf.gz emd_6742_full_validation.pdf.gz | 695.7 KB | 表示 | |
| XML形式データ |  emd_6742_validation.xml.gz emd_6742_validation.xml.gz | 6.3 KB | 表示 | |
| CIF形式データ |  emd_6742_validation.cif.gz emd_6742_validation.cif.gz | 7.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6742 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6742 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6742 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6742 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6742.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6742.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
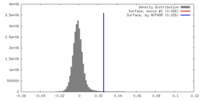
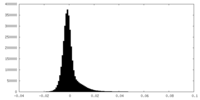
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: #1
| ファイル | emd_6742_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
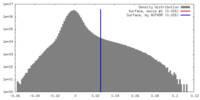
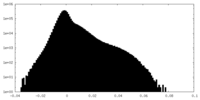
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : C2S2M2-type PSII-LHCII supercomplex from Pisum sativum
+超分子 #1: C2S2M2-type PSII-LHCII supercomplex from Pisum sativum
+分子 #1: Chlorophyll a-b binding protein 8, chloroplastic
+分子 #2: Chlorophyll a-b binding protein, chloroplastic
+分子 #3: Light harvesting chlorophyll a/b-binding protein Lhcb6, CP24
+分子 #4: Photosystem II protein D1
+分子 #5: Photosystem II CP47 reaction center protein
+分子 #6: Photosystem II CP43 reaction center protein
+分子 #7: Photosystem II D2 protein
+分子 #8: Cytochrome b559 subunit alpha
+分子 #9: Cytochrome b559 subunit beta, PsbF
+分子 #10: Photosystem II reaction center protein H
+分子 #11: Photosystem II reaction center protein I, PsbI
+分子 #12: Photosystem II reaction center protein J
+分子 #13: Photosystem II reaction center protein K
+分子 #14: Photosystem II reaction center protein L
+分子 #15: Photosystem II reaction center protein M
+分子 #16: Oxygen-evolving enhancer protein 1, chloroplastic
+分子 #17: Light harvesting chlorophyll a/b-binding protein Lhcb4, CP29
+分子 #18: Light harvesting chlorophyll a/b-binding protein Lhcb5, CP26
+分子 #19: Photosystem II reaction center protein T
+分子 #20: Photosystem II luminal extrinsic protein Tn, PsbTn
+分子 #21: Photosystem II reaction center protein W, PSBW
+分子 #22: Photosystem II reaction center protein X
+分子 #23: Photosystem II reaction center protein Z
+分子 #24: CHLOROPHYLL B
+分子 #25: CHLOROPHYLL A
+分子 #26: (3R,3'R,6S)-4,5-DIDEHYDRO-5,6-DIHYDRO-BETA,BETA-CAROTENE-3,3'-DIOL
+分子 #27: (3S,5R,6S,3'S,5'R,6'S)-5,6,5',6'-DIEPOXY-5,6,5',6'- TETRAHYDRO-BE...
+分子 #28: (1R,3R)-6-{(3E,5E,7E,9E,11E,13E,15E,17E)-18-[(1S,4R,6R)-4-HYDROXY...
+分子 #29: 1,2-DIPALMITOYL-PHOSPHATIDYL-GLYCEROLE
+分子 #30: BETA-CAROTENE
+分子 #31: CA-MN4-O5 CLUSTER
+分子 #32: FE (II) ION
+分子 #33: PHEOPHYTIN A
+分子 #34: 1,2-DI-O-ACYL-3-O-[6-DEOXY-6-SULFO-ALPHA-D-GLUCOPYRANOSYL]-SN-GLYCEROL
+分子 #35: 1,2-DISTEAROYL-MONOGALACTOSYL-DIGLYCERIDE
+分子 #36: 2,3-DIMETHYL-5-(3,7,11,15,19,23,27,31,35-NONAMETHYL-2,6,10,14,18,...
+分子 #37: DIGALACTOSYL DIACYL GLYCEROL (DGDG)
+分子 #38: BICARBONATE ION
+分子 #39: PROTOPORPHYRIN IX CONTAINING FE
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.0 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C2 (2回回転対称) / 解像度のタイプ: BY AUTHOR / 解像度: 3.2 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 50237 |
| 初期 角度割当 | タイプ: PROJECTION MATCHING |
| 最終 角度割当 | タイプ: PROJECTION MATCHING |
-原子モデル構築 1
| 精密化 | 空間: REAL |
|---|---|
| 得られたモデル |  PDB-5xnm: |
 ムービー
ムービー コントローラー
コントローラー





















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)