+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6639 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-electron microscopy of a deletion mutant (d385-399) of Flock House virus VLP | |||||||||
 マップデータ マップデータ | Reconstruction of deletion mutant (d385-399) Flock House virus VLP | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Flock House virus / VLP / gamma | |||||||||
| 生物種 |  Flock house virus (ウイルス) Flock house virus (ウイルス) | |||||||||
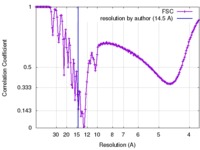
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 14.5 Å | |||||||||
 データ登録者 データ登録者 | Bajaj S / Dey D / Kumar M / Banerjee M | |||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2016 ジャーナル: J Mol Biol / 年: 2016タイトル: Non-Enveloped Virus Entry: Structural Determinants and Mechanism of Functioning of a Viral Lytic Peptide. 著者: Saumya Bajaj / Debajit Dey / Rohan Bhukar / Mohit Kumar / Manidipa Banerjee /  要旨: In the absence of lipid envelopes and associated fusion proteins, non-enveloped viruses employ membrane lytic peptides to breach the limiting membranes of host cells. Although several of these lytic ...In the absence of lipid envelopes and associated fusion proteins, non-enveloped viruses employ membrane lytic peptides to breach the limiting membranes of host cells. Although several of these lytic peptides have been identified and characterized, their manner of deployment and interaction with host membranes remains unclear in most cases. We are using the gamma peptide of Flock House Virus (FHV), a model non-enveloped virus, to understand the mechanistic details of non-enveloped virus interaction with host cell membranes. We utilized a combination of biophysical assays, molecular dynamics simulation studies, and single-particle cryo-electron microscopy to elucidate the functional and structural determinants for membrane penetration by gamma in context of the FHV capsid. Although the amphipathic, helical N-terminal region of gamma (γ1) was previously thought to be the membrane-penetrating module, with the C-terminal region having a supporting role in correct structural positioning of γ1, we demonstrate that the C terminus of gamma directly participates in membrane penetration. Our studies suggest that full-length gamma, including the hydrophobic C terminus, forms an alpha-helical hairpin motif, and any disruption in this motif drastically reduces its functionality, in spite of the correct positioning of amphipathic γ1 in the virus capsid. Taken together, our data suggest that the most effective module for membrane disruption is a pentameric unit of full-length gamma, released from the virus, which associates with membranes via both N- and C-terminal ends. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6639.map.gz emd_6639.map.gz | 47.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6639-v30.xml emd-6639-v30.xml emd-6639.xml emd-6639.xml | 10.5 KB 10.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_6639_fsc.xml emd_6639_fsc.xml | 15.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_6639.tif emd_6639.tif | 2.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6639 http://ftp.pdbj.org/pub/emdb/structures/EMD-6639 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6639 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6639 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6639_validation.pdf.gz emd_6639_validation.pdf.gz | 78.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6639_full_validation.pdf.gz emd_6639_full_validation.pdf.gz | 77.2 KB | 表示 | |
| XML形式データ |  emd_6639_validation.xml.gz emd_6639_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6639 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6639 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6639 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6639 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6639.map.gz / 形式: CCP4 / 大きさ: 210.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6639.map.gz / 形式: CCP4 / 大きさ: 210.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of deletion mutant (d385-399) Flock House virus VLP | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Virus-like particle of deletion mutant (d385-399) of Flock House virus
| 全体 | 名称: Virus-like particle of deletion mutant (d385-399) of Flock House virus |
|---|---|
| 要素 |
|
-超分子 #1000: Virus-like particle of deletion mutant (d385-399) of Flock House virus
| 超分子 | 名称: Virus-like particle of deletion mutant (d385-399) of Flock House virus タイプ: sample / ID: 1000 集合状態: 180 molecules of capsid protein make up one particle Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 9 MDa |
-超分子 #1: Flock house virus
| 超分子 | 名称: Flock house virus / タイプ: virus / ID: 1 詳細: Residues 385-399 of the capsid protein have been deleted NCBI-ID: 12287 / 生物種: Flock house virus / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Costelytra zealandica (甲虫) / 別称: INVERTEBRATES Costelytra zealandica (甲虫) / 別称: INVERTEBRATES |
| Host system | 生物種:  組換細胞: Sf21 / 組換プラスミド: pBacPAK9 |
| 分子量 | 理論値: 9 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: Alpha / 直径: 336 Å / T番号(三角分割数): 3 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: 50 mM HEPES, 5mM CaCl2 |
| グリッド | 詳細: 300 mesh copper, Quantifoil R2/2 grid |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 77 K / 装置: FEI VITROBOT MARK IV 手法: Blot for 3 seconds at a blot force of 0 before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 温度 | 最低: 77 K / 最高: 97 K / 平均: 94 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 80,000x magnification |
| 日付 | 2016年1月8日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: FEI EAGLE (4k x 4k) / 実像数: 1199 / 平均電子線量: 10 e/Å2 / ビット/ピクセル: 2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 1.2 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 80000 |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)