+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of a gp140 SpyTag-SpyCatcher mi3 nanoparticle including mi3 density only. | |||||||||
 マップデータ マップデータ | CAP255-gp140 SpyTag-SpyCatcher mi3 masked | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Vaccine / HIV-1 / mi3 / Nanoparticle / VLP / IMMUNE SYSTEM | |||||||||
| 機能・相同性 | KDPG/KHG aldolase / KDPG and KHG aldolase / Aldolase-type TIM barrel / lyase activity / 2-dehydro-3-deoxyphosphogluconate aldolase/4-hydroxy-2-oxoglutarate aldolase 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Thermotoga maritima (バクテリア) Thermotoga maritima (バクテリア) | |||||||||
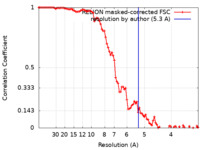
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 5.3 Å | |||||||||
 データ登録者 データ登録者 | Woodward JD / Malebo K / Chapman R | |||||||||
| 資金援助 |  南アフリカ, 南アフリカ,  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Vaccines (Basel) / 年: 2024 ジャーナル: Vaccines (Basel) / 年: 2024タイトル: Development of a Two-Component Nanoparticle Vaccine Displaying an HIV-1 Envelope Glycoprotein that Elicits Tier 2 Neutralising Antibodies. 著者: Kegomoditswe Malebo / Jeremy Woodward / Phindile Ximba / Qiniso Mkhize / Sanele Cingo / Thandeka Moyo-Gwete / Penny L Moore / Anna-Lise Williamson / Rosamund Chapman /  要旨: Despite treatment and other interventions, an effective prophylactic HIV vaccine is still an essential goal in the control of HIV. Inducing robust and long-lasting antibody responses is one of the ...Despite treatment and other interventions, an effective prophylactic HIV vaccine is still an essential goal in the control of HIV. Inducing robust and long-lasting antibody responses is one of the main targets of an HIV vaccine. The delivery of HIV envelope glycoproteins (Env) using nanoparticle (NP) platforms has been shown to elicit better immunogenicity than soluble HIV Env. In this paper, we describe the development of a nanoparticle-based vaccine decorated with HIV Env using the SpyCatcher/SpyTag system. The Env utilised in this study, CAP255, was derived from a transmitted founder virus isolated from a patient who developed broadly neutralising antibodies. Negative stain and cryo-electron microscopy analyses confirmed the assembly and stability of the mi3 into uniform icosahedral NPs surrounded by regularly spaced CAP255 gp140 Env trimers. A three-dimensional reconstruction of CAP255 gp140 SpyTag-SpyCatcher mi3 clearly showed Env trimers projecting from the centre of each of the pentagonal dodecahedral faces of the NP. To our knowledge, this is the first study to report the formation of SpyCatcher pentamers on the dodecahedral faces of mi3 NPs. To investigate the immunogenicity, rabbits were primed with two doses of DNA vaccines expressing the CAP255 gp150 and a mosaic subtype C Gag and boosted with three doses of the NP-developed autologous Tier 2 CAP255 neutralising antibodies (Nabs) and low levels of heterologous CAP256SU NAbs. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_50615.map.gz emd_50615.map.gz | 6.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-50615-v30.xml emd-50615-v30.xml emd-50615.xml emd-50615.xml | 17.3 KB 17.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_50615_fsc.xml emd_50615_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_50615.png emd_50615.png | 75.7 KB | ||
| マスクデータ |  emd_50615_msk_1.map emd_50615_msk_1.map | 91.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-50615.cif.gz emd-50615.cif.gz | 6.4 KB | ||
| その他 |  emd_50615_half_map_1.map.gz emd_50615_half_map_1.map.gz emd_50615_half_map_2.map.gz emd_50615_half_map_2.map.gz | 56.9 MB 57.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-50615 http://ftp.pdbj.org/pub/emdb/structures/EMD-50615 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50615 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50615 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_50615_validation.pdf.gz emd_50615_validation.pdf.gz | 811 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_50615_full_validation.pdf.gz emd_50615_full_validation.pdf.gz | 810.6 KB | 表示 | |
| XML形式データ |  emd_50615_validation.xml.gz emd_50615_validation.xml.gz | 17.4 KB | 表示 | |
| CIF形式データ |  emd_50615_validation.cif.gz emd_50615_validation.cif.gz | 22.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50615 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50615 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50615 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50615 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9fo3MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_50615.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_50615.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CAP255-gp140 SpyTag-SpyCatcher mi3 masked | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.662 Å | ||||||||||||||||||||||||||||||||||||
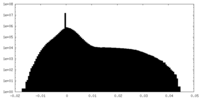
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_50615_msk_1.map emd_50615_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
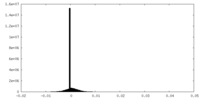
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_50615_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
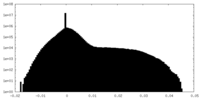
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_50615_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
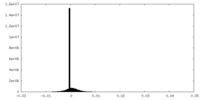
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CAP255 gp140 SpyTag-SpyCatcher mi3 nanoparticle
| 全体 | 名称: CAP255 gp140 SpyTag-SpyCatcher mi3 nanoparticle |
|---|---|
| 要素 |
|
-超分子 #1: CAP255 gp140 SpyTag-SpyCatcher mi3 nanoparticle
| 超分子 | 名称: CAP255 gp140 SpyTag-SpyCatcher mi3 nanoparticle / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: CAP255 gp140 SpyTag was expressed in HEK293T cells and purified. SpyCatcher mi3 was expressed in |
|---|---|
| 由来(天然) | 生物種:   Thermotoga maritima (バクテリア) Thermotoga maritima (バクテリア) |
| 分子量 | 理論値: 4.75 MDa |
-分子 #1: CAP255 gp140 SpyTag-SpyCatcher mi3,2-dehydro-3-deoxyphosphoglucon...
| 分子 | 名称: CAP255 gp140 SpyTag-SpyCatcher mi3,2-dehydro-3-deoxyphosphogluconate aldolase/4-hydroxy-2-oxoglutarate aldolase,2-dehydro-3-deoxyphosphogluconate aldolase/4-hydroxy-2-oxoglutarate aldolase タイプ: protein_or_peptide / ID: 1 詳細: Model just contains mi3 cage component of NP. Note that SpyCatcher and gp140 are not included in the model.,Model just contains mi3 cage component of NP. Note that SpyCatcher and gp140 are ...詳細: Model just contains mi3 cage component of NP. Note that SpyCatcher and gp140 are not included in the model.,Model just contains mi3 cage component of NP. Note that SpyCatcher and gp140 are not included in the model.,Model just contains mi3 cage component of NP. Note that SpyCatcher and gp140 are not included in the model.,Model just contains mi3 cage component of NP. Note that SpyCatcher and gp140 are not included in the model. コピー数: 60 / 光学異性体: LEVO / EC番号: 2-dehydro-3-deoxy-phosphogluconate aldolase |
|---|---|
| 由来(天然) | 生物種:   Thermotoga maritima (バクテリア) Thermotoga maritima (バクテリア) |
| 分子量 | 理論値: 33.894777 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: HHHHHHGSGD SATHIKFSKR DEDGKELAGA TMELRDSSGK TISTWISDGQ VKDFYLYPGK YTFVETAAPD GYEVATAITF TVNEQGQVT VNGKATKGDA HIGGSGGSGG SGGSMKMEEL FKKHKIVAVL RANSVEEAKK KALAVFLGGV HLIEITFTVP D ADTVIKEL ...文字列: HHHHHHGSGD SATHIKFSKR DEDGKELAGA TMELRDSSGK TISTWISDGQ VKDFYLYPGK YTFVETAAPD GYEVATAITF TVNEQGQVT VNGKATKGDA HIGGSGGSGG SGGSMKMEEL FKKHKIVAVL RANSVEEAKK KALAVFLGGV HLIEITFTVP D ADTVIKEL SFLKEMGAII GAGTVTSVEQ ARKAVESGAE FIVSPHLDEE ISQFAKEKGV FYMPGVMTPT ELVKAMKLGH TI LKLFPGE VVGPQFVKAM KGPFPNVKFV PTGGVNLDNV CEWFKAGVLA VGVGSALVKG TPVEVAEKAK AFVEKIRGCT E UniProtKB: 2-dehydro-3-deoxyphosphogluconate aldolase/4-hydroxy-2-oxoglutarate aldolase |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 構成要素:
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 平均電子線量: 45.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL 当てはまり具合の基準: Cross-correlation coefficient |
| 得られたモデル |  PDB-9fo3: |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)