| 登録情報 | データベース: PDB / ID: 4bds
|
|---|
| タイトル | Human butyrylcholinesterase in complex with tacrine |
|---|
 要素 要素 | CHOLINESTERASE |
|---|
 キーワード キーワード | HYDROLASE / NERVE TRANSMISSION / INHIBITIOR / ALPHA-BETA HYDROLASE |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
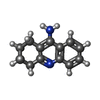
cholinesterase / : / neuroblast differentiation / response to folic acid / choline binding / Neurotransmitter clearance / cholinesterase activity / choline metabolic process / response to alkaloid / acetylcholine catabolic process ...cholinesterase / : / neuroblast differentiation / response to folic acid / choline binding / Neurotransmitter clearance / cholinesterase activity / choline metabolic process / response to alkaloid / acetylcholine catabolic process / negative regulation of synaptic transmission / peptide hormone processing / hydrolase activity, acting on ester bonds / acetylcholinesterase activity / Aspirin ADME / nuclear envelope lumen / Synthesis of PC / catalytic activity / Synthesis, secretion, and deacylation of Ghrelin / xenobiotic metabolic process / response to glucocorticoid / learning / amyloid-beta binding / blood microparticle / endoplasmic reticulum lumen / negative regulation of cell population proliferation / enzyme binding / extracellular space / extracellular region / identical protein binding / plasma membrane類似検索 - 分子機能 Acetylcholinesterase, tetramerisation domain / Acetylcholinesterase tetramerisation domain / : / Cholinesterase / Carboxylesterase type B, active site / Carboxylesterases type-B serine active site. / Carboxylesterase type B, conserved site / Carboxylesterases type-B signature 2. / Carboxylesterase, type B / Carboxylesterase family ...Acetylcholinesterase, tetramerisation domain / Acetylcholinesterase tetramerisation domain / : / Cholinesterase / Carboxylesterase type B, active site / Carboxylesterases type-B serine active site. / Carboxylesterase type B, conserved site / Carboxylesterases type-B signature 2. / Carboxylesterase, type B / Carboxylesterase family / Alpha/Beta hydrolase fold, catalytic domain / Alpha/Beta hydrolase fold / Rossmann fold / 3-Layer(aba) Sandwich / Alpha Beta類似検索 - ドメイン・相同性 1-formyl-L-proline / alpha-L-fucopyranose / TACRINE / Cholinesterase類似検索 - 構成要素 |
|---|
| 生物種 |  HOMO SAPIENS (ヒト) HOMO SAPIENS (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.1 Å 分子置換 / 解像度: 2.1 Å |
|---|
 データ登録者 データ登録者 | Nachon, F. / Carletti, E. / Ronco, C. / Trovaslet, M. / Nicolet, Y. / Jean, L. / Renard, P.-Y. |
|---|
 引用 引用 |  ジャーナル: Biochem.J. / 年: 2013 ジャーナル: Biochem.J. / 年: 2013
タイトル: Crystal Structures of Human Cholinesterases in Complex with Huprine W and Tacrine: Elements of Specificity for Anti-Alzheimer'S Drugs Targeting Acetyl- and Butyrylcholinesterase.
著者: Nachon, F. / Carletti, E. / Ronco, C. / Trovaslet, M. / Nicolet, Y. / Jean, L. / Renard, P. |
|---|
| 履歴 | | 登録 | 2012年10月6日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2013年5月29日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2013年7月31日 | Group: Database references |
|---|
| 改定 1.2 | 2014年4月9日 | Group: Atomic model / Derived calculations / Non-polymer description |
|---|
| 改定 2.0 | 2017年11月15日 | Group: Advisory / Atomic model ...Advisory / Atomic model / Derived calculations / Non-polymer description / Structure summary
カテゴリ: atom_site / entity ...atom_site / entity / pdbx_unobs_or_zero_occ_atoms / pdbx_validate_close_contact / struct_site_gen
Item: _atom_site.auth_comp_id / _atom_site.label_comp_id ..._atom_site.auth_comp_id / _atom_site.label_comp_id / _chem_comp.formula / _chem_comp.id / _chem_comp.name / _entity.formula_weight / _entity.pdbx_description / _pdbx_validate_close_contact.auth_comp_id_1 / _struct_site_gen.auth_comp_id / _struct_site_gen.label_comp_id |
|---|
| 改定 2.1 | 2019年3月6日 | Group: Data collection / Derived calculations / Experimental preparation
カテゴリ: exptl_crystal_grow / struct_conn
Item: _exptl_crystal_grow.temp / _struct_conn.pdbx_leaving_atom_flag |
|---|
| 改定 3.0 | 2020年7月29日 | Group: Advisory / Atomic model ...Advisory / Atomic model / Data collection / Derived calculations / Other / Structure summary
カテゴリ: atom_site / chem_comp ...atom_site / chem_comp / entity / pdbx_branch_scheme / pdbx_chem_comp_identifier / pdbx_database_status / pdbx_entity_branch / pdbx_entity_branch_descriptor / pdbx_entity_branch_link / pdbx_entity_branch_list / pdbx_entity_nonpoly / pdbx_nonpoly_scheme / pdbx_struct_assembly_gen / pdbx_struct_special_symmetry / pdbx_unobs_or_zero_occ_atoms / struct_asym / struct_conn / struct_site / struct_site_gen
Item: _atom_site.B_iso_or_equiv / _atom_site.Cartn_x ..._atom_site.B_iso_or_equiv / _atom_site.Cartn_x / _atom_site.Cartn_y / _atom_site.Cartn_z / _atom_site.auth_asym_id / _atom_site.auth_atom_id / _atom_site.auth_comp_id / _atom_site.auth_seq_id / _atom_site.label_asym_id / _atom_site.label_atom_id / _atom_site.label_comp_id / _atom_site.label_entity_id / _atom_site.type_symbol / _chem_comp.name / _chem_comp.type / _entity.formula_weight / _entity.pdbx_description / _entity.pdbx_number_of_molecules / _entity.type / _pdbx_database_status.status_code_sf / _pdbx_struct_assembly_gen.asym_id_list / _pdbx_struct_special_symmetry.label_asym_id / _pdbx_unobs_or_zero_occ_atoms.label_asym_id / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.pdbx_role / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_comp_id
解説: Carbohydrate remediation / Provider: repository / タイプ: Remediation |
|---|
| 改定 4.0 | 2023年11月15日 | Group: Atomic model / Data collection ...Atomic model / Data collection / Database references / Derived calculations / Structure summary
カテゴリ: atom_site / chem_comp ...atom_site / chem_comp / chem_comp_atom / chem_comp_bond / database_2 / struct_conn
Item: _atom_site.auth_atom_id / _atom_site.label_atom_id ..._atom_site.auth_atom_id / _atom_site.label_atom_id / _chem_comp.pdbx_synonyms / _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.pdbx_leaving_atom_flag |
|---|
| 改定 4.1 | 2023年12月20日 | Group: Refinement description / カテゴリ: pdbx_initial_refinement_model |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 HOMO SAPIENS (ヒト)
HOMO SAPIENS (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.1 Å
分子置換 / 解像度: 2.1 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Biochem.J. / 年: 2013
ジャーナル: Biochem.J. / 年: 2013 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4bds.cif.gz
4bds.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4bds.ent.gz
pdb4bds.ent.gz PDB形式
PDB形式 4bds.json.gz
4bds.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/bd/4bds
https://data.pdbj.org/pub/pdb/validation_reports/bd/4bds ftp://data.pdbj.org/pub/pdb/validation_reports/bd/4bds
ftp://data.pdbj.org/pub/pdb/validation_reports/bd/4bds リンク
リンク 集合体
集合体
 要素
要素 HOMO SAPIENS (ヒト) / プラスミド: PGS / 細胞株 (発現宿主): CHO-K1
HOMO SAPIENS (ヒト) / プラスミド: PGS / 細胞株 (発現宿主): CHO-K1














 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ESRF
ESRF  / ビームライン: ID14-2 / 波長: 0.933
/ ビームライン: ID14-2 / 波長: 0.933  解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー















































 PDBj
PDBj

















