+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Septin Hexameric Complex SEPT2/SEPT6/SEPT7 of Ciona intestinalis by Cryo-EM | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Complex / SPA / Cytoskeleton / Septin / CELL CYCLE | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報septin complex / septin ring / cytoskeleton-dependent cytokinesis / cell division site / cleavage furrow / cilium assembly / cilium / kinetochore / microtubule cytoskeleton / protein localization ...septin complex / septin ring / cytoskeleton-dependent cytokinesis / cell division site / cleavage furrow / cilium assembly / cilium / kinetochore / microtubule cytoskeleton / protein localization / molecular adaptor activity / GTPase activity / GTP binding 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  | ||||||||||||
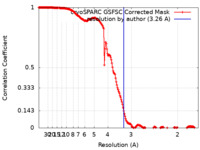
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.26 Å | ||||||||||||
 データ登録者 データ登録者 | Mendonca DC / Pereira HM / Garratt RC | ||||||||||||
| 資金援助 |  ブラジル, 3件 ブラジル, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2024 ジャーナル: J Mol Biol / 年: 2024タイトル: Structural Insights into Ciona intestinalis Septins: Complexes Suggest a Mechanism for Nucleotide-dependent Interfacial Cross-talk. 著者: Deborah C Mendonça / Sinara T B Morais / Heloísa Ciol / Andressa P A Pinto / Diego A Leonardo / Humberto D'Muniz Pereira / Napoleão F Valadares / Rodrigo V Portugal / Bruno P Klaholz / ...著者: Deborah C Mendonça / Sinara T B Morais / Heloísa Ciol / Andressa P A Pinto / Diego A Leonardo / Humberto D'Muniz Pereira / Napoleão F Valadares / Rodrigo V Portugal / Bruno P Klaholz / Richard C Garratt / Ana P U Araujo /   要旨: Septins are filamentous nucleotide-binding proteins which can associate with membranes in a curvature-dependent manner leading to structural remodelling and barrier formation. Ciona intestinalis, a ...Septins are filamentous nucleotide-binding proteins which can associate with membranes in a curvature-dependent manner leading to structural remodelling and barrier formation. Ciona intestinalis, a model for exploring the development and evolution of the chordate lineage, has only four septin-coding genes within its genome. These represent orthologues of the four classical mammalian subgroups, making it a minimalist non-redundant model for studying the modular assembly of septins into linear oligomers and thereby filamentous polymers. Here, we show that C. intestinalis septins present a similar biochemistry to their human orthologues and also provide the cryo-EM structures of an octamer, a hexamer and a tetrameric sub-complex. The octamer, which has the canonical arrangement (2-6-7-9-9-7-6-2) clearly shows an exposed NC-interface at its termini enabling copolymerization with hexamers into mixed filaments. Indeed, only combinations of septins which had CiSEPT2 occupying the terminal position were able to assemble into filaments via NC-interface association. The CiSEPT7-CiSEPT9 tetramer is the smallest septin particle to be solved by Cryo-EM to date and its good resolution (2.7 Å) provides a well-defined view of the central NC-interface. On the other hand, the CiSEPT7-CiSEPT9 G-interface shows signs of fragility permitting toggling between hexamers and octamers, similar to that seen in human septins but not in yeast. The new structures provide insights concerning the molecular mechanism for cross-talk between adjacent interfaces. This indicates that C. intestinalis may represent a valuable tool for future studies, fulfilling the requirements of a complete but simpler system to understand the mechanisms behind the assembly and dynamics of septin filaments. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_44552.map.gz emd_44552.map.gz | 254.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-44552-v30.xml emd-44552-v30.xml emd-44552.xml emd-44552.xml | 21.9 KB 21.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_44552_fsc.xml emd_44552_fsc.xml | 17 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_44552.png emd_44552.png | 58.6 KB | ||
| マスクデータ |  emd_44552_msk_1.map emd_44552_msk_1.map | 512 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-44552.cif.gz emd-44552.cif.gz | 6.3 KB | ||
| その他 |  emd_44552_additional_1.map.gz emd_44552_additional_1.map.gz emd_44552_additional_2.map.gz emd_44552_additional_2.map.gz emd_44552_half_map_1.map.gz emd_44552_half_map_1.map.gz emd_44552_half_map_2.map.gz emd_44552_half_map_2.map.gz | 483.4 MB 449 MB 475.2 MB 475.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-44552 http://ftp.pdbj.org/pub/emdb/structures/EMD-44552 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44552 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44552 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_44552_validation.pdf.gz emd_44552_validation.pdf.gz | 721 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_44552_full_validation.pdf.gz emd_44552_full_validation.pdf.gz | 720.6 KB | 表示 | |
| XML形式データ |  emd_44552_validation.xml.gz emd_44552_validation.xml.gz | 26.8 KB | 表示 | |
| CIF形式データ |  emd_44552_validation.cif.gz emd_44552_validation.cif.gz | 35 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44552 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44552 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44552 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44552 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9bhtMC  9bhwC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_44552.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_44552.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.862 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_44552_msk_1.map emd_44552_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
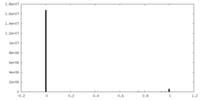
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Sharppened map
| ファイル | emd_44552_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharppened map | ||||||||||||
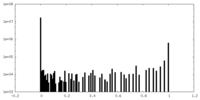
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: DeepEMhancer map
| ファイル | emd_44552_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | DeepEMhancer map | ||||||||||||
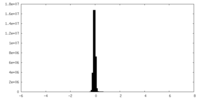
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_44552_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
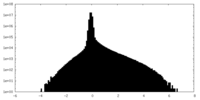
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_44552_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ciona intestinalis Septin Hexameric Complex SEPT2/SEPT6/SEPT7
| 全体 | 名称: Ciona intestinalis Septin Hexameric Complex SEPT2/SEPT6/SEPT7 |
|---|---|
| 要素 |
|
-超分子 #1: Ciona intestinalis Septin Hexameric Complex SEPT2/SEPT6/SEPT7
| 超分子 | 名称: Ciona intestinalis Septin Hexameric Complex SEPT2/SEPT6/SEPT7 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: CiSeptin-2
| 分子 | 名称: CiSeptin-2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 50.766543 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH SSGLVPRGSH MASMTGGQQM GRGSMEKHQL ERHRSIRKNA RIVSHFSPDP KYNPSEIRKK YEGYTSPASK EREEQVKFK FTNPETPGYV GFANLPNQVH RKSVKKGFEF TLMVAGESGL GKSTLVNSLF LTDLYPERCI PPAADKITQT V KIDASTVE ...文字列: MGSSHHHHHH SSGLVPRGSH MASMTGGQQM GRGSMEKHQL ERHRSIRKNA RIVSHFSPDP KYNPSEIRKK YEGYTSPASK EREEQVKFK FTNPETPGYV GFANLPNQVH RKSVKKGFEF TLMVAGESGL GKSTLVNSLF LTDLYPERCI PPAADKITQT V KIDASTVE IEERGVKLRL TVVDTPGYGD AINCSDCYKP IIQYIDDQFE RYLHDESGLN RRHIVDNRVH SCFYFISSQG HG LKPLDIE FMKALHNKVN IVPVLSKADS LTMPEVKRLK RRILEEIAAH EIQIYQLPDA DEDEDEEFKE QTRCLKESIP FAV VGSTQM IEVKGKKVRG RLYPWGVVEV ENPDHCDFLK LRTMLITHMQ DLQEVTHDLH YENFRARRLQ SKDGEPANVS VDSG SNNRA LQEKEAELQR MQEMVQQMQA QIKAQSKSSP TGSKVVSHQI |
-分子 #2: CiSeptin-6
| 分子 | 名称: CiSeptin-6 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 49.661637 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MVKMEKDEAA GDYNGSVDEG MRTLNLSGHV GFDSLPDQLV NKATNQGFCF NILCIGETGL GKSTLMNTLF NTNFENEPQH HNMPGVKLK ANTYELQESN VRLKLTIVDS VGFGDQINKE ESFKPVVEYI NQQFENYLQE ELKICRSLFS YHDTRIHACL Y FISPTGHG ...文字列: MVKMEKDEAA GDYNGSVDEG MRTLNLSGHV GFDSLPDQLV NKATNQGFCF NILCIGETGL GKSTLMNTLF NTNFENEPQH HNMPGVKLK ANTYELQESN VRLKLTIVDS VGFGDQINKE ESFKPVVEYI NQQFENYLQE ELKICRSLFS YHDTRIHACL Y FISPTGHG LKSLDLKTMK NLDNKVNIIP VIAKADIVSK GELHKFKIKI MNELVTNGVQ IYQFPTDDET VAEVNASMNT HL PFAVVGS TEEIKLGNKM VKARQYPWGT VQVENENHCD FVKLREMLVR VNMEDLREKT HTKHYELYRR SKLTEMGFID DND GGKNFS LQEVYESKRN DHLNELQRKE DEMRQMFVLR VKDKEGELKK SEKELHDKFD KLKKMHMEEK KKLEEKKKSL QDEI TGFEK KRQQAETLGK EHLAMSQNKT GKKK UniProtKB: Septin |
-分子 #3: CiSeptin-7
| 分子 | 名称: CiSeptin-7 / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 48.769039 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MVLLNQDMAQ QPKALDSYVG FANLPNQLYR KSVKQGFEFT LMVVGESGLG KSTLINSLFL SDLYSNEYPG PSLRIKKTVK VETTKVLIK ENGVTLRLTI DDTPGFGDAV DNSNCWQAVI NHIEKKFEDY LNAESMVTRS KLADNRVHCC LYFIAPTGHG L KPLDVEFM ...文字列: MVLLNQDMAQ QPKALDSYVG FANLPNQLYR KSVKQGFEFT LMVVGESGLG KSTLINSLFL SDLYSNEYPG PSLRIKKTVK VETTKVLIK ENGVTLRLTI DDTPGFGDAV DNSNCWQAVI NHIEKKFEDY LNAESMVTRS KLADNRVHCC LYFIAPTGHG L KPLDVEFM KNLHDKVNII PLIAKADTMT PEECLRFKKQ IMKEIHEHKI QLYEFPECED EEENRLNRKL KSRVPFAVVG SN TVLEIGG RRVRGRQYPW GVAEVENIDH CDFTVLRNML VRTHMQDLKD VTNNVHYENY RSKKLSSVTT TTMDGRSKAQ TKS PLAQME EEKSEHMQKM KKMEMEEVFR MKVHEKKQKL QESEADLSRR HEAMKKKLEA EYAALDEKQR QYEKDKQDFE ALQL KLMED RKSDNRTLGR KKKW UniProtKB: Septin |
-分子 #4: GUANOSINE-5'-DIPHOSPHATE
| 分子 | 名称: GUANOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 4 / 式: GDP |
|---|---|
| 分子量 | 理論値: 443.201 Da |
| Chemical component information |  ChemComp-GDP: |
-分子 #5: GUANOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: GUANOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 5 / コピー数: 2 / 式: GTP |
|---|---|
| 分子量 | 理論値: 523.18 Da |
| Chemical component information |  ChemComp-GTP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.3 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X