+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The cryo-EM structure of the H2A.Z-H3.3 double-variant nucleosome | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | histone variant / nucleosome / chromatin / complex / STRUCTURAL PROTEIN / STRUCTURAL PROTEIN-DNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of chromosome condensation / Barr body / regulation of centromere complex assembly / muscle cell differentiation / pericentric heterochromatin formation / inner kinetochore / oocyte maturation / nucleus organization / spermatid development / single fertilization ...negative regulation of chromosome condensation / Barr body / regulation of centromere complex assembly / muscle cell differentiation / pericentric heterochromatin formation / inner kinetochore / oocyte maturation / nucleus organization / spermatid development / single fertilization / subtelomeric heterochromatin formation / RNA polymerase II core promoter sequence-specific DNA binding / heterochromatin / Replacement of protamines by nucleosomes in the male pronucleus / heterochromatin organization / nucleosomal DNA binding / Inhibition of DNA recombination at telomere / telomere organization / embryo implantation / RNA Polymerase I Promoter Opening / Assembly of the ORC complex at the origin of replication / DNA methylation / Condensation of Prophase Chromosomes / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / SIRT1 negatively regulates rRNA expression / Chromatin modifications during the maternal to zygotic transition (MZT) / PRC2 methylates histones and DNA / Defective pyroptosis / cellular response to estradiol stimulus / RNA Polymerase I Promoter Escape / Transcriptional regulation by small RNAs / Formation of the beta-catenin:TCF transactivating complex / euchromatin / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / NoRC negatively regulates rRNA expression / Activated PKN1 stimulates transcription of AR (androgen receptor) regulated genes KLK2 and KLK3 / B-WICH complex positively regulates rRNA expression / multicellular organism growth / Meiotic recombination / Pre-NOTCH Transcription and Translation / Activation of anterior HOX genes in hindbrain development during early embryogenesis / osteoblast differentiation / Transcriptional regulation of granulopoiesis / structural constituent of chromatin / cellular response to insulin stimulus / male gonad development / nucleosome / nucleosome assembly / chromatin organization / RUNX1 regulates transcription of genes involved in differentiation of HSCs / Factors involved in megakaryocyte development and platelet production / Senescence-Associated Secretory Phenotype (SASP) / positive regulation of cell growth / Oxidative Stress Induced Senescence / Estrogen-dependent gene expression / cell population proliferation / chromosome, telomeric region / protein heterodimerization activity / Amyloid fiber formation / RNA polymerase II cis-regulatory region sequence-specific DNA binding / positive regulation of transcription by RNA polymerase II / protein-containing complex / DNA binding / extracellular exosome / extracellular region / nucleoplasm / nucleus 類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Tan D / Sokolova V | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Epigenomes / 年: 2024 ジャーナル: Epigenomes / 年: 2024タイトル: Structural and Biochemical Characterization of the Nucleosome Containing Variants H3.3 and H2A.Z 著者: Jung H / Sokolova V / Lee G / Stevens VR / Tan D | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_44148.map.gz emd_44148.map.gz | 4.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-44148-v30.xml emd-44148-v30.xml emd-44148.xml emd-44148.xml | 26.7 KB 26.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_44148.png emd_44148.png | 114.1 KB | ||
| Filedesc metadata |  emd-44148.cif.gz emd-44148.cif.gz | 7.2 KB | ||
| その他 |  emd_44148_half_map_1.map.gz emd_44148_half_map_1.map.gz emd_44148_half_map_2.map.gz emd_44148_half_map_2.map.gz | 46.2 MB 46.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-44148 http://ftp.pdbj.org/pub/emdb/structures/EMD-44148 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44148 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44148 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_44148_validation.pdf.gz emd_44148_validation.pdf.gz | 682.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_44148_full_validation.pdf.gz emd_44148_full_validation.pdf.gz | 682.4 KB | 表示 | |
| XML形式データ |  emd_44148_validation.xml.gz emd_44148_validation.xml.gz | 12.1 KB | 表示 | |
| CIF形式データ |  emd_44148_validation.cif.gz emd_44148_validation.cif.gz | 14.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44148 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44148 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44148 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44148 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9b3pMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_44148.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_44148.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||
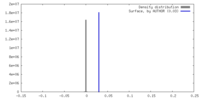
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_44148_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
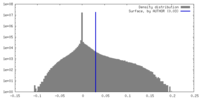
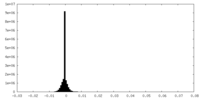
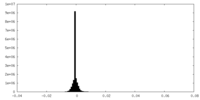
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_44148_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
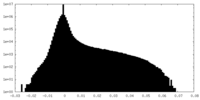
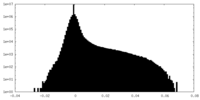
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : nucleosome containing histone variants H3.3 and H2A.Z
| 全体 | 名称: nucleosome containing histone variants H3.3 and H2A.Z |
|---|---|
| 要素 |
|
-超分子 #1: nucleosome containing histone variants H3.3 and H2A.Z
| 超分子 | 名称: nucleosome containing histone variants H3.3 and H2A.Z タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: The complex contains Xenopus histone H2B and H4, human H3.3, and mouse H2A.Z |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 288 KDa |
-分子 #1: Histone H3.3
| 分子 | 名称: Histone H3.3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 15.360983 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MARTKQTARK STGGKAPRKQ LATKAARKSA PSTGGVKKPH RYRPGTVALR EIRRYQKSTE LLIRKLPFQR LVREIAQDFK TDLRFQSAA IGALQEASEA YLVGLFEDTN LCAIHAKRVT IMPKDIQLAR RIRGERA UniProtKB: Histone H3.3 |
-分子 #2: Histone H4
| 分子 | 名称: Histone H4 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 11.394426 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSGRGKGGKG LGKGGAKRHR KVLRDNIQGI TKPAIRRLAR RGGVKRISGL IYEETRGVLK VFLENVIRDA VTYTEHAKRK TVTAMDVVY ALKRQGRTLY GFGG UniProtKB: Histone H4 |
-分子 #3: Histone H2A.Z
| 分子 | 名称: Histone H2A.Z / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 13.581796 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAGGKAGKDS GKAKTKAVSR SQRAGLQFPV GRIHRHLKSR TTSHGRVGAT AAVYSAAILE YLTAEVLELA GNASKDLKVK RITPRHLQL AIRGDEELDS LIKATIAGGG VIPHIHKSLI GKKGQQKTV UniProtKB: Histone H2A.Z |
-分子 #4: Histone H2B 1.1
| 分子 | 名称: Histone H2B 1.1 / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 13.965265 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MPEPAKSAPA PKKGSKKAVT KTQKKDGKKR RKSRKESYAI YVYKVLKQVH PDTGISSKAM SIMNSFVNDV FERIAGEASR LAHYNKRST ITSREIQTAV RLLLPGELAK HAVSEGTKAV TKYTSAK UniProtKB: Histone H2B 1.1 |
-分子 #5: DNA (128-MER)
| 分子 | 名称: DNA (128-MER) / タイプ: dna / ID: 5 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 45.604047 KDa |
| 配列 | 文字列: (DA)(DC)(DA)(DG)(DG)(DA)(DT)(DG)(DT)(DA) (DT)(DA)(DT)(DA)(DT)(DC)(DT)(DG)(DA)(DC) (DA)(DC)(DG)(DT)(DG)(DC)(DC)(DT)(DG) (DG)(DA)(DG)(DA)(DC)(DT)(DA)(DG)(DG)(DG) (DA) (DG)(DT)(DA)(DA)(DT) ...文字列: (DA)(DC)(DA)(DG)(DG)(DA)(DT)(DG)(DT)(DA) (DT)(DA)(DT)(DA)(DT)(DC)(DT)(DG)(DA)(DC) (DA)(DC)(DG)(DT)(DG)(DC)(DC)(DT)(DG) (DG)(DA)(DG)(DA)(DC)(DT)(DA)(DG)(DG)(DG) (DA) (DG)(DT)(DA)(DA)(DT)(DC)(DC)(DC) (DC)(DT)(DT)(DG)(DG)(DC)(DG)(DG)(DT)(DT) (DA)(DA) (DA)(DA)(DC)(DG)(DC)(DG)(DG) (DG)(DG)(DG)(DA)(DC)(DA)(DG)(DC)(DG)(DC) (DG)(DT)(DA) (DC)(DG)(DT)(DG)(DC)(DG) (DT)(DT)(DT)(DA)(DA)(DG)(DC)(DG)(DG)(DT) (DG)(DC)(DT)(DA) (DG)(DA)(DG)(DC)(DT) (DG)(DT)(DC)(DT)(DA)(DC)(DG)(DA)(DC)(DC) (DA)(DA)(DT)(DT)(DG) (DA)(DG)(DC)(DG) (DG)(DC)(DC)(DT)(DC)(DG)(DG)(DC)(DA)(DC) (DC)(DG)(DG)(DG)(DA)(DT) (DT)(DC)(DT) (DC)(DC)(DA)(DG) |
-分子 #6: DNA (128-MER)
| 分子 | 名称: DNA (128-MER) / タイプ: dna / ID: 6 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 51.269656 KDa |
| 配列 | 文字列: (DA)(DT)(DC)(DC)(DC)(DG)(DC)(DC)(DC)(DT) (DG)(DG)(DA)(DG)(DA)(DA)(DT)(DC)(DC)(DC) (DG)(DG)(DT)(DG)(DC)(DC)(DG)(DA)(DG) (DG)(DC)(DC)(DG)(DC)(DT)(DC)(DA)(DA)(DT) (DT) (DG)(DG)(DT)(DC)(DG) ...文字列: (DA)(DT)(DC)(DC)(DC)(DG)(DC)(DC)(DC)(DT) (DG)(DG)(DA)(DG)(DA)(DA)(DT)(DC)(DC)(DC) (DG)(DG)(DT)(DG)(DC)(DC)(DG)(DA)(DG) (DG)(DC)(DC)(DG)(DC)(DT)(DC)(DA)(DA)(DT) (DT) (DG)(DG)(DT)(DC)(DG)(DT)(DA)(DG) (DA)(DC)(DA)(DG)(DC)(DT)(DC)(DT)(DA)(DG) (DC)(DA) (DC)(DC)(DG)(DC)(DT)(DT)(DA) (DA)(DA)(DC)(DG)(DC)(DA)(DC)(DG)(DT)(DA) (DC)(DG)(DC) (DG)(DC)(DT)(DG)(DT)(DC) (DC)(DC)(DC)(DC)(DG)(DC)(DG)(DT)(DT)(DT) (DT)(DA)(DA)(DC) (DC)(DG)(DC)(DC)(DA) (DA)(DG)(DG)(DG)(DG)(DA)(DT)(DT)(DA)(DC) (DT)(DC)(DC)(DC)(DT) (DA)(DG)(DT)(DC) (DT)(DC)(DC)(DA)(DG)(DG)(DC)(DA)(DC)(DG) (DT)(DG)(DT)(DC)(DA)(DG) (DA)(DT)(DA) (DT)(DA)(DT)(DA)(DC)(DA)(DT)(DC)(DC)(DT) (DG)(DT)(DG)(DC)(DA)(DT)(DG) (DA)(DC) (DT)(DA)(DG)(DA)(DT) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.8 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 20mM Tris-HCl, 5mM NaCl |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 気圧: 0.0002 kPa |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 281 K / 装置: FEI VITROBOT MARK IV 詳細: Freezing condition: blot force 0, blot time 4.5 second. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 10 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 2 / 実像数: 5140 / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 倍率(補正後): 81000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.25 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 詳細 | Phenix real-space refinement was used. | ||||||||||||||||||||||
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 温度因子: 87.42 | ||||||||||||||||||||||
| 得られたモデル |  PDB-9b3p: |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)