+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Acanthamoeba polyphaga mimivirus Fanzor2 ternary complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Transposon / Fanzor / cryo-EM / DNA BINDING PROTEIN-DNA-RNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Acanthamoeba polyphaga mimivirus (ウイルス) Acanthamoeba polyphaga mimivirus (ウイルス) | |||||||||
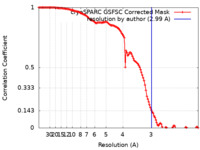
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.99 Å | |||||||||
 データ登録者 データ登録者 | Qayyum MZ / Schargel RD / Tanwar AS / Kellogg EH / Kalathur RC | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: Structure of Fanzor2 reveals insights into the evolution of the TnpB superfamily. 著者: Richard D Schargel / M Zuhaib Qayyum / Ajay Singh Tanwar / Ravi C Kalathur / Elizabeth H Kellogg /  要旨: RNA-guided endonucleases, once thought to be exclusive to prokaryotes, have been recently identified in eukaryotes and are called Fanzors. They are classified into two clades, Fanzor1 and Fanzor2. ...RNA-guided endonucleases, once thought to be exclusive to prokaryotes, have been recently identified in eukaryotes and are called Fanzors. They are classified into two clades, Fanzor1 and Fanzor2. Here we present the cryo-electron microscopy structure of Acanthamoeba polyphaga mimivirus Fanzor2, revealing its ωRNA architecture, active site and features involved in transposon-adjacent motif recognition. A comparison to Fanzor1 and TnpB structures highlights divergent evolutionary paths, advancing our understanding of RNA-guided endonucleases. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
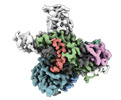
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_44046.map.gz emd_44046.map.gz | 769.7 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-44046-v30.xml emd-44046-v30.xml emd-44046.xml emd-44046.xml | 17.4 KB 17.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_44046_fsc.xml emd_44046_fsc.xml | 11.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_44046.png emd_44046.png | 110.4 KB | ||
| Filedesc metadata |  emd-44046.cif.gz emd-44046.cif.gz | 6 KB | ||
| その他 |  emd_44046_half_map_1.map.gz emd_44046_half_map_1.map.gz emd_44046_half_map_2.map.gz emd_44046_half_map_2.map.gz | 165.3 MB 165.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-44046 http://ftp.pdbj.org/pub/emdb/structures/EMD-44046 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44046 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44046 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_44046_validation.pdf.gz emd_44046_validation.pdf.gz | 641.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_44046_full_validation.pdf.gz emd_44046_full_validation.pdf.gz | 641 KB | 表示 | |
| XML形式データ |  emd_44046_validation.xml.gz emd_44046_validation.xml.gz | 20.5 KB | 表示 | |
| CIF形式データ |  emd_44046_validation.cif.gz emd_44046_validation.cif.gz | 26.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44046 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44046 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44046 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44046 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9b0lMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_44046.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_44046.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.044 Å | ||||||||||||||||||||||||||||||||||||
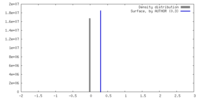
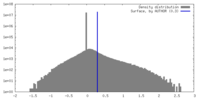
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_44046_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_44046_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Acanthamoeba polyphaga mimivirus Fanzor2 ternary complex
| 全体 | 名称: Acanthamoeba polyphaga mimivirus Fanzor2 ternary complex |
|---|---|
| 要素 |
|
-超分子 #1: Acanthamoeba polyphaga mimivirus Fanzor2 ternary complex
| 超分子 | 名称: Acanthamoeba polyphaga mimivirus Fanzor2 ternary complex タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|---|
| 由来(天然) | 生物種:   Acanthamoeba polyphaga mimivirus (ウイルス) Acanthamoeba polyphaga mimivirus (ウイルス) |
-分子 #1: DNA (5'-D(P*GP*AP*CP*GP*GP*TP*CP*GP*GP*GP*C)-3')
| 分子 | 名称: DNA (5'-D(P*GP*AP*CP*GP*GP*TP*CP*GP*GP*GP*C)-3') / タイプ: dna / ID: 1 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:   Acanthamoeba polyphaga mimivirus (ウイルス) Acanthamoeba polyphaga mimivirus (ウイルス) |
| 分子量 | 理論値: 3.415222 KDa |
| 配列 | 文字列: (DG)(DA)(DC)(DG)(DG)(DT)(DC)(DG)(DG)(DG) (DC) |
-分子 #3: DNA (5'-D(P*CP*CP*CP*A)-3')
| 分子 | 名称: DNA (5'-D(P*CP*CP*CP*A)-3') / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:   Acanthamoeba polyphaga mimivirus (ウイルス) Acanthamoeba polyphaga mimivirus (ウイルス) |
| 分子量 | 理論値: 1.135795 KDa |
| 配列 | 文字列: (DC)(DC)(DC)(DA) |
-分子 #4: DNA (5'-D(P*AP*GP*GP*AP*GP*GP*TP*GP*GP*CP*TP*GP*CP*CP*CP*CP*GP*AP...
| 分子 | 名称: DNA (5'-D(P*AP*GP*GP*AP*GP*GP*TP*GP*GP*CP*TP*GP*CP*CP*CP*CP*GP*AP*CP*CP*GP*TP*C)-3') タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:   Acanthamoeba polyphaga mimivirus (ウイルス) Acanthamoeba polyphaga mimivirus (ウイルス) |
| 分子量 | 理論値: 7.083547 KDa |
| 配列 | 文字列: (DA)(DG)(DG)(DA)(DG)(DG)(DT)(DG)(DG)(DC) (DT)(DG)(DC)(DC)(DC)(DC)(DG)(DA)(DC)(DC) (DG)(DT)(DC) |
-分子 #2: TnpB-like protein L79
| 分子 | 名称: TnpB-like protein L79 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Acanthamoeba polyphaga mimivirus (ウイルス) Acanthamoeba polyphaga mimivirus (ウイルス) |
| 分子量 | 理論値: 60.572387 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKEAVKNVKP KVPAKKRIIT GSKTKKKVFV KKKPPDKKPL KKPVKKTVKT DKPKSIYVPN KDLKISKWIP TPKKEFTEIE TNSWYEHRK FENPNKSPVQ TYNKIVPVVP PESIKQQNLA NKRKKTNRPI VFISSEKIRI YPTKDQQKIL QTWFRLFAYM Y NCTIDYIN ...文字列: MKEAVKNVKP KVPAKKRIIT GSKTKKKVFV KKKPPDKKPL KKPVKKTVKT DKPKSIYVPN KDLKISKWIP TPKKEFTEIE TNSWYEHRK FENPNKSPVQ TYNKIVPVVP PESIKQQNLA NKRKKTNRPI VFISSEKIRI YPTKDQQKIL QTWFRLFAYM Y NCTIDYIN SKKVVLESGR INVAATRKVC NKISVRKAQK TIRDNLIQST NPSIMTHIID EAIGLACSNY KTCLTNYIER HI KKFDIKP WNMSKRKKII IIEANFFKKG TFCPTVFPKM ESSKPLTMID KTVTLQYDSD TRKYILFVPR VTPKYSVNKE KNS CGIDPG LRDFLTVYSE NETQSICPIE IVVNTTKNEY KKIDKINEII KTKPNLNSKR KKKLNRGLRK YHRRVTNKMK DMHY KVSHE LVNTFDKICI GKLNVKSILS KANTVLKSAL KRKLATLSFY RFTQRLTHMG YKYGTEVVNV NEYLTTKTCS NCGKI KDLG ASKIYECESC GMYADRDENA AKNILKVGLK PWYKQK UniProtKB: TnpB-like protein L79 |
-分子 #5: RNA (117-MER)
| 分子 | 名称: RNA (117-MER) / タイプ: rna / ID: 5 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:   Acanthamoeba polyphaga mimivirus (ウイルス) Acanthamoeba polyphaga mimivirus (ウイルス) |
| 分子量 | 理論値: 79.330805 KDa |
| 配列 | 文字列: AAAAAUAGUC UAAUAAAAUC AGGGGUACAU UCCGCUAGUA CUCCACCCUA CGGGUUAAGC AAAUGAGAAU AUCGAAACGG UAUGCACAG GAUUCUUCGA GUGAUAAUCU UAGGAUGACU CACUAAGGAG AUGACUAAAG UGUAUCAUUC AAUAUUGUAU U GAACGGUA ...文字列: AAAAAUAGUC UAAUAAAAUC AGGGGUACAU UCCGCUAGUA CUCCACCCUA CGGGUUAAGC AAAUGAGAAU AUCGAAACGG UAUGCACAG GAUUCUUCGA GUGAUAAUCU UAGGAUGACU CACUAAGGAG AUGACUAAAG UGUAUCAUUC AAUAUUGUAU U GAACGGUA UUCUUCCAUA GAGAGUUGAU UUUUGGAGUA UCCAAAAAUA UCAACUUUUU AUGAGCGGGC AGCCACCUCC UU GUUAUUG GENBANK: GENBANK: AY653733.1 |
-分子 #6: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 6 / コピー数: 1 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-分子 #7: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 7 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 51.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: OTHER / 最大 デフォーカス(公称値): 2.25 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)