+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of biofilm-forming functional amyloid PSMa1 from Staphylococcus aureus | |||||||||
 マップデータ マップデータ | DeepEM processed map. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | functional amyloid fibril / biofilm / bacterial biofilm / phenol soluble modulin alpha1 / PSMa1 / STRUCTURAL PROTEIN | |||||||||
| 機能・相同性 | Phenol-soluble modulin alpha peptide / Phenol-soluble modulin alpha peptide family / killing of cells of another organism / Phenol-soluble modulin alpha 1 peptide 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
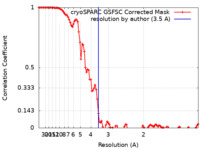
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Hansen KH / Byeon CH / Liu Q / Drace T / Boesen T / Conway JF / Andreasen M / Akbey U | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Proceedings of the National Academy of Sciences of the United States of America ジャーナル: Proceedings of the National Academy of Sciences of the United States of America年: 2024 タイトル: Structure of biofilm-forming functional amyloid PSMa1 from Staphylococcus aureus 著者: Hansen KH / Byeon CH / Liu Q / Drace T / Boesen T / Conway JF / Andreasen M / Akbey U | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_43835.map.gz emd_43835.map.gz | 93.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-43835-v30.xml emd-43835-v30.xml emd-43835.xml emd-43835.xml | 22.5 KB 22.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_43835_fsc.xml emd_43835_fsc.xml | 9.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_43835.png emd_43835.png | 61 KB | ||
| マスクデータ |  emd_43835_msk_1.map emd_43835_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-43835.cif.gz emd-43835.cif.gz | 5.6 KB | ||
| その他 |  emd_43835_additional_1.map.gz emd_43835_additional_1.map.gz emd_43835_additional_2.map.gz emd_43835_additional_2.map.gz emd_43835_additional_3.map.gz emd_43835_additional_3.map.gz emd_43835_half_map_1.map.gz emd_43835_half_map_1.map.gz emd_43835_half_map_2.map.gz emd_43835_half_map_2.map.gz | 50.6 MB 52.4 MB 97 MB 95.5 MB 95.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-43835 http://ftp.pdbj.org/pub/emdb/structures/EMD-43835 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43835 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43835 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_43835_validation.pdf.gz emd_43835_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_43835_full_validation.pdf.gz emd_43835_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_43835_validation.xml.gz emd_43835_validation.xml.gz | 18.7 KB | 表示 | |
| CIF形式データ |  emd_43835_validation.cif.gz emd_43835_validation.cif.gz | 23.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43835 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43835 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43835 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43835 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9atwMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_43835.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_43835.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | DeepEM processed map. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.647 Å | ||||||||||||||||||||||||||||||||||||
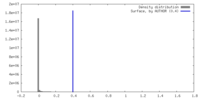
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_43835_msk_1.map emd_43835_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
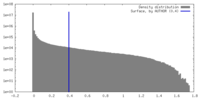
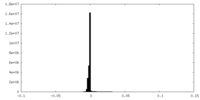
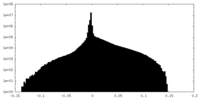
| 密度ヒストグラム |
-追加マップ: CryoSparc output unsharpened.
| ファイル | emd_43835_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoSparc output unsharpened. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
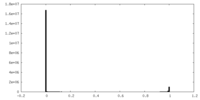
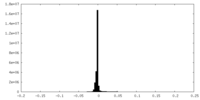
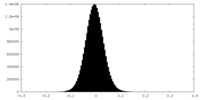
| 密度ヒストグラム |
-追加マップ: CryoSparc sharpened to B:-100
| ファイル | emd_43835_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoSparc sharpened to B:-100 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
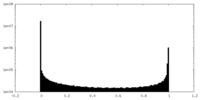
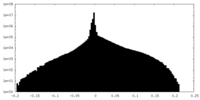
| 密度ヒストグラム |
-追加マップ: CryoSparc auto-sharpened
| ファイル | emd_43835_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoSparc auto-sharpened | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
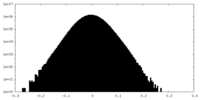
| 密度ヒストグラム |
-ハーフマップ: Half Map B from CryoSparc
| ファイル | emd_43835_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map B from CryoSparc | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
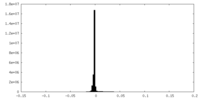
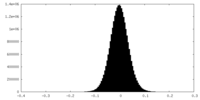
| 密度ヒストグラム |
-ハーフマップ: Half Map A from CryoSparc
| ファイル | emd_43835_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map A from CryoSparc | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Phenol Soluble Modulin alpha1 (PSMAlpha1) (PSMa1)
| 全体 | 名称: Phenol Soluble Modulin alpha1 (PSMAlpha1) (PSMa1) |
|---|---|
| 要素 |
|
-超分子 #1: Phenol Soluble Modulin alpha1 (PSMAlpha1) (PSMa1)
| 超分子 | 名称: Phenol Soluble Modulin alpha1 (PSMAlpha1) (PSMa1) / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Biofilm forming functional amyloid from Staphylococcus aureus PSMa1 is produced by peptide-synthesis |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Phenol-soluble modulin alpha 1 peptide
| 分子 | 名称: Phenol-soluble modulin alpha 1 peptide / タイプ: protein_or_peptide / ID: 1 / コピー数: 64 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 2.262817 KDa |
| 配列 | 文字列: MGIIAGIIKV IKSLIEQFTG K UniProtKB: Phenol-soluble modulin alpha 1 peptide |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.8 / 詳細: water |
| グリッド | モデル: Quantifoil / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. 詳細: C-Flat R2/2 Cu 300 mesh holey carbon grids (Protochips) were glow discharged for 45 s at 15 mA using a Quorum GloQube Plus |
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 99 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: plunge-frozen in liquid ethane using a Vitrobot Mark IV plunge freezer with a blot force of zero at 4C and 99% humidity.. |
| 詳細 | 3 microL of PSMa1 (0.5 mg/mL) was applied to grids Blotted for 6s |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 3002 / 平均露光時間: 1.5 sec. / 平均電子線量: 63.0 e/Å2 詳細: A total of 3,002 movies of the PSMa1 sample were collected |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: FLEXIBLE FIT / 温度因子: 50 |
|---|---|
| 得られたモデル |  PDB-9atw: |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)