+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Aca2 from Pectobacterium phage ZF40 bound to RNA | ||||||||||||
 マップデータ マップデータ | Sharpened map of Aca2 bound to RNA | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | HTH protein / CRISPR / RNA-binding protein / RNA BINDING PROTEIN | ||||||||||||
| 機能・相同性 | Protein of unknown function DUF1870 / Domain of unknown function (DUF1870) / YdiL domain superfamily / Lambda repressor-like, DNA-binding domain superfamily / DNA binding / metal ion binding / DUF1870 family protein 機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |  Pectobacterium phage ZF40 (ファージ) Pectobacterium phage ZF40 (ファージ) | ||||||||||||
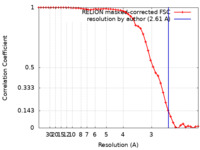
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.61 Å | ||||||||||||
 データ登録者 データ登録者 | Wilkinson ME / Birkholz N / Kimanius D / Fineran PC | ||||||||||||
| 資金援助 |  米国, 米国,  ニュージーランド, ニュージーランド,  ドイツ, 3件 ドイツ, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2024 ジャーナル: Nature / 年: 2024タイトル: Phage anti-CRISPR control by an RNA- and DNA-binding helix-turn-helix protein. 著者: Nils Birkholz / Kotaro Kamata / Maximilian Feussner / Max E Wilkinson / Christian Cuba Samaniego / Angela Migur / Dari Kimanius / Marijn Ceelen / Sam C Went / Ben Usher / Tim R Blower / Chris ...著者: Nils Birkholz / Kotaro Kamata / Maximilian Feussner / Max E Wilkinson / Christian Cuba Samaniego / Angela Migur / Dari Kimanius / Marijn Ceelen / Sam C Went / Ben Usher / Tim R Blower / Chris M Brown / Chase L Beisel / Zasha Weinberg / Robert D Fagerlund / Simon A Jackson / Peter C Fineran /      要旨: In all organisms, regulation of gene expression must be adjusted to meet cellular requirements and frequently involves helix-turn-helix (HTH) domain proteins. For instance, in the arms race between ...In all organisms, regulation of gene expression must be adjusted to meet cellular requirements and frequently involves helix-turn-helix (HTH) domain proteins. For instance, in the arms race between bacteria and bacteriophages, rapid expression of phage anti-CRISPR (acr) genes upon infection enables evasion from CRISPR-Cas defence; transcription is then repressed by an HTH-domain-containing anti-CRISPR-associated (Aca) protein, probably to reduce fitness costs from excessive expression. However, how a single HTH regulator adjusts anti-CRISPR production to cope with increasing phage genome copies and accumulating acr mRNA is unknown. Here we show that the HTH domain of the regulator Aca2, in addition to repressing Acr synthesis transcriptionally through DNA binding, inhibits translation of mRNAs by binding conserved RNA stem-loops and blocking ribosome access. The cryo-electron microscopy structure of the approximately 40 kDa Aca2-RNA complex demonstrates how the versatile HTH domain specifically discriminates RNA from DNA binding sites. These combined regulatory modes are widespread in the Aca2 family and facilitate CRISPR-Cas inhibition in the face of rapid phage DNA replication without toxic acr overexpression. Given the ubiquity of HTH-domain-containing proteins, it is anticipated that many more of them elicit regulatory control by dual DNA and RNA binding. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_43762.map.gz emd_43762.map.gz | 7.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-43762-v30.xml emd-43762-v30.xml emd-43762.xml emd-43762.xml | 19.8 KB 19.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_43762_fsc.xml emd_43762_fsc.xml | 4.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_43762.png emd_43762.png | 122.6 KB | ||
| マスクデータ |  emd_43762_msk_1.map emd_43762_msk_1.map | 8 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-43762.cif.gz emd-43762.cif.gz | 6.3 KB | ||
| その他 |  emd_43762_half_map_1.map.gz emd_43762_half_map_1.map.gz emd_43762_half_map_2.map.gz emd_43762_half_map_2.map.gz | 6 MB 6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-43762 http://ftp.pdbj.org/pub/emdb/structures/EMD-43762 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43762 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43762 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_43762_validation.pdf.gz emd_43762_validation.pdf.gz | 810.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_43762_full_validation.pdf.gz emd_43762_full_validation.pdf.gz | 810.3 KB | 表示 | |
| XML形式データ |  emd_43762_validation.xml.gz emd_43762_validation.xml.gz | 9.9 KB | 表示 | |
| CIF形式データ |  emd_43762_validation.cif.gz emd_43762_validation.cif.gz | 13.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43762 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43762 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43762 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43762 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8w35MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_43762.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_43762.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map of Aca2 bound to RNA | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.0618 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_43762_msk_1.map emd_43762_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
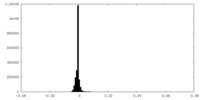
| 密度ヒストグラム |
-ハーフマップ: Aca2-RNA refinement, half-map 1
| ファイル | emd_43762_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Aca2-RNA refinement, half-map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
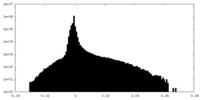
| 密度ヒストグラム |
-ハーフマップ: Aca2-RNA refinement, half-map 2
| ファイル | emd_43762_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Aca2-RNA refinement, half-map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
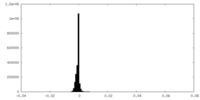
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Anti-CRISPR associated (Aca) protein, Aca2 bound to its 5'UTR RNA...
| 全体 | 名称: Anti-CRISPR associated (Aca) protein, Aca2 bound to its 5'UTR RNA (IR2 and IR-RBS) |
|---|---|
| 要素 |
|
-超分子 #1: Anti-CRISPR associated (Aca) protein, Aca2 bound to its 5'UTR RNA...
| 超分子 | 名称: Anti-CRISPR associated (Aca) protein, Aca2 bound to its 5'UTR RNA (IR2 and IR-RBS) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Pectobacterium phage ZF40 (ファージ) Pectobacterium phage ZF40 (ファージ) |
| 分子量 | 理論値: 39.5 KDa |
-分子 #1: Anti-CRISPR associated (Aca) protein, Aca2
| 分子 | 名称: Anti-CRISPR associated (Aca) protein, Aca2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pectobacterium phage ZF40 (ファージ) Pectobacterium phage ZF40 (ファージ) |
| 分子量 | 理論値: 13.710401 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SHGSMTNKEL QAIRKLLMLD VSEAAEHIGR VSARSWQYWE SGRSAVPDDV EQEMLDLASV RIEMMSAIDK RLADGERPKL RFYNKLDEY LADNPDHNVI GWRLSQSVAA LYYTEGHADL I UniProtKB: DUF1870 family protein |
-分子 #2: IR2 and IR-RBS RNA
| 分子 | 名称: IR2 and IR-RBS RNA / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Pectobacterium phage ZF40 (ファージ) Pectobacterium phage ZF40 (ファージ) |
| 分子量 | 理論値: 13.463021 KDa |
| 配列 | 文字列: AUCGGUUCGA GAUGGCUCGA AUCGCUCCUA ACGAGGAUUC CA |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5.1 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR / 詳細: 25 mA plasma | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 285 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 11232 / 平均露光時間: 1.33 sec. / 平均電子線量: 72.2 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 165000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-8w35: |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)