+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | T33-ml35 Assembly Intermediate - Designed Tetrahedral Protein Cage Using Machine Learning Algorithms | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Nanohedra / protein cage / tetrahedral / de novo protein interface / machine learning / two components / ProteinMPNN / nanoparticle / DE NOVO PROTEIN | |||||||||
| 生物種 | synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.42 Å | |||||||||
 データ登録者 データ登録者 | Castells-Graells R / Meador K / Sawaya MR / Yeates TO | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2024 ジャーナル: Structure / 年: 2024タイトル: A suite of designed protein cages using machine learning and protein fragment-based protocols. 著者: Kyle Meador / Roger Castells-Graells / Roman Aguirre / Michael R Sawaya / Mark A Arbing / Trent Sherman / Chethaka Senarathne / Todd O Yeates /  要旨: Designed protein cages and related materials provide unique opportunities for applications in biotechnology and medicine, but their creation remains challenging. Here, we apply computational ...Designed protein cages and related materials provide unique opportunities for applications in biotechnology and medicine, but their creation remains challenging. Here, we apply computational approaches to design a suite of tetrahedrally symmetric, self-assembling protein cages. For the generation of docked conformations, we emphasize a protein fragment-based approach, while for sequence design of the de novo interface, a comparison of knowledge-based and machine learning protocols highlights the power and increased experimental success achieved using ProteinMPNN. An analysis of design outcomes provides insights for improving interface design protocols, including prioritizing fragment-based motifs, balancing interface hydrophobicity and polarity, and identifying preferred polar contact patterns. In all, we report five structures for seven protein cages, along with two structures of intermediate assemblies, with the highest resolution reaching 2.0 Å using cryo-EM. This set of designed cages adds substantially to the body of available protein nanoparticles, and to methodologies for their creation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42382.map.gz emd_42382.map.gz | 38.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42382-v30.xml emd-42382-v30.xml emd-42382.xml emd-42382.xml | 15.8 KB 15.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_42382.png emd_42382.png | 100.6 KB | ||
| Filedesc metadata |  emd-42382.cif.gz emd-42382.cif.gz | 5.4 KB | ||
| その他 |  emd_42382_half_map_1.map.gz emd_42382_half_map_1.map.gz emd_42382_half_map_2.map.gz emd_42382_half_map_2.map.gz | 37.7 MB 37.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42382 http://ftp.pdbj.org/pub/emdb/structures/EMD-42382 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42382 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42382 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_42382_validation.pdf.gz emd_42382_validation.pdf.gz | 889.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_42382_full_validation.pdf.gz emd_42382_full_validation.pdf.gz | 889.3 KB | 表示 | |
| XML形式データ |  emd_42382_validation.xml.gz emd_42382_validation.xml.gz | 11.1 KB | 表示 | |
| CIF形式データ |  emd_42382_validation.cif.gz emd_42382_validation.cif.gz | 13.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42382 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42382 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42382 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42382 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42382.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42382.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_42382_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
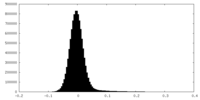
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_42382_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
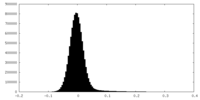
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : T33-ml35 Assembly Intermediate - Designed Tetrahedral Protein Cag...
| 全体 | 名称: T33-ml35 Assembly Intermediate - Designed Tetrahedral Protein Cage Using Machine Learning |
|---|---|
| 要素 |
|
-超分子 #1: T33-ml35 Assembly Intermediate - Designed Tetrahedral Protein Cag...
| 超分子 | 名称: T33-ml35 Assembly Intermediate - Designed Tetrahedral Protein Cage Using Machine Learning タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 400 KDa |
-分子 #1: T33-ml35-redesigned-TPR-domain-fold
| 分子 | 名称: T33-ml35-redesigned-TPR-domain-fold / タイプ: protein_or_peptide / ID: 1 / コピー数: 9 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 13.557165 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MNLAEKMYKA GQIEFAKGNY ETAIIAYTLA LLKDPNNAEA WYNLGEAYLA LGNYEEAIEA YQKALELDPN NAEAWYNLGE AYLALGDYD NAIEAFTKAL ELDPNNKTAK AGLKLAKEKK ALE |
-分子 #2: T33-ml35-redesigned-CutA-fold
| 分子 | 名称: T33-ml35-redesigned-CutA-fold / タイプ: protein_or_peptide / ID: 2 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 14.324456 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTDLSSLIET ADLRLLLTTV PTETEALYLA LAAVEKGLAA EVLITPVTRV RRENGKLVVE DVYRLSFKTT RERLDALVAW LQRRHPLAL PECLVLTPIA SSVAYRDWLR SSLQGGSHHW GGHHHHHH |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 291 K |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 47.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 4.42 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC / 使用した粒子像数: 66561 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
-原子モデル構築 1
| 精密化 | プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-8umr: |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)