+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | S1V2-72 Fab bound to EHA2 from influenza B/Malaysia/2506/2004 | ||||||||||||
 マップデータ マップデータ | CryoEM map of S1V2-72 Fab bound to EHA2 from influenza B/Malaysia/2506/2004 | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | immunoglobulin / complex / hemagglutinin / VIRAL PROTEIN / VIRAL PROTEIN-IMMUNE SYSTEM complex | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報viral budding from plasma membrane / endocytosis involved in viral entry into host cell / host cell surface receptor binding / apical plasma membrane / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane / virion membrane 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Influenza B virus (B/Malaysia/2506/2004) (B型インフルエンザウイルス) / Influenza B virus (B/Malaysia/2506/2004) (B型インフルエンザウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
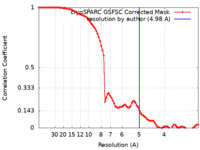
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.98 Å | ||||||||||||
 データ登録者 データ登録者 | Finney J / Kong S / Walsh Jr RM / Harrison SC / Kelsoe G | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2024 ジャーナル: Proc Natl Acad Sci U S A / 年: 2024タイトル: Protective human antibodies against a conserved epitope in pre- and postfusion influenza hemagglutinin. 著者: Joel Finney / Annie Park Moseman / Susan Kong / Akiko Watanabe / Shengli Song / Richard M Walsh / Masayuki Kuraoka / Ryutaro Kotaki / E Ashley Moseman / Kevin R McCarthy / Dongmei Liao / ...著者: Joel Finney / Annie Park Moseman / Susan Kong / Akiko Watanabe / Shengli Song / Richard M Walsh / Masayuki Kuraoka / Ryutaro Kotaki / E Ashley Moseman / Kevin R McCarthy / Dongmei Liao / Xiaoe Liang / Xiaoyan Nie / Olivia Lavidor / Richard Abbott / Stephen C Harrison / Garnett Kelsoe /  要旨: Phylogenetically and antigenically distinct influenza A and B viruses (IAV and IBV) circulate in human populations, causing widespread morbidity. Antibodies (Abs) that bind epitopes conserved in both ...Phylogenetically and antigenically distinct influenza A and B viruses (IAV and IBV) circulate in human populations, causing widespread morbidity. Antibodies (Abs) that bind epitopes conserved in both IAV and IBV hemagglutinins (HAs) could protect against disease by diverse virus subtypes. Only one reported HA Ab, isolated from a combinatorial display library, protects against both IAV and IBV. Thus, there has been so far no information on the likelihood of finding naturally occurring human Abs that bind HAs of diverse IAV subtypes and IBV lineages. We have now recovered from several unrelated human donors five clonal Abs that bind a conserved epitope preferentially exposed in the postfusion conformation of IAV and IVB HA2. These Abs lack neutralizing activity in vitro but in mice provide strong, IgG subtype-dependent protection against lethal IAV and IBV infections. Strategies to elicit similar Abs routinely might contribute to more effective influenza vaccines. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42149.map.gz emd_42149.map.gz | 59.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42149-v30.xml emd-42149-v30.xml emd-42149.xml emd-42149.xml | 18.8 KB 18.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_42149_fsc.xml emd_42149_fsc.xml | 8.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_42149.png emd_42149.png | 63.5 KB | ||
| Filedesc metadata |  emd-42149.cif.gz emd-42149.cif.gz | 6.3 KB | ||
| その他 |  emd_42149_half_map_1.map.gz emd_42149_half_map_1.map.gz emd_42149_half_map_2.map.gz emd_42149_half_map_2.map.gz | 59.4 MB 59.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42149 http://ftp.pdbj.org/pub/emdb/structures/EMD-42149 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42149 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42149 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8udgMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42149.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42149.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM map of S1V2-72 Fab bound to EHA2 from influenza B/Malaysia/2506/2004 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.54688 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: cryoEM Half-map A
| ファイル | emd_42149_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cryoEM Half-map A | ||||||||||||
| 投影像・断面図 |
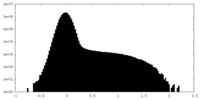
| ||||||||||||
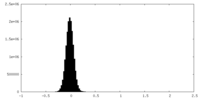
| 密度ヒストグラム |
-ハーフマップ: cryoEM Half-map B
| ファイル | emd_42149_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cryoEM Half-map B | ||||||||||||
| 投影像・断面図 |
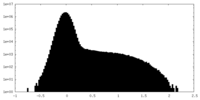
| ||||||||||||
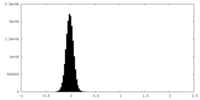
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of two S1V2-72 Fabs with postfusion HA2
| 全体 | 名称: Complex of two S1V2-72 Fabs with postfusion HA2 |
|---|---|
| 要素 |
|
-超分子 #1: Complex of two S1V2-72 Fabs with postfusion HA2
| 超分子 | 名称: Complex of two S1V2-72 Fabs with postfusion HA2 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #2-#3, #1 詳細: Fab fragment generated by proteolytic cleavage of S1V2-72 IgG |
|---|---|
| 由来(天然) | 生物種:  Influenza B virus (B/Malaysia/2506/2004) (B型インフルエンザウイルス) Influenza B virus (B/Malaysia/2506/2004) (B型インフルエンザウイルス) |
-分子 #1: Hemagglutinin
| 分子 | 名称: Hemagglutinin / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Influenza B virus (B/Malaysia/2506/2004) (B型インフルエンザウイルス) Influenza B virus (B/Malaysia/2506/2004) (B型インフルエンザウイルス) |
| 分子量 | 理論値: 17.16916 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GYTSHGAHGV AVAADLKSTQ EAINKITKNL NSLSELEVKN LQRLSGAMDE IHNEILELDE KVDDLRADTI SSQIELAVLL SNEGIINSE DEHLLALERK LKKMLGPSAV DIGNGCFETK HKCNQTCLDR IAAGTFNAGE FSLPTFDSLN ITAASLNDDG UniProtKB: Hemagglutinin |
-分子 #2: S1V2-72 heavy chain
| 分子 | 名称: S1V2-72 heavy chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 24.135121 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVQLVQSGAE LKKPGASVKV SCKASGYTFT GNYIHWMRQV PGQGLEWMGW INPRTGDTHH AQKFQGRVDM TRDTSINTAY LELTRLESD DTALYYCARC VFATSQFDPW GQGTLVTVSS ASTKGPSVFP LAPSSKSTSG GTAALGCLVK DYFPEPVTVS W NSGALTSG ...文字列: QVQLVQSGAE LKKPGASVKV SCKASGYTFT GNYIHWMRQV PGQGLEWMGW INPRTGDTHH AQKFQGRVDM TRDTSINTAY LELTRLESD DTALYYCARC VFATSQFDPW GQGTLVTVSS ASTKGPSVFP LAPSSKSTSG GTAALGCLVK DYFPEPVTVS W NSGALTSG VHTFPAVLQS SGLYSLSSVV TVPSSSLGTQ TYICNVNHKP SNTKVDKRVE PKSCDK |
-分子 #3: S1V2-72 light chain
| 分子 | 名称: S1V2-72 light chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 22.755045 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QSALTQPASV SGSPGQSITI SCTGTNSDIG SHNLVSWYQQ HPGKAPKVMI YDDSKRPSGV SNRFSGSKSG STASLTISGL QSEDEADYY CCSYAGSSNW VFGGGTKLTL LGQPKAAPSV TLFPPSSEEL QANKATLVCL ISDFYPGAVT VAWKADSSPV K AGVETTTP ...文字列: QSALTQPASV SGSPGQSITI SCTGTNSDIG SHNLVSWYQQ HPGKAPKVMI YDDSKRPSGV SNRFSGSKSG STASLTISGL QSEDEADYY CCSYAGSSNW VFGGGTKLTL LGQPKAAPSV TLFPPSSEEL QANKATLVCL ISDFYPGAVT VAWKADSSPV K AGVETTTP SKQSNNKYAA SSYLSLTPEQ WKSHRSYSCQ VTHEGSTVEK TVAPTECS |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: Quantifoil R0.6/1 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 76.12 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 105000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 得られたモデル |  PDB-8udg: |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)