+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the phage immune evasion protein Gad1 bound to the Gabija GajAB complex | ||||||||||||||||||
 マップデータ マップデータ | Main cryo-EM Map | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | viral immune evasion / phage / bacteria / anti-phage defense complex / DNA nuclease / DNA helicase / VIRAL PROTEIN | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報DNA helicase complex / recombinational repair / 3'-5' DNA helicase activity / endonuclease activity / defense response to virus / 加水分解酵素; エステル加水分解酵素 / hydrolase activity / DNA binding / ATP binding / metal ion binding / cytosol 類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |  Bacillus phage phi3T (ファージ) / Bacillus phage phi3T (ファージ) /  | ||||||||||||||||||
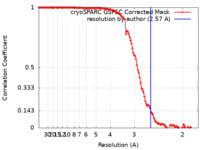
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.57 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Antine SP / Johnson AG / Mooney SE / Mayer ML / Kranzsuch PJ | ||||||||||||||||||
| 資金援助 |  米国, 5件 米国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2024 ジャーナル: Nature / 年: 2024タイトル: Structural basis of Gabija anti-phage defence and viral immune evasion. 著者: Sadie P Antine / Alex G Johnson / Sarah E Mooney / Azita Leavitt / Megan L Mayer / Erez Yirmiya / Gil Amitai / Rotem Sorek / Philip J Kranzusch /   要旨: Bacteria encode hundreds of diverse defence systems that protect them from viral infection and inhibit phage propagation. Gabija is one of the most prevalent anti-phage defence systems, occurring in ...Bacteria encode hundreds of diverse defence systems that protect them from viral infection and inhibit phage propagation. Gabija is one of the most prevalent anti-phage defence systems, occurring in more than 15% of all sequenced bacterial and archaeal genomes, but the molecular basis of how Gabija defends cells from viral infection remains poorly understood. Here we use X-ray crystallography and cryo-electron microscopy (cryo-EM) to define how Gabija proteins assemble into a supramolecular complex of around 500 kDa that degrades phage DNA. Gabija protein A (GajA) is a DNA endonuclease that tetramerizes to form the core of the anti-phage defence complex. Two sets of Gabija protein B (GajB) dimers dock at opposite sides of the complex and create a 4:4 GajA-GajB assembly (hereafter, GajAB) that is essential for phage resistance in vivo. We show that a phage-encoded protein, Gabija anti-defence 1 (Gad1), directly binds to the Gabija GajAB complex and inactivates defence. A cryo-EM structure of the virally inhibited state shows that Gad1 forms an octameric web that encases the GajAB complex and inhibits DNA recognition and cleavage. Our results reveal the structural basis of assembly of the Gabija anti-phage defence complex and define a unique mechanism of viral immune evasion. #1:  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Phages overcome bacterial immunity via diverse anti-defense proteins 著者: Yirmiya E / Leavitt A / Lu A / Avraham C / Osterman I / Garb J / Antine SP / Mooney SE / Hobbs SJ / Kranzusch PJ / Amitai G / Sorek R | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41983.map.gz emd_41983.map.gz | 80.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41983-v30.xml emd-41983-v30.xml emd-41983.xml emd-41983.xml | 20.6 KB 20.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_41983_fsc.xml emd_41983_fsc.xml | 11.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_41983.png emd_41983.png | 96.6 KB | ||
| Filedesc metadata |  emd-41983.cif.gz emd-41983.cif.gz | 6.8 KB | ||
| その他 |  emd_41983_half_map_1.map.gz emd_41983_half_map_1.map.gz emd_41983_half_map_2.map.gz emd_41983_half_map_2.map.gz | 151.7 MB 151.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41983 http://ftp.pdbj.org/pub/emdb/structures/EMD-41983 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41983 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41983 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8u7iMC  8sm3C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41983.map.gz / 形式: CCP4 / 大きさ: 163.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41983.map.gz / 形式: CCP4 / 大きさ: 163.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Main cryo-EM Map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.89 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half map B
| ファイル | emd_41983_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half Map A
| ファイル | emd_41983_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Phage Phi3T Gad1 bound to the Bacillus cereus Gabija GajAB complex
| 全体 | 名称: Phage Phi3T Gad1 bound to the Bacillus cereus Gabija GajAB complex |
|---|---|
| 要素 |
|
-超分子 #1: Phage Phi3T Gad1 bound to the Bacillus cereus Gabija GajAB complex
| 超分子 | 名称: Phage Phi3T Gad1 bound to the Bacillus cereus Gabija GajAB complex タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Bacillus phage phi3T (ファージ) Bacillus phage phi3T (ファージ) |
| 分子量 | 理論値: 280 KDa |
-超分子 #2: Gabija GajAB complex
| 超分子 | 名称: Gabija GajAB complex / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: Gad1 Octamer
| 超分子 | 名称: Gad1 Octamer / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #3 |
|---|---|
| 由来(天然) | 生物種:  Bacillus phage phi3T (ファージ) Bacillus phage phi3T (ファージ) |
-分子 #1: Endonuclease GajA
| 分子 | 名称: Endonuclease GajA / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 78.045656 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH GSGVKTENND HINLKVAGQD GSVVQFKIKR HTPLSKLMKA YCERQGLSMR QIRFRFDGQP INETDTPAQL EMEDEDTID VFQQQTGGSK FSNITIKNFR NFEKVNINLD NKNVIFGMND IGKTNFLYAL RFLLDKEIRK FGFNKSDYHK H DTSKKIEI ...文字列: MGSSHHHHHH GSGVKTENND HINLKVAGQD GSVVQFKIKR HTPLSKLMKA YCERQGLSMR QIRFRFDGQP INETDTPAQL EMEDEDTID VFQQQTGGSK FSNITIKNFR NFEKVNINLD NKNVIFGMND IGKTNFLYAL RFLLDKEIRK FGFNKSDYHK H DTSKKIEI ILTLDLSNYE KDEDTKKLIS VVKGARTSAN ADVFYIALES KYDDKELYGN IILKWGSELD NLIDIPGRGN IN ALDNVFK VIYINPLVDL DKLFAQNKKY IFEESQGNES DEGILNNIKS LTDQVNQQIG EMTIIKGFQQ EITSEYRSLK KEE VSIELK SEMAIKGFFS DIIPYIKKDG DSNYYPTSGD GRRKMLSYSI YNYLAKKKYE DKIVIYLIEE PEISLHRSMQ IALS KQLFE QSTYKYFFLS THSPELLYEM DNTRLIRVHS TEKVVCSSHM YNVEEAYGSV KKKLNKALSS ALFAERVLLI EGPSE KILF EKVLDEVEPE YELNGGFLLE VGGTYFNHYV CTLNDLGITH IIKTDNDLKS KKGKKGVYEL LGLNRCLNLL GRENLD EIT IDIPEDIKGK KKKERLNERK KEIFKQYKNE VGEFLGERIY LSEIDLENDL YSAIGESMKR IFENEDPVHY LQKSKLF NM VELVNNLSTK DCFDVFEHEK FACLKELVGS DRG UniProtKB: Endonuclease GajA |
-分子 #2: Gabija protein GajB
| 分子 | 名称: Gabija protein GajB / タイプ: protein_or_peptide / ID: 2 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 57.139992 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSREQIIKDG GNILVTAGAG SGKTTILVSK IEADLKENKT HYSIAAVTFT NKAAKEIEGR LGYSSRGNFI GTNDGFVESE IIRPFIKDA FGNDYPDNFT AEYFDNQFAS YDKGLQVLKY QNILGTYSNP KKNFKFQLAL DILKKSLVAR QYIFSKYFKI F IDEYQDSD ...文字列: MSREQIIKDG GNILVTAGAG SGKTTILVSK IEADLKENKT HYSIAAVTFT NKAAKEIEGR LGYSSRGNFI GTNDGFVESE IIRPFIKDA FGNDYPDNFT AEYFDNQFAS YDKGLQVLKY QNILGTYSNP KKNFKFQLAL DILKKSLVAR QYIFSKYFKI F IDEYQDSD KDMHNLFMYL KDQLKIKLFI VGDPKQSIYI WRGAEPENFN GLIENSTDFN KYHLTSNFRC CQDIQNYSNL FN EETRSLI KEKNEVQNVI SIADDMPISD ILLKLTEEKQ VLNIEAELVI LVRRRNQAIE IMKELNEEGF NFIFIPQTPL DRA TPNATL LKEVIKYVKN DRYSIYDLAA EIVGNLSSRE IKEIQKIINE LLVPNINQVL INQVLINLFA KLEITLDTRE ITAF TEVMM TNEFDIAFDT NEYLHKIFTV HSAKGLEFNQ VIITASDYNV HYNRDTNEHY VATTRAKDKL IVIMDNKKYS DYIET LMKE LKIKNIIKSI UniProtKB: Gabija protein GajB |
-分子 #3: Gabija Anti-Defense 1
| 分子 | 名称: Gabija Anti-Defense 1 / タイプ: protein_or_peptide / ID: 3 / コピー数: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Bacillus phage phi3T (ファージ) Bacillus phage phi3T (ファージ) |
| 分子量 | 理論値: 34.919184 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKLIGIKTSN CFLVSDNIEG KRYFHSQLDE LLFDGKRATE TYKSDWFKLE KEPSVIEKQM PAKKINHRYE LKEGFQESEL TPKVIKASY IGEDSEYYEV KGLYDLKFEE IPQQNEKIEF EMNVIEEIDG ELKLQSHNFN LNYNLLDRIQ THPMLLETKP C YLSQEESY ...文字列: MKLIGIKTSN CFLVSDNIEG KRYFHSQLDE LLFDGKRATE TYKSDWFKLE KEPSVIEKQM PAKKINHRYE LKEGFQESEL TPKVIKASY IGEDSEYYEV KGLYDLKFEE IPQQNEKIEF EMNVIEEIDG ELKLQSHNFN LNYNLLDRIQ THPMLLETKP C YLSQEESY KIIRNHIKAN INPKFARITS DYDFCLTVVK VLELYKPHEY IVDLNAMYKR RKPKLEKRFQ TKREVEIYKV AP KAYQSYP IVEPFSGKDV EDLKSNIKKF LDDLMAKINE PLVECKCCKG RGVILNEN UniProtKB: Transposase |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | 3D array |
- 試料調製
試料調製
| 濃度 | 1.00 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 20 mM HEPES-KOH pH 7.5, 20 mM KCl, and 1 mM TCEP-KOH |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 41.1 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 1.9000000000000001 µm 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X