+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the HSP90 dimer (NTD-MD) in the semi-open state | |||||||||
 マップデータ マップデータ | Sharpened Map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | HSP / HSP90 / CHAPERONE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ATP-dependent protein folding chaperone / unfolded protein binding / ATP hydrolysis activity / ATP binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) | |||||||||
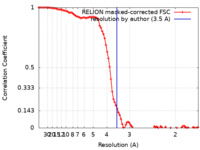
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Finci LI / Simanshu DK | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2024 ジャーナル: Commun Biol / 年: 2024タイトル: Structural dynamics of RAF1-HSP90-CDC37 and HSP90 complexes reveal asymmetric client interactions and key structural elements. 著者: Lorenzo I Finci / Mayukh Chakrabarti / Gulcin Gulten / Joseph Finney / Carissa Grose / Tara Fox / Renbin Yang / Dwight V Nissley / Frank McCormick / Dominic Esposito / Trent E Balius / Dhirendra K Simanshu /  要旨: RAF kinases are integral to the RAS-MAPK signaling pathway, and proper RAF1 folding relies on its interaction with the chaperone HSP90 and the cochaperone CDC37. Understanding the intricate molecular ...RAF kinases are integral to the RAS-MAPK signaling pathway, and proper RAF1 folding relies on its interaction with the chaperone HSP90 and the cochaperone CDC37. Understanding the intricate molecular interactions governing RAF1 folding is crucial for comprehending this process. Here, we present a cryo-EM structure of the closed-state RAF1-HSP90-CDC37 complex, where the C-lobe of the RAF1 kinase domain binds to one side of the HSP90 dimer, and an unfolded N-lobe segment of the RAF1 kinase domain threads through the center of the HSP90 dimer. CDC37 binds to the kinase C-lobe, mimicking the N-lobe with its HxNI motif. We also describe structures of HSP90 dimers without RAF1 and CDC37, displaying only N-terminal and middle domains, which we term the semi-open state. Employing 1 μs atomistic simulations, energetic decomposition, and comparative structural analysis, we elucidate the dynamics and interactions within these complexes. Our quantitative analysis reveals that CDC37 bridges the HSP90-RAF1 interaction, RAF1 binds HSP90 asymmetrically, and that HSP90 structural elements engage RAF1's unfolded region. Additionally, N- and C-terminal interactions stabilize HSP90 dimers, and molecular interactions in HSP90 dimers rearrange between the closed and semi-open states. Our findings provide valuable insight into the contributions of HSP90 and CDC37 in mediating client folding. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41817.map.gz emd_41817.map.gz | 31.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41817-v30.xml emd-41817-v30.xml emd-41817.xml emd-41817.xml | 20.8 KB 20.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_41817_fsc.xml emd_41817_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_41817.png emd_41817.png | 31.4 KB | ||
| Filedesc metadata |  emd-41817.cif.gz emd-41817.cif.gz | 6.2 KB | ||
| その他 |  emd_41817_additional_1.map.gz emd_41817_additional_1.map.gz emd_41817_half_map_1.map.gz emd_41817_half_map_1.map.gz emd_41817_half_map_2.map.gz emd_41817_half_map_2.map.gz | 49.2 MB 49.6 MB 49.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41817 http://ftp.pdbj.org/pub/emdb/structures/EMD-41817 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41817 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41817 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_41817_validation.pdf.gz emd_41817_validation.pdf.gz | 644.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_41817_full_validation.pdf.gz emd_41817_full_validation.pdf.gz | 644.3 KB | 表示 | |
| XML形式データ |  emd_41817_validation.xml.gz emd_41817_validation.xml.gz | 16.1 KB | 表示 | |
| CIF形式データ |  emd_41817_validation.cif.gz emd_41817_validation.cif.gz | 21.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41817 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41817 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41817 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41817 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41817.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41817.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
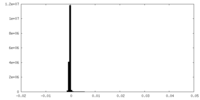
| 注釈 | Sharpened Map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.858 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Unsharpened Map
| ファイル | emd_41817_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
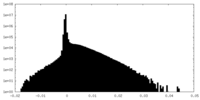
| 注釈 | Unsharpened Map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Unfiltered, unsharpened half map 1
| ファイル | emd_41817_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
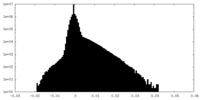
| 注釈 | Unfiltered, unsharpened half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Unfiltered, unsharpened half map 2
| ファイル | emd_41817_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
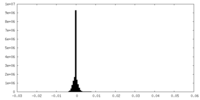
| 注釈 | Unfiltered, unsharpened half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Heat shock protein 90
| 全体 | 名称: Heat shock protein 90 |
|---|---|
| 要素 |
|
-超分子 #1: Heat shock protein 90
| 超分子 | 名称: Heat shock protein 90 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 分子量 | 理論値: 166 KDa |
-分子 #1: Heat shock protein 83
| 分子 | 名称: Heat shock protein 83 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 分子量 | 理論値: 83.322133 KDa |
| 配列 | 文字列: MPEEMQTDSG EVETFAFQAE IAQLMSLIIN TFYSNKEIFL RELISNSSDA LDKIRYESLT DPSKLDSGKE LYIKIIPNKS EGTFTIIDT GIGMTKADLV NNLGTIAKSG TKAFMEALQA GADISMIGQF GVGFYSCYLV ADRVTVHSKH NDDEQYMWES S AGGSFTVR ...文字列: MPEEMQTDSG EVETFAFQAE IAQLMSLIIN TFYSNKEIFL RELISNSSDA LDKIRYESLT DPSKLDSGKE LYIKIIPNKS EGTFTIIDT GIGMTKADLV NNLGTIAKSG TKAFMEALQA GADISMIGQF GVGFYSCYLV ADRVTVHSKH NDDEQYMWES S AGGSFTVR TDHGEPLGRG TKIVLHIKED LAEYLEVNKI KEIVKKHSQF IGYPIKLTVE KEREKELAYD EEEEKKEGEE DK KEDEKED EKPKIEDVGE DDEEDKDKKK KKTIKEKYTE DEELNKTKPI WTRNADDITQ EEYGDFYKSL TNDWEDHLAV KHF SVEGQL EFRALLFVPR RAPFDLFENK KRKNNIKLYV RRVFIMDNCE DLIPEYLNFI KGVVDSEDLP LNISREMLQQ NKIL KVIRK NLVKKCLELF EELAEDKENY KKYYEQFSKN LKLGIHEDAQ NRTKLADLLR YHTSASGDEA CSLKEYVSRM KENQK HIYY ITGENRDQVA NSSFVERVKK RGYEVVYMTE PIDEYVVQQM REYDGKTLVS VTKEGLELPE DEEEKKKREE DKVKFE GLC KVMKNILDNK VEKVVVSNRL VESPCCIVTA QYGWSANMER IMKAQALRDT STMGYMAAKK HLEINPDHSI VETLRQK AE ADKNDKAVKD LVILLYETAL LSSGFTLDEP QVHASRIYRM IKLGLGIDED EPIQVEESSV GDVPPLEGDA DDASRMEE V D UniProtKB: Heat shock protein 83 |
-分子 #2: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 2 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.2 mg/mL | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.3 構成要素:
| |||||||||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR | |||||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV 詳細: Grids were blotted for 4.5 seconds before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)