+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of HGSNAT-acetyl-CoA complex at pH 7.5 | |||||||||
 マップデータ マップデータ | Sharpened map resulting from Non-Uniform refinement (C2) in cryoSPARC. This is the primary map used for model building and PDB validation. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Heparan-alpha-glucosaminide N-acetyltransferase / Transmembrane protein 76 / membrane protein / TRANSFERASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報heparan-alpha-glucosaminide N-acetyltransferase / heparan-alpha-glucosaminide N-acetyltransferase activity / MPS IIIC - Sanfilippo syndrome C / heparan sulfate proteoglycan catabolic process / HS-GAG degradation / lysosomal transport / acyltransferase activity / tertiary granule membrane / specific granule membrane / lysosomal lumen ...heparan-alpha-glucosaminide N-acetyltransferase / heparan-alpha-glucosaminide N-acetyltransferase activity / MPS IIIC - Sanfilippo syndrome C / heparan sulfate proteoglycan catabolic process / HS-GAG degradation / lysosomal transport / acyltransferase activity / tertiary granule membrane / specific granule membrane / lysosomal lumen / protein complex oligomerization / lysosomal membrane / Neutrophil degranulation / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
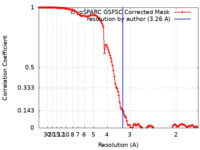
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.26 Å | |||||||||
 データ登録者 データ登録者 | Navratna V / Kumar A / Mosalaganti S | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 | ジャーナル: bioRxiv / 年: 2024 タイトル: Structure of the human heparan-α-glucosaminide -acetyltransferase (HGSNAT). 著者: Vikas Navratna / Arvind Kumar / Jaimin K Rana / Shyamal Mosalaganti /  要旨: Degradation of heparan sulfate (HS), a glycosaminoglycan (GAG) comprised of repeating units of -acetylglucosamine and glucuronic acid, begins in the cytosol and is completed in the lysosomes. ...Degradation of heparan sulfate (HS), a glycosaminoglycan (GAG) comprised of repeating units of -acetylglucosamine and glucuronic acid, begins in the cytosol and is completed in the lysosomes. Acetylation of the terminal non-reducing amino group of a-D-glucosamine of HS is essential for its complete breakdown into monosaccharides and free sulfate. Heparan-a-glucosaminide -acetyltransferase (HGSNAT), a resident of the lysosomal membrane, catalyzes this essential acetylation reaction by accepting and transferring the acetyl group from cytosolic acetyl-CoA to terminal a-D-glucosamine of HS in the lysosomal lumen. Mutation-induced dysfunction in HGSNAT causes abnormal accumulation of HS within the lysosomes and leads to an autosomal recessive neurodegenerative lysosomal storage disorder called mucopolysaccharidosis IIIC (MPS IIIC). There are no approved drugs or treatment strategies to cure or manage the symptoms of, MPS IIIC. Here, we use cryo-electron microscopy (cryo-EM) to determine a high-resolution structure of the HGSNAT-acetyl-CoA complex, the first step in HGSNAT catalyzed acetyltransferase reaction. In addition, we map the known MPS IIIC mutations onto the structure and elucidate the molecular basis for mutation-induced HGSNAT dysfunction. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41620.map.gz emd_41620.map.gz | 167.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41620-v30.xml emd-41620-v30.xml emd-41620.xml emd-41620.xml | 43 KB 43 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_41620_fsc.xml emd_41620_fsc.xml | 11.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_41620.png emd_41620.png | 89.4 KB | ||
| Filedesc metadata |  emd-41620.cif.gz emd-41620.cif.gz | 7.8 KB | ||
| その他 |  emd_41620_additional_1.map.gz emd_41620_additional_1.map.gz emd_41620_additional_10.map.gz emd_41620_additional_10.map.gz emd_41620_additional_11.map.gz emd_41620_additional_11.map.gz emd_41620_additional_2.map.gz emd_41620_additional_2.map.gz emd_41620_additional_3.map.gz emd_41620_additional_3.map.gz emd_41620_additional_4.map.gz emd_41620_additional_4.map.gz emd_41620_additional_5.map.gz emd_41620_additional_5.map.gz emd_41620_additional_6.map.gz emd_41620_additional_6.map.gz emd_41620_additional_7.map.gz emd_41620_additional_7.map.gz emd_41620_additional_8.map.gz emd_41620_additional_8.map.gz emd_41620_additional_9.map.gz emd_41620_additional_9.map.gz emd_41620_half_map_1.map.gz emd_41620_half_map_1.map.gz emd_41620_half_map_2.map.gz emd_41620_half_map_2.map.gz | 786.6 KB 791.2 KB 779.1 KB 779.1 KB 779.1 KB 167.7 MB 164.8 MB 779.1 KB 164.8 MB 88.5 MB 165.2 MB 164.8 MB 164.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41620 http://ftp.pdbj.org/pub/emdb/structures/EMD-41620 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41620 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41620 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_41620_validation.pdf.gz emd_41620_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_41620_full_validation.pdf.gz emd_41620_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_41620_validation.xml.gz emd_41620_validation.xml.gz | 20.7 KB | 表示 | |
| CIF形式データ |  emd_41620_validation.cif.gz emd_41620_validation.cif.gz | 26.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41620 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41620 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41620 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41620 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8tu9MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41620.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41620.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map resulting from Non-Uniform refinement (C2) in cryoSPARC. This is the primary map used for model building and PDB validation. | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.85 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
+追加マップ: Mask (auto-tightening) used for Non-Uniform refinement in cryoSPARC.
+追加マップ: Half map (half-map A) resulting from local refinement...
+追加マップ: Half map (half-map B) resulting from local refinement...
+追加マップ: Mask used for Non-uniform refinement (C2) in cryoSPARC.
+追加マップ: Mask used for Non-Uniform refinement (C2) in cryoSPARC.
+追加マップ: Mask Used for refinement (C1) during local refinement...
+追加マップ: Unsharpened map resulting from Non-Uniform refinement (C2) in...
+追加マップ: Composite map for model validation generated from focused...
+追加マップ: Mask (auto-tightening) used for refinement during local refinement...
+追加マップ: Mask used for refinement during local refinement (C1)...
+追加マップ: Unsharpened map resulting from local refinement (C1) in...
+ハーフマップ: Half map (half-map A) resulting from Non-Uniform refinement...
+ハーフマップ: Half map (half-map B) resulting from Non-Uniform refinement...
- 試料の構成要素
試料の構成要素
-全体 : Heparan acetyl-CoA: alpha-glucosaminide N-acetyltransferase (HGSNAT)
| 全体 | 名称: Heparan acetyl-CoA: alpha-glucosaminide N-acetyltransferase (HGSNAT) |
|---|---|
| 要素 |
|
-超分子 #1: Heparan acetyl-CoA: alpha-glucosaminide N-acetyltransferase (HGSNAT)
| 超分子 | 名称: Heparan acetyl-CoA: alpha-glucosaminide N-acetyltransferase (HGSNAT) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: Full length (Isoform 2) human HGSNAT expressed as a recombinant fusion protein with N-terminal GFP, in mammalian cells. |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / Organelle: Lysosomes / 細胞中の位置: Lysosomal membrane Homo sapiens (ヒト) / Organelle: Lysosomes / 細胞中の位置: Lysosomal membrane |
| 分子量 | 理論値: 100.7 KDa |
-分子 #1: Enhanced green fluorescent protein,Heparan-alpha-glucosaminide N-...
| 分子 | 名称: Enhanced green fluorescent protein,Heparan-alpha-glucosaminide N-acetyltransferase,Isoform 2 of Heparan-alpha-glucosaminide N-acetyltransferase タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: heparan-alpha-glucosaminide N-acetyltransferase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 100.817172 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MWSHPQFEKG SGVSKGEELF TGVVPILVEL DGDVNGHKFS VSGEGEGDAT YGKLTLKFIC TTGKLPVPWP TLVTTLTYGV QCFSRYPDH MKQHDFFKSA MPEGYVQERT IFFKDDGNYK TRAEVKFEGD TLVNRIELKG IDFKEDGNIL GHKLEYNYNS H NVYIMADK ...文字列: MWSHPQFEKG SGVSKGEELF TGVVPILVEL DGDVNGHKFS VSGEGEGDAT YGKLTLKFIC TTGKLPVPWP TLVTTLTYGV QCFSRYPDH MKQHDFFKSA MPEGYVQERT IFFKDDGNYK TRAEVKFEGD TLVNRIELKG IDFKEDGNIL GHKLEYNYNS H NVYIMADK QKNGIKVNFK IRHNIEDGSV QLADHYQQNT PIGDGPVLLP DNHYLSTQSK LSKDPNEKRD HMVLLEFVTA AG ITLGMDE LYKSGLRSGL EVLFQGPEFE FMSGAGRALA ALLLAASVLS AALLAPGGSS GRDAQAAPPR DLDKKRHAEL KMD QALLLI HNELLWTNLT VYWKSECCYH CLFQVLVNVP QSPKAGKPSA AAASVSTQHG SILQLNDTLE EKEVCRLEYR FGEF GNYSL LVKNIHNGVS EIACDLAVNE DPVDSNLPVS IAFLIGLAVI IVISFLRLLL SLDDFNNWIS KAISSRETDR LINSE LGSP SRTDPLDGDV QPATWRLSAL PPRLRSVDTF RGIALILMVF VNYGGGKYWY FKHASWNGLT VADLVFPWFV FIMGSS IFL SMTSILQRGC SKFRLLGKIA WRSFLLICIG IIIVNPNYCL GPLSWDKVRI PGVLQRLGVT YFVVAVLELL FAKPVPE HC ASERSCLSLR DITSSWPQWL LILVLEGLWL GLTFLLPVPG CPTGYLGPGG IGDFGKYPNC TGGAAGYIDR LLLGDDHL Y QHPSSAVLYH TEVAYDPEGI LGTINSIVMA FLGVQAGKIL LYYKARTKDI LIRFTAWCCI LGLISVALTK VSENEGFIP VNKNLWSLSY VTTLSSFAFF ILLVLYPVVD VKGLWTGTPF FYPGMNSILV YVGHEVFENY FPFQWKLKDN QSHKEHLTQN IVATALWVL IAYILYRKKI FWKI UniProtKB: Green fluorescent protein, Heparan-alpha-glucosaminide N-acetyltransferase |
-分子 #2: ACETYL COENZYME *A
| 分子 | 名称: ACETYL COENZYME *A / タイプ: ligand / ID: 2 / コピー数: 2 / 式: ACO |
|---|---|
| 分子量 | 理論値: 809.571 Da |
| Chemical component information |  ChemComp-ACO: |
-分子 #3: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 3 / コピー数: 6 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.9 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 気圧: 0.026000000000000002 kPa / 詳細: 15 mA current | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 291 K / 装置: FEI VITROBOT MARK IV | ||||||||||||
| 詳細 | HGSNAT in digitonin micelle, purified by Strep-Tactin affinity chromatography. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 2 / 実像数: 10000 / 平均露光時間: 2.0 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: Other / Chain - Initial model type: in silico model / 詳細: Made using Model angelo |
|---|---|
| 精密化 | プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-8tu9: |
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X