+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human VPS35L/VPS29/VPS26C Complex | |||||||||
 マップデータ マップデータ | Retriever VPS35L/VPS29/VPS26C Complex | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | COMMD / Retriever / Commander / CCC / PROTEIN TRANSPORT | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報retromer, cargo-selective complex / WNT ligand biogenesis and trafficking / retromer complex / Golgi to plasma membrane transport / endocytic recycling / retrograde transport, endosome to Golgi / ficolin-1-rich granule membrane / intracellular protein transport / protein transport / late endosome ...retromer, cargo-selective complex / WNT ligand biogenesis and trafficking / retromer complex / Golgi to plasma membrane transport / endocytic recycling / retrograde transport, endosome to Golgi / ficolin-1-rich granule membrane / intracellular protein transport / protein transport / late endosome / early endosome / endosome membrane / endosome / intracellular membrane-bounded organelle / Neutrophil degranulation / nucleus / metal ion binding / plasma membrane / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
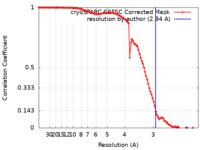
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.94 Å | |||||||||
 データ登録者 データ登録者 | Chen Z / Chen B / Burstein E / Han Y | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: Structural organization of the retriever-CCC endosomal recycling complex. 著者: Daniel J Boesch / Amika Singla / Yan Han / Daniel A Kramer / Qi Liu / Kohei Suzuki / Puneet Juneja / Xuefeng Zhao / Xin Long / Michael J Medlyn / Daniel D Billadeau / Zhe Chen / Baoyu Chen / Ezra Burstein /  要旨: The recycling of membrane proteins from endosomes to the cell surface is vital for cell signaling and survival. Retriever, a trimeric complex of vacuolar protein-sorting-associated protein (VPS)35L, ...The recycling of membrane proteins from endosomes to the cell surface is vital for cell signaling and survival. Retriever, a trimeric complex of vacuolar protein-sorting-associated protein (VPS)35L, VPS26C and VPS29, together with the CCC complex comprising coiled-coil domain-containing (CCDC)22, CCDC93 and copper metabolism domain-containing (COMMD) proteins, plays a crucial role in this process. The precise mechanisms underlying retriever assembly and its interaction with CCC have remained elusive. Here, we present a high-resolution structure of retriever in humans determined using cryogenic electron microscopy. The structure reveals a unique assembly mechanism, distinguishing it from its remotely related paralog retromer. By combining AlphaFold predictions and biochemical, cellular and proteomic analyses, we further elucidate the structural organization of the entire retriever-CCC complex across evolution and uncover how cancer-associated mutations in humans disrupt complex formation and impair membrane protein homeostasis. These findings provide a fundamental framework for understanding the biological and pathological implications associated with retriever-CCC-mediated endosomal recycling. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40885.map.gz emd_40885.map.gz | 91.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40885-v30.xml emd-40885-v30.xml emd-40885.xml emd-40885.xml | 23 KB 23 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_40885_fsc.xml emd_40885_fsc.xml | 10.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_40885.png emd_40885.png | 58.4 KB | ||
| マスクデータ |  emd_40885_msk_1.map emd_40885_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-40885.cif.gz emd-40885.cif.gz | 7.3 KB | ||
| その他 |  emd_40885_half_map_1.map.gz emd_40885_half_map_1.map.gz emd_40885_half_map_2.map.gz emd_40885_half_map_2.map.gz | 95.5 MB 95.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40885 http://ftp.pdbj.org/pub/emdb/structures/EMD-40885 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40885 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40885 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_40885_validation.pdf.gz emd_40885_validation.pdf.gz | 655.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_40885_full_validation.pdf.gz emd_40885_full_validation.pdf.gz | 655 KB | 表示 | |
| XML形式データ |  emd_40885_validation.xml.gz emd_40885_validation.xml.gz | 18 KB | 表示 | |
| CIF形式データ |  emd_40885_validation.cif.gz emd_40885_validation.cif.gz | 23.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40885 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40885 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40885 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40885 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40885.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40885.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Retriever VPS35L/VPS29/VPS26C Complex | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.0624 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_40885_msk_1.map emd_40885_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half Map 1
| ファイル | emd_40885_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half Map 2
| ファイル | emd_40885_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Trimeric Complex of full-length VPS35L/VPS29/VPS26C
| 全体 | 名称: Trimeric Complex of full-length VPS35L/VPS29/VPS26C |
|---|---|
| 要素 |
|
-超分子 #1: Trimeric Complex of full-length VPS35L/VPS29/VPS26C
| 超分子 | 名称: Trimeric Complex of full-length VPS35L/VPS29/VPS26C / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: VPS35 endosomal protein-sorting factor-like
| 分子 | 名称: VPS35 endosomal protein-sorting factor-like / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 109.700453 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAVFPWHSRN RNYKAEFASC RLEAVPLEFG DYHPLKPITV TESKTKKVNR KGSTSSTSSS SSSSVVDPLS SVLDGTDPLS MFAATADPA ALAAAMDSSR RKRDRDDNSV VGSDFEPWTN KRGEILARYT TTEKLSINLF MGSEKGKAGT ATLAMSEKVR T RLEELDDF ...文字列: MAVFPWHSRN RNYKAEFASC RLEAVPLEFG DYHPLKPITV TESKTKKVNR KGSTSSTSSS SSSSVVDPLS SVLDGTDPLS MFAATADPA ALAAAMDSSR RKRDRDDNSV VGSDFEPWTN KRGEILARYT TTEKLSINLF MGSEKGKAGT ATLAMSEKVR T RLEELDDF EEGSQKELLN LTQQDYVNRI EELNQSLKDA WASDQKVKAL KIVIQCSKLL SDTSVIQFYP SKFVLITDIL DT FGKLVYE RIFSMCVDSR SVLPDHFSPE NANDTAKETC LNWFFKIASI RELIPRFYVE ASILKCNKFL SKTGISECLP RLT CMIRGI GDPLVSVYAR AYLCRVGMEV APHLKETLNK NFFDFLLTFK QIHGDTVQNQ LVVQGVELPS YLPLYPPAMD WIFQ CISYH APEALLTEMM ERCKKLGNNA LLLNSVMSAF RAEFIATRSM DFIGMIKECD ESGFPKHLLF RSLGLNLALA DPPES DRLQ ILNEAWKVIT KLKNPQDYIN CAEVWVEYTC KHFTKREVNT VLADVIKHMT PDRAFEDSYP QLQLIIKKVI AHFHDF SVL FSVEKFLPFL DMFQKESVRV EVCKCIMDAF IKHQQEPTKD PVILNALLHV CKTMHDSVNA LTLEDEKRML SYLINGF IK MVSFGRDFEQ QLSFYVESRS MFCNLEPVLV QLIHSVNRLA METRKVMKGN HSRKTAAFVR ACVAYCFITI PSLAGIFT R LNLYLHSGQV ALANQCLSQA DAFFKAAISL VPEVPKMINI DGKMRPSESF LLEFLCNFFS TLLIVPDHPE HGVLFLVRE LLNVIQDYTW EDNSDEKIRI YTCVLHLLSA MSQETYLYHI DKVDSNDSLY GGDSKFLAEN NKLCETVMAQ ILEHLKTLAK DEALKRQSS LGLSFFNSIL AHGDLRNNKL NQLSVNLWHL AQRHGCADTR TMVKTLEYIK KQSKQPDMTH LTELALRLPL Q TRT UniProtKB: VPS35 endosomal protein-sorting factor-like |
-分子 #2: Vacuolar protein sorting-associated protein 29
| 分子 | 名称: Vacuolar protein sorting-associated protein 29 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.03832 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAGHRLVLVL GDLHIPHRCN SLPAKFKKLL VPGKIQHILC TGNLCTKESY DYLKTLAGDV HIVRGDFDEN LNYPEQKVVT VGQFKIGLI HGHQVIPWGD MASLALLQRQ FDVDILISGH THKFEAFEHE NKFYINPGSA TGAYNALETN IIPSFVLMDI Q ASTVVTYV ...文字列: MAGHRLVLVL GDLHIPHRCN SLPAKFKKLL VPGKIQHILC TGNLCTKESY DYLKTLAGDV HIVRGDFDEN LNYPEQKVVT VGQFKIGLI HGHQVIPWGD MASLALLQRQ FDVDILISGH THKFEAFEHE NKFYINPGSA TGAYNALETN IIPSFVLMDI Q ASTVVTYV YQLIGDDVKV ERIEYKKPEN LYFQGGGSGG SHHHHHH UniProtKB: Vacuolar protein sorting-associated protein 29 |
-分子 #3: Vacuolar protein sorting-associated protein 26C
| 分子 | 名称: Vacuolar protein sorting-associated protein 26C / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 33.049344 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGTALDIKIK RANKVYHAGE VLSGVVVISS KDSVQHQGVS LTMEGTVNLQ LSAKSVGVFE AFYNSVKPIQ IINSTIEMVK PGKFPSGKT EIPFEFPLHL KGNKVLYETY HGVFVNIQYT LRCDMKRSLL AKDLTKTCEF IVHSAPQKGK FTPSPVDFTI T PETLQNVK ...文字列: MGTALDIKIK RANKVYHAGE VLSGVVVISS KDSVQHQGVS LTMEGTVNLQ LSAKSVGVFE AFYNSVKPIQ IINSTIEMVK PGKFPSGKT EIPFEFPLHL KGNKVLYETY HGVFVNIQYT LRCDMKRSLL AKDLTKTCEF IVHSAPQKGK FTPSPVDFTI T PETLQNVK ERALLPKFLL RGHLNSTNCV ITQPLTGELV VESSEAAIRS VELQLVRVET CGCAEGYARD ATEIQNIQIA DG DVCRGLS VPIYMVFPRL FTCPTLETTN FKVEFEVNIV VLLHPDHLIT ENFPLKLCRI UniProtKB: Vacuolar protein sorting-associated protein 26C |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.25 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7 構成要素:
詳細: 10 mM HEPES (pH 7.0), 150 mM NaCl, 2 mM MgCl2, 2 mM DTT, and 5% (v/v) glycerol | ||||||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 278.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum ER / エネルギーフィルター - スリット幅: 20 eV |
| ソフトウェア | 名称: SerialEM |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 3594 / 平均露光時間: 5.0 sec. / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| ソフトウェア | 名称:  Coot Coot |
| 精密化 | 空間: REAL / 温度因子: 107.9 |
| 得られたモデル |  PDB-8syn: |
 ムービー
ムービー コントローラー
コントローラー









 Z
Z Y
Y X
X