+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the C-terminal protease CtpA-LbcA complex of Pseudomonas aeruginosa | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CtpA / LbcA / C-terminal protease / Pseudomonas aeruginosa / HYDROLASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   | |||||||||
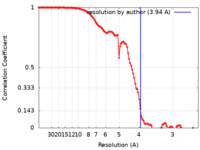
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.94 Å | |||||||||
 データ登録者 データ登録者 | Hsu H-C / Li H | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: EMBO J / 年: 2024 ジャーナル: EMBO J / 年: 2024タイトル: P. aeruginosa CtpA protease adopts a novel activation mechanism to initiate the proteolytic process. 著者: Hao-Chi Hsu / Michelle Wang / Amanda Kovach / Andrew J Darwin / Huilin Li /  要旨: During bacterial cell growth, hydrolases cleave peptide cross-links between strands of the peptidoglycan sacculus to allow new strand insertion. The Pseudomonas aeruginosa carboxyl-terminal ...During bacterial cell growth, hydrolases cleave peptide cross-links between strands of the peptidoglycan sacculus to allow new strand insertion. The Pseudomonas aeruginosa carboxyl-terminal processing protease (CTP) CtpA regulates some of these hydrolases by degrading them. CtpA assembles as an inactive hexamer composed of a trimer-of-dimers, but its lipoprotein binding partner LbcA activates CtpA by an unknown mechanism. Here, we report the cryo-EM structures of the CtpA-LbcA complex. LbcA has an N-terminal adaptor domain that binds to CtpA, and a C-terminal superhelical tetratricopeptide repeat domain. One LbcA molecule attaches to each of the three vertices of a CtpA hexamer. LbcA triggers relocation of the CtpA PDZ domain, remodeling of the substrate binding pocket, and realignment of the catalytic residues. Surprisingly, only one CtpA molecule in a CtpA dimer is activated upon LbcA binding. Also, a long loop from one CtpA dimer inserts into a neighboring dimer to facilitate the proteolytic activity. This work has revealed an activation mechanism for a bacterial CTP that is strikingly different from other CTPs that have been characterized structurally. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40852.map.gz emd_40852.map.gz | 118 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40852-v30.xml emd-40852-v30.xml emd-40852.xml emd-40852.xml | 21.7 KB 21.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_40852_fsc.xml emd_40852_fsc.xml | 11.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_40852.png emd_40852.png | 97.7 KB | ||
| マスクデータ |  emd_40852_msk_1.map emd_40852_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-40852.cif.gz emd-40852.cif.gz | 6.8 KB | ||
| その他 |  emd_40852_half_map_1.map.gz emd_40852_half_map_1.map.gz emd_40852_half_map_2.map.gz emd_40852_half_map_2.map.gz | 116 MB 116 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40852 http://ftp.pdbj.org/pub/emdb/structures/EMD-40852 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40852 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40852 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_40852_validation.pdf.gz emd_40852_validation.pdf.gz | 765.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_40852_full_validation.pdf.gz emd_40852_full_validation.pdf.gz | 765.1 KB | 表示 | |
| XML形式データ |  emd_40852_validation.xml.gz emd_40852_validation.xml.gz | 18.9 KB | 表示 | |
| CIF形式データ |  emd_40852_validation.cif.gz emd_40852_validation.cif.gz | 24.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40852 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40852 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40852 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40852 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40852.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40852.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.242 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_40852_msk_1.map emd_40852_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
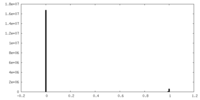
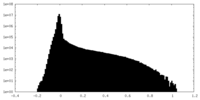
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_40852_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
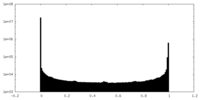
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_40852_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
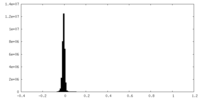
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CtpA-LbcA complex of Pseudomonas aeruginosa
| 全体 | 名称: CtpA-LbcA complex of Pseudomonas aeruginosa |
|---|---|
| 要素 |
|
-超分子 #1: CtpA-LbcA complex of Pseudomonas aeruginosa
| 超分子 | 名称: CtpA-LbcA complex of Pseudomonas aeruginosa / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 420 KDa |
-分子 #1: Carboxyl-terminal protease
| 分子 | 名称: Carboxyl-terminal protease / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 42.920438 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSHMSAPLPL DELRTFAEVL DRVKAAYVEP VDDKTLLENA IKGMLSNLDP HSAYLGPEDF AELQESTSGE FGGLGIEVGS EDGFIKVVS PIDDTPAARA GIQPGDLIVQ IDGKPTKGQS MTEAVDSMRG KAGSPITLTI VRDGGRPFDV ELKRAIIKVK S VKSQVLEP ...文字列: GSHMSAPLPL DELRTFAEVL DRVKAAYVEP VDDKTLLENA IKGMLSNLDP HSAYLGPEDF AELQESTSGE FGGLGIEVGS EDGFIKVVS PIDDTPAARA GIQPGDLIVQ IDGKPTKGQS MTEAVDSMRG KAGSPITLTI VRDGGRPFDV ELKRAIIKVK S VKSQVLEP GYAYLRITQF QVNTGEEVVK ALNQLRKDNK GRLKGLVLDL RNNPGGVLQS AVEVADAFLT KGLIVYTKGR IA NSELRFS ADPADPSDKV PLVVLINGGS AAAAEIVAGA LQDQKRAILM GTDSFGKGSV QTVLPLNNDR ALKLTTALYY TPN GRSIQA QGIVPDIEVG RAKVTQERSS FEGFKEADLQ GHLANGNGGA DRPTGKRAAP SERPQDSDYQ LSQALSLLKG LSVT RGN UniProtKB: Carboxy-terminal processing protease CtpB |
-分子 #2: TPR repeat-containing protein PA4667
| 分子 | 名称: TPR repeat-containing protein PA4667 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 61.742066 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MEDTAVETKA KPEKYGSFSE DSLYSLLVAE LAGQRNRFDI ALSNYVVQAQ KTRDPGVSER AFRIAEYLGA DQEALDTSLL WARSAPDNL DAQRAAAIQL ARAGRYEESM VYMEKVLNGQ GDTHFDFLAL SAAETDPDTR AGLLQSFDHL LKKYPNNGQL L FGKALLLQ ...文字列: MEDTAVETKA KPEKYGSFSE DSLYSLLVAE LAGQRNRFDI ALSNYVVQAQ KTRDPGVSER AFRIAEYLGA DQEALDTSLL WARSAPDNL DAQRAAAIQL ARAGRYEESM VYMEKVLNGQ GDTHFDFLAL SAAETDPDTR AGLLQSFDHL LKKYPNNGQL L FGKALLLQ QDGRPDEALT LLEDNSASRH EVAPLLLRSR LLQSMKRSDE ALPLLKAGIK EHPDDKRVRL AYARLLVEQN RL DDAKAEF AGLVQQFPDD DDLRFSLALV CLEAQAWDEA RIYLEELVER DSHVDAAHFN LGRLAEEQKD TARALDEYAQ VGP GNDFLP AQLRQTDVLL KAGRVDEAAQ RLDKARSEQP DYAIQLYLIE AEALSNNDQQ EKAWQAIQEG LKQYPEDLNL LYTR SMLAE KRNDLAQMEK DLRFVIAREP DNAMALNALG YTLADRTTRY GEARELILKA HKLNPDDPAI LDSMGWINYR QGKLA DAER YLRQALQRYP DHEVAAHLGE VLWAQGRQGD ARAIWREYLD KQPDSDVLRR TIKRLTGAET P UniProtKB: TPR repeat-containing protein PA4667 |
-分子 #3: unidentified peptide
| 分子 | 名称: unidentified peptide / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 613.749 Da |
| 組換発現 | 生物種:  |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.8 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: OTHER | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 279 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 11520 pixel / デジタル化 - サイズ - 縦: 8184 pixel / 撮影したグリッド数: 1 / 実像数: 26257 / 平均露光時間: 1.5 sec. / 平均電子線量: 66.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 1.3 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z
Z Y
Y X
X