+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | HIV-1 Env subtype C CZA97.12 SOSIP.664 in complex with 3BNC117 Fab | |||||||||
 マップデータ マップデータ | sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | HIV Envelope / SOSIP / broadly neutralizing antibody / clade C / VIRAL PROTEIN / VIRAL PROTEIN-IMMUNE SYSTEM complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane / viral envelope ...positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / apoptotic process / host cell plasma membrane / structural molecule activity / virion membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
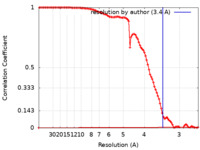
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Ozorowski G / Lee JH / Ward AB | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: PLoS Pathog / 年: 2023 ジャーナル: PLoS Pathog / 年: 2023タイトル: Glycan heterogeneity as a cause of the persistent fraction in HIV-1 neutralization. 著者: Rajesh P Ringe / Philippe Colin / Gabriel Ozorowski / Joel D Allen / Anila Yasmeen / Gemma E Seabright / Jeong Hyun Lee / Aleksandar Antanasijevic / Kimmo Rantalainen / Thomas Ketas / John P ...著者: Rajesh P Ringe / Philippe Colin / Gabriel Ozorowski / Joel D Allen / Anila Yasmeen / Gemma E Seabright / Jeong Hyun Lee / Aleksandar Antanasijevic / Kimmo Rantalainen / Thomas Ketas / John P Moore / Andrew B Ward / Max Crispin / P J Klasse /   要旨: Neutralizing antibodies (NAbs) to multiple epitopes on the HIV-1-envelope glycoprotein (Env) have been isolated from infected persons. The potency of NAbs is measured more often than the size of the ...Neutralizing antibodies (NAbs) to multiple epitopes on the HIV-1-envelope glycoprotein (Env) have been isolated from infected persons. The potency of NAbs is measured more often than the size of the persistent fraction of infectivity at maximum neutralization, which may also influence preventive efficacy of active or passive immunization and the therapeutic outcome of the latter. Many NAbs neutralize HIV-1 CZA97.012, a clone of a Clade-C isolate, to ~100%. But here NAb PGT151, directed to a fusion-peptide epitope, left a persistent fraction of 15%. NAb PGT145, ligating the Env-trimer apex, left no detectable persistent fraction. The divergence in persistent fractions was further analyzed by depletion of pseudoviral populations of the most PGT151- and PGT145-reactive virions. Thereby, neutralization by the non-depleting NAb increased, whereas neutralization by the depleting NAb decreased. Furthermore, depletion by PGT151 increased sensitivity to autologous neutralization by sera from rabbits immunized with soluble native-like CZA97.012 trimer: substantial persistent fractions were reduced. NAbs in these sera target epitopes comprising residue D411 at the V4-β19 transition in a defect of the glycan shield on CZA97.012 Env. NAb binding to affinity-fractionated soluble native-like CZA97.012 trimer differed commensurately with neutralization in analyses by ELISA and surface plasmon resonance. Glycan differences between PGT151- and PGT145-purified trimer fractions were then demonstrated by mass spectrometry, providing one explanation for the differential antigenicity. These differences were interpreted in relation to a new structure at 3.4-Å resolution of the soluble CZA97.012 trimer determined by cryo-electron microscopy. The trimer adopted a closed conformation, refuting apex opening as the cause of reduced PGT145 binding to the PGT151-purified form. The evidence suggests that differences in binding and neutralization after trimer purification or pseudovirus depletion with PGT145 or PGT151 are caused by variation in glycosylation, and that some glycan variants affect antigenicity through direct effects on antibody contacts, whereas others act allosterically. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40088.map.gz emd_40088.map.gz | 86 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40088-v30.xml emd-40088-v30.xml emd-40088.xml emd-40088.xml | 20.7 KB 20.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_40088_fsc.xml emd_40088_fsc.xml | 10.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_40088.png emd_40088.png | 58.7 KB | ||
| マスクデータ |  emd_40088_msk_1.map emd_40088_msk_1.map | 91.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-40088.cif.gz emd-40088.cif.gz | 7 KB | ||
| その他 |  emd_40088_half_map_1.map.gz emd_40088_half_map_1.map.gz emd_40088_half_map_2.map.gz emd_40088_half_map_2.map.gz | 84.5 MB 84.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40088 http://ftp.pdbj.org/pub/emdb/structures/EMD-40088 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40088 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40088 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_40088_validation.pdf.gz emd_40088_validation.pdf.gz | 903.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_40088_full_validation.pdf.gz emd_40088_full_validation.pdf.gz | 902.9 KB | 表示 | |
| XML形式データ |  emd_40088_validation.xml.gz emd_40088_validation.xml.gz | 17.9 KB | 表示 | |
| CIF形式データ |  emd_40088_validation.cif.gz emd_40088_validation.cif.gz | 23.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40088 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40088 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40088 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40088 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8gjeMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40088.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40088.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.31 Å | ||||||||||||||||||||||||||||||||||||
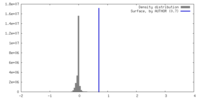
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_40088_msk_1.map emd_40088_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
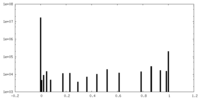
| 密度ヒストグラム |
-ハーフマップ: half map A
| ファイル | emd_40088_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
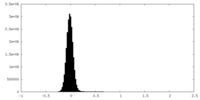
| 密度ヒストグラム |
-ハーフマップ: half map B
| ファイル | emd_40088_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
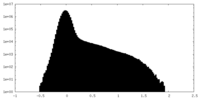
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CZA97.12 SOSIP.664 in complex with 3BNC117 Fab
| 全体 | 名称: CZA97.12 SOSIP.664 in complex with 3BNC117 Fab |
|---|---|
| 要素 |
|
-超分子 #1: CZA97.12 SOSIP.664 in complex with 3BNC117 Fab
| 超分子 | 名称: CZA97.12 SOSIP.664 in complex with 3BNC117 Fab / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 分子量 | 理論値: 570 KDa |
-分子 #1: CZA97.12 SOSIP.664 Envelope glycoprotein gp120
| 分子 | 名称: CZA97.12 SOSIP.664 Envelope glycoprotein gp120 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 56.995121 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDAMKRGLCC VLLLCGAVFV SPSQEIHARF RRGARVGNMW VTVYYGVPVW TDAKTTLFCA SDTKAYDREV HNVWATHACV PTDPNPQEI VLENVTENFN MWKNDMVDQM HEDIISLWDQ SLKPCVKLTP LCVTLHCTNA TFKNNVTNDM NKEIRNCSFN T TTEIRDKK ...文字列: MDAMKRGLCC VLLLCGAVFV SPSQEIHARF RRGARVGNMW VTVYYGVPVW TDAKTTLFCA SDTKAYDREV HNVWATHACV PTDPNPQEI VLENVTENFN MWKNDMVDQM HEDIISLWDQ SLKPCVKLTP LCVTLHCTNA TFKNNVTNDM NKEIRNCSFN T TTEIRDKK QQGYALFYRP DIVLLKENRN NSNNSEYILI NCNASTITQA CPKVNFDPIP IHYCAPAGYA ILKCNNKTFS GK GPCNNVS TVQCTHGIKP VVSTQLLLNG SLAEKEIIIR SENLTDNVKT IIVHLNKSVE IVCTRPNNNT RKSMRIGPGQ TFY ATGDII GDIRQAYCNI SGSKWNETLK RVKEKLQENY NNNKTIKFAP SSGGDLEITT HSFNCRGEFF YCNTTRLFNN NATE DETIT LPCRIKQIIN MWQGVGRAMY APPIAGNITC KSNITGLLLV RDGGEDNKTE EIFRPGGGNM KDNWRSELYK YKVIE LKPL GIAPTGCKRR VVERRRRRR UniProtKB: Envelope glycoprotein gp160 |
-分子 #2: CZA97.12 SOSIP.664 Envelope glycoprotein gp41
| 分子 | 名称: CZA97.12 SOSIP.664 Envelope glycoprotein gp41 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 17.27967 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AVGIGAVFLG FLGAAGSTMG AASMTLTVQA RQLLSSIVQQ QSNLLRAPEA QQHMLKLTVW GIKQLQTRVL AIERYLKDQQ LLGIWGCSG KLICCTNVPW NSSWSNKSQT DIWNNMTWME WDREISNYTD TIYRLLEDSQ TQQEKNEKDL LALD UniProtKB: Envelope glycoprotein gp160 |
-分子 #3: 3BNC117 Fab heavy chain
| 分子 | 名称: 3BNC117 Fab heavy chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 24.656484 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVQLLQSGAA VTKPGASVRV SCEASGYNIR DYFIHWWRQA PGQGLQWVGW INPKTGQPNN PRQFQGRVSL TRHASWDFDT YSFYMDLKA LRSDDTAVYF CARQRSDYWD FDVWGSGTQV TVSSASTKGP SVFPLAPSSK STSGGTAALG CLVKDYFPEP V TVSWNSGA ...文字列: QVQLLQSGAA VTKPGASVRV SCEASGYNIR DYFIHWWRQA PGQGLQWVGW INPKTGQPNN PRQFQGRVSL TRHASWDFDT YSFYMDLKA LRSDDTAVYF CARQRSDYWD FDVWGSGTQV TVSSASTKGP SVFPLAPSSK STSGGTAALG CLVKDYFPEP V TVSWNSGA LTSGVHTFPA VLQSSGLYSL SSVVTVPSSS LGTQTYICNV NHKPSNTKVD KKVEPKSC |
-分子 #4: 3BNC117 kappa light chain
| 分子 | 名称: 3BNC117 kappa light chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.022658 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DIQMTQSPSS LSASVGDTVT ITCQANGYLN WYQQRRGKAP KLLIYDGSKL ERGVPSRFSG RRWGQEYNLT INNLQPEDIA TYFCQVYEF VVPGTRLDLK RTVAAPSVFI FPPSDEQLKS GTASVVCLLN NFYPREAKVQ WKVDNALQSG NSQESVTEQD S KDSTYSLS ...文字列: DIQMTQSPSS LSASVGDTVT ITCQANGYLN WYQQRRGKAP KLLIYDGSKL ERGVPSRFSG RRWGQEYNLT INNLQPEDIA TYFCQVYEF VVPGTRLDLK RTVAAPSVFI FPPSDEQLKS GTASVVCLLN NFYPREAKVQ WKVDNALQSG NSQESVTEQD S KDSTYSLS STLTLSKADY EKHKVYACEV THQGLSSPVT KSFNRGEC |
-分子 #9: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 9 / コピー数: 33 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
詳細: Detergent added shortly before freezing | ||||||||||||
| グリッド | モデル: C-flat-2/2 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING | ||||||||||||
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 実像数: 1142 / 平均電子線量: 58.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)