+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Mycobacterium phage Patience | |||||||||
 マップデータ マップデータ | Sharpened map of ewald sphere corrected postprocess. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | HK97-fold / T=7 / tailed bacteriophage / VIRUS | |||||||||
| 機能・相同性 | Uncharacterized protein / Uncharacterized protein / Capsid protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Mycobacterium phage Patience (ファージ) Mycobacterium phage Patience (ファージ) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.39 Å | |||||||||
 データ登録者 データ登録者 | Podgorski JM / White SJ | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: A novel accessory protein stabilizes the capsid of two actinobacteriophages 著者: Podgorski JM / Porgorski J / Gosselin S / Abad L / Jacobs-Sera D / Brown C / Hatfull G / Gogarten P / Luque A / White SJ | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
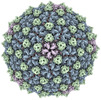
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40077.map.gz emd_40077.map.gz | 1.7 GB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40077-v30.xml emd-40077-v30.xml emd-40077.xml emd-40077.xml | 22.6 KB 22.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_40077.png emd_40077.png | 312.7 KB | ||
| Filedesc metadata |  emd-40077.cif.gz emd-40077.cif.gz | 6.1 KB | ||
| その他 |  emd_40077_additional_1.map.gz emd_40077_additional_1.map.gz emd_40077_additional_2.map.gz emd_40077_additional_2.map.gz emd_40077_half_map_1.map.gz emd_40077_half_map_1.map.gz emd_40077_half_map_2.map.gz emd_40077_half_map_2.map.gz | 1.8 GB 1.6 GB 1.6 GB 1.6 GB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40077 http://ftp.pdbj.org/pub/emdb/structures/EMD-40077 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40077 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40077 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_40077_validation.pdf.gz emd_40077_validation.pdf.gz | 996.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_40077_full_validation.pdf.gz emd_40077_full_validation.pdf.gz | 996 KB | 表示 | |
| XML形式データ |  emd_40077_validation.xml.gz emd_40077_validation.xml.gz | 25.2 KB | 表示 | |
| CIF形式データ |  emd_40077_validation.cif.gz emd_40077_validation.cif.gz | 30.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40077 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40077 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40077 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40077 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8giuMC  8sajC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40077.map.gz / 形式: CCP4 / 大きさ: 1.9 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40077.map.gz / 形式: CCP4 / 大きさ: 1.9 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map of ewald sphere corrected postprocess. | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.2 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Ewald sphere corrected map
| ファイル | emd_40077_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
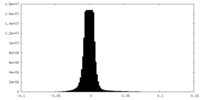
| 注釈 | Ewald sphere corrected map | ||||||||||||
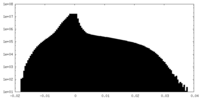
| 投影像・断面図 |
| ||||||||||||
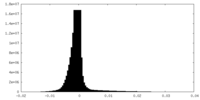
| 密度ヒストグラム |
-追加マップ: Map after CTF Refinement.
| ファイル | emd_40077_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
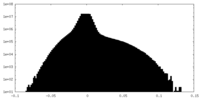
| 注釈 | Map after CTF Refinement. | ||||||||||||
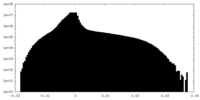
| 投影像・断面図 |
| ||||||||||||
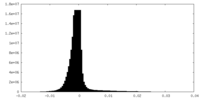
| 密度ヒストグラム |
-ハーフマップ: Half map of ewald sphere corrected Refine3D After CTFRefine.mrc.
| ファイル | emd_40077_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
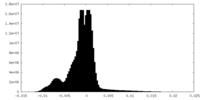
| 注釈 | Half map of ewald sphere corrected Refine3D_After_CTFRefine.mrc. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map of ewald sphere corrected Refine3D After CTFRefine.mrc.
| ファイル | emd_40077_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
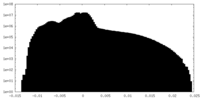
| 注釈 | Half map of ewald sphere corrected Refine3D_After_CTFRefine.mrc. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Mycobacterium phage Patience
| 全体 | 名称:  Mycobacterium phage Patience (ファージ) Mycobacterium phage Patience (ファージ) |
|---|---|
| 要素 |
|
-超分子 #1: Mycobacterium phage Patience
| 超分子 | 名称: Mycobacterium phage Patience / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 1074308 / 生物種: Mycobacterium phage Patience / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) |
| ウイルス殻 | Shell ID: 1 / 直径: 760.0 Å / T番号(三角分割数): 7 |
-分子 #1: gp_4 (capsid accessory protein)
| 分子 | 名称: gp_4 (capsid accessory protein) / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycobacterium phage Patience (ファージ) Mycobacterium phage Patience (ファージ) |
| 分子量 | 理論値: 10.549938 KDa |
| 配列 | 文字列: MANRTVSPST QGVRPAMRQM YNGRNVATRP IPLIVDTSEI RAIMAAAADA RPKTSAVNFP QSGPRPAGAA VVFGTKVSGA PGNVVSNNA ATFAPLTGTQ NFE UniProtKB: Uncharacterized protein |
-分子 #2: Capsid protein
| 分子 | 名称: Capsid protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycobacterium phage Patience (ファージ) Mycobacterium phage Patience (ファージ) |
| 分子量 | 理論値: 43.845391 KDa |
| 配列 | 文字列: MATKELKIGG VPVFPIFGGT APVRQEGIMT QGDLVTVTSD GIDLNALWNS FAESIAIYNE AMDNLIQLLT YPVTVPVEPV VQIGETTFE EATELGVPRG AGLPIEVFQM GYDLRHYDKR NAYSWMFLAD ADGRQVEAIH DAVLWADKRL VFRKVMEALF D NRTRRANI ...文字列: MATKELKIGG VPVFPIFGGT APVRQEGIMT QGDLVTVTSD GIDLNALWNS FAESIAIYNE AMDNLIQLLT YPVTVPVEPV VQIGETTFE EATELGVPRG AGLPIEVFQM GYDLRHYDKR NAYSWMFLAD ADGRQVEAIH DAVLWADKRL VFRKVMEALF D NRTRRANI RNQAYNVYPL YNGDGVPPPR FKNNVFDETH SHYVISHNSV VDSSDLEDLM ELLAEHGYSP QAGTQFLLLA NK AETDAIR QFRRGVVNNN GATAGYDFIP SPTQPAMMLP NAEGLLGNQP APTFGGLAVI GSYGFWNIVE EDYIPPGYLV GVG YGGAFN LGNPVGLRQH ANPAMQGLRI IAGNYQRYPL VDGFYARSFG TGVRQRGGAA IMQIKASGAY ECPPIYKKGG GFLV UniProtKB: Capsid protein |
-分子 #3: gp_22 (Minor Capsid Protein)
| 分子 | 名称: gp_22 (Minor Capsid Protein) / タイプ: protein_or_peptide / ID: 3 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycobacterium phage Patience (ファージ) Mycobacterium phage Patience (ファージ) |
| 分子量 | 理論値: 23.845107 KDa |
| 配列 | 文字列: MALKTKPRWD KYDGYVGNYR GVLGEDIDLD TEANRVLAVG TNSNGAIVVG AGQTGIKGLM IVAVGADIHG AMLDGGINNH AGDPQDVGK HGEITNFQPT VFGRTFGVAI SATEGNVKLA VNGVDTGNIA YDTSAANLKS GIVAVDDGFT ADDFTVTGTA P NFTIVTTR ...文字列: MALKTKPRWD KYDGYVGNYR GVLGEDIDLD TEANRVLAVG TNSNGAIVVG AGQTGIKGLM IVAVGADIHG AMLDGGINNH AGDPQDVGK HGEITNFQPT VFGRTFGVAI SATEGNVKLA VNGVDTGNIA YDTSAANLKS GIVAVDDGFT ADDFTVTGTA P NFTIVTTR TDVTITASGE GVTVTEATSV AAAGTNYYGH ADGTVNAVKG SDGVYVGHTQ EADRLIVNVK DEED UniProtKB: Uncharacterized protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 10 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 検出モード: COUNTING / 実像数: 8640 / 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Amino acid sequence built into the map for a single major capsid protein and refined with Phenix. Model then used for rest of asymmetric unit and refined with Phenix. Final step involved using Isolde. |
|---|---|
| 精密化 | プロトコル: AB INITIO MODEL |
| 得られたモデル |  PDB-8giu: |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X