| 登録情報 | データベース: PDB / ID: 3lgp
|
|---|
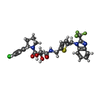
| タイトル | Crystal structure of catalytic domain of tace with benzimidazolyl-thienyl-tartrate based inhibitor |
|---|
 要素 要素 | Disintegrin and metalloproteinase domain-containing protein 17 |
|---|
 キーワード キーワード | HYDROLASE / GLYCOPROTEIN / MEMBRANE / METAL-BINDING / METALLOPROTEASE / NOTCH SIGNALING PATHWAY / PHOSPHOPROTEIN / PROTEASE / TRANSMEMBRANE / ZYMOGEN / Cleavage on pair of basic residues / Disulfide bond / SH3-binding |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
Metallo-peptidase family M12 / Domain of unknown function DUF3850 / Domain of unknown function (DUF3850) / ASCH / ASCH domain / Collagenase (Catalytic Domain) / Collagenase (Catalytic Domain) / PUA-like superfamily / 3-Layer(aba) Sandwich / Alpha Beta類似検索 - ドメイン・相同性 Chem-50X / DUF3850 domain-containing protein類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / pdb entry 1bkc / 解像度: 1.9 Å X線回折 / pdb entry 1bkc / 解像度: 1.9 Å |
|---|
 データ登録者 データ登録者 | Orth, P. |
|---|
 引用 引用 |  ジャーナル: Bioorg.Med.Chem.Lett. / 年: 2010 ジャーナル: Bioorg.Med.Chem.Lett. / 年: 2010
タイトル: Structure and activity relationships of tartrate-based TACE inhibitors.
著者: Li, D. / Popovici-Muller, J. / Belanger, D.B. / Caldwell, J. / Dai, C. / David, M. / Girijavallabhan, V.M. / Lavey, B.J. / Lee, J.F. / Liu, Z. / Mazzola, R. / Rizvi, R. / Rosner, K.E. / ...著者: Li, D. / Popovici-Muller, J. / Belanger, D.B. / Caldwell, J. / Dai, C. / David, M. / Girijavallabhan, V.M. / Lavey, B.J. / Lee, J.F. / Liu, Z. / Mazzola, R. / Rizvi, R. / Rosner, K.E. / Shankar, B. / Spitler, J. / Ting, P.C. / Vaccaro, H. / Yu, W. / Zhou, G. / Zhu, Z. / Niu, X. / Sun, J. / Guo, Z. / Orth, P. / Chen, S. / Kozlowski, J.A. / Lundell, D.J. / Madison, V. / McKittrick, B. / Piwinski, J.J. / Shih, N.Y. / Shipps, G.W. / Siddiqui, M.A. / Strickland, C.O. |
|---|
| 履歴 | | 登録 | 2010年1月21日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2010年7月28日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2021年10月13日 | Group: Database references / Derived calculations
カテゴリ: database_2 / pdbx_struct_conn_angle ...database_2 / pdbx_struct_conn_angle / struct_conn / struct_ref_seq_dif / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_struct_conn_angle.ptnr1_auth_asym_id / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_asym_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr2_auth_asym_id / _pdbx_struct_conn_angle.ptnr2_auth_seq_id / _pdbx_struct_conn_angle.ptnr2_label_asym_id / _pdbx_struct_conn_angle.ptnr3_auth_asym_id / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id / _struct_ref_seq_dif.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
| 改定 1.3 | 2024年11月6日 | Group: Data collection / Structure summary
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / pdbx_entry_details / pdbx_modification_feature |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 / pdb entry 1bkc / 解像度: 1.9 Å
X線回折 / pdb entry 1bkc / 解像度: 1.9 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Bioorg.Med.Chem.Lett. / 年: 2010
ジャーナル: Bioorg.Med.Chem.Lett. / 年: 2010 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3lgp.cif.gz
3lgp.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3lgp.ent.gz
pdb3lgp.ent.gz PDB形式
PDB形式 3lgp.json.gz
3lgp.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/lg/3lgp
https://data.pdbj.org/pub/pdb/validation_reports/lg/3lgp ftp://data.pdbj.org/pub/pdb/validation_reports/lg/3lgp
ftp://data.pdbj.org/pub/pdb/validation_reports/lg/3lgp リンク
リンク 集合体
集合体


 要素
要素 Homo sapiens (ヒト) / 遺伝子: ADAM17, CSVP, TACE / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: ADAM17, CSVP, TACE / 発現宿主:  Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P78536, EC: 3.4.24.86
Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P78536, EC: 3.4.24.86 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 回転陽極 / タイプ: RIGAKU FR-E DW / 波長: 1.54178 Å
回転陽極 / タイプ: RIGAKU FR-E DW / 波長: 1.54178 Å 解析
解析 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj