+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The cryo-EM structure of the Mycobacterium tuberculosis CRISPR-Csm complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Mycobacteria CRISPR-Csm complexes /  RNA BINDING PROTEIN (RNA結合タンパク質) RNA BINDING PROTEIN (RNA結合タンパク質) | |||||||||
| 機能・相同性 | CRISPR-associated protein Csm5 / CRISPR-associated RAMP Csm3 / CRISPR type III-associated protein / RAMP superfamily /  endonuclease activity / defense response to virus / endonuclease activity / defense response to virus /  RNA binding / CRISPR system Cms endoribonuclease Csm3 / CRISPR system Cms protein Csm5 RNA binding / CRISPR system Cms endoribonuclease Csm3 / CRISPR system Cms protein Csm5 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Mycobacterium tuberculosis (結核菌) Mycobacterium tuberculosis (結核菌) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.1 Å クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Liu MX / Li ZK | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 | ジャーナル: FASEB J / 年: 2019 タイトル: Mycobacterium tuberculosis type III-A CRISPR/Cas system crRNA and its maturation have atypical features. 著者: Wenjing Wei / Shuai Zhang / Joy Fleming / Ying Chen / Zihui Li / Shanghua Fan / Yi Liu / Wei Wang / Ting Wang / Ying Liu / Baiguang Ren / Ming Wang / Jianjian Jiao / Yuanyuan Chen / Ying Zhou ...著者: Wenjing Wei / Shuai Zhang / Joy Fleming / Ying Chen / Zihui Li / Shanghua Fan / Yi Liu / Wei Wang / Ting Wang / Ying Liu / Baiguang Ren / Ming Wang / Jianjian Jiao / Yuanyuan Chen / Ying Zhou / Yafeng Zhou / Shoujin Gu / Xiaoli Zhang / Li Wan / Tao Chen / Lin Zhou / Yong Chen / Xian-En Zhang / Chuanyou Li / Hongtai Zhang / Lijun Bi /  要旨: Clustered regularly interspaced short palindromic repeat (CRISPR)/CRISPR-associated protein (Cas) systems are prokaryotic adaptive immune systems against invading nucleic acids. CRISPR locus ...Clustered regularly interspaced short palindromic repeat (CRISPR)/CRISPR-associated protein (Cas) systems are prokaryotic adaptive immune systems against invading nucleic acids. CRISPR locus variability has been exploited in evolutionary and epidemiological studies of Mycobacterium tuberculosis, the causative agent of tuberculosis, for over 20 yr, yet the biological function of this type III-A system is largely unexplored. Here, using cell biology and biochemical, mutagenic, and RNA-seq approaches, we show it is active in invader defense and has features atypical of type III-A systems: mature CRISPR RNA (crRNA) in its crRNA-CRISPR/Cas protein complex are of uniform length (∼71 nt) and appear not to be subject to 3'-end processing after Cas6 cleavage of repeat RNA 8 nt from its 3' end. crRNAs generated resemble mature crRNA in type I systems, having both 5' (8 nt) and 3' (28 nt) repeat tags. Cas6 cleavage of repeat RNA is ion dependent, and accurate cleavage depends on the presence of a 3' hairpin in the repeat RNA and the sequence of its stem base nucleotides. This study unveils further diversity among CRISPR/Cas systems and provides insight into the crRNA recognition mechanism in M. tuberculosis, providing a foundation for investigating the potential of a type III-A-based genome editing system.-Wei, W., Zhang, S., Fleming, J., Chen, Y., Li, Z., Fan, S., Liu, Y., Wang, W., Wang, T., Liu, Y., Ren, B., Wang, M., Jiao, J., Chen, Y., Zhou, Y., Zhou, Y., Gu, S., Zhang, X., Wan, L., Chen, T., Zhou, L., Chen, Y., Zhang, X.-E., Li, C., Zhang, H., Bi, L. Mycobacterium tuberculosis type III-A CRISPR/Cas system crRNA and its maturation have atypical features. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_38066.map.gz emd_38066.map.gz | 450.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-38066-v30.xml emd-38066-v30.xml emd-38066.xml emd-38066.xml | 18 KB 18 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_38066.png emd_38066.png | 14.9 KB | ||
| Filedesc metadata |  emd-38066.cif.gz emd-38066.cif.gz | 5.9 KB | ||
| その他 |  emd_38066_half_map_1.map.gz emd_38066_half_map_1.map.gz emd_38066_half_map_2.map.gz emd_38066_half_map_2.map.gz | 442.4 MB 442.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-38066 http://ftp.pdbj.org/pub/emdb/structures/EMD-38066 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-38066 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-38066 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8x5dMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_38066.map.gz / 形式: CCP4 / 大きさ: 476.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_38066.map.gz / 形式: CCP4 / 大きさ: 476.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.855 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_38066_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
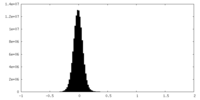
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_38066_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
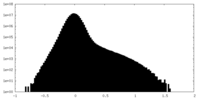
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Mycobacteria CRISPR-Csm complexes
| 全体 | 名称: Mycobacteria CRISPR-Csm complexes |
|---|---|
| 要素 |
|
-超分子 #1: Mycobacteria CRISPR-Csm complexes
| 超分子 | 名称: Mycobacteria CRISPR-Csm complexes / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:   Mycobacterium tuberculosis (結核菌) Mycobacterium tuberculosis (結核菌) |
-分子 #1: CRISPR system Cms endoribonuclease Csm3
| 分子 | 名称: CRISPR system Cms endoribonuclease Csm3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Mycobacterium tuberculosis (結核菌) Mycobacterium tuberculosis (結核菌) |
| 分子量 | 理論値: 26.147736 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MAHMTTSYAK IEITGTLTVL TGLQIGAGDG FSAIGAVDKP VVRDPLSRLP MIPGTSLKGK VRTLLSRQYG ADTETFYRKP NEDHAHIRR LFGDTEEYMT GRLVFRDTKL TNKDDLEARG AKTLTEVKFE NAINRVTAKA NLRQMERVIP GSEFAFSLVY E VSFGTPGE ...文字列: MAHMTTSYAK IEITGTLTVL TGLQIGAGDG FSAIGAVDKP VVRDPLSRLP MIPGTSLKGK VRTLLSRQYG ADTETFYRKP NEDHAHIRR LFGDTEEYMT GRLVFRDTKL TNKDDLEARG AKTLTEVKFE NAINRVTAKA NLRQMERVIP GSEFAFSLVY E VSFGTPGE EQKASLPSSD EIIEDFNAIA RGLKLLELDY LGGSGTRGYG QVKFSNLKAR AAVGALDGSL LEKLNHELAA V UniProtKB: CRISPR system Cms endoribonuclease Csm3 |
-分子 #2: CRISPR system Cms protein Csm5
| 分子 | 名称: CRISPR system Cms protein Csm5 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Mycobacterium tuberculosis (結核菌) Mycobacterium tuberculosis (結核菌) |
| 分子量 | 理論値: 42.752129 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MAHMNTYLKP FELTLRCLGP VFIGSGEKRT SKEYHVEGDR VYFPDMELLY ADIPAHKRKS FEAFVMNTDG AQATAPLKEW VEPNAVKLD PAKHRGYEVK IGSIEPRRAS RGRGGRMTRK KLTLNEIHAF IKDPLGRPYV PGSTVKGMLR SIYLQSLVHK R TAQPVRVP ...文字列: MAHMNTYLKP FELTLRCLGP VFIGSGEKRT SKEYHVEGDR VYFPDMELLY ADIPAHKRKS FEAFVMNTDG AQATAPLKEW VEPNAVKLD PAKHRGYEVK IGSIEPRRAS RGRGGRMTRK KLTLNEIHAF IKDPLGRPYV PGSTVKGMLR SIYLQSLVHK R TAQPVRVP GHQTREHRQY GERFERKELR KSGRPNTRPQ DAVNDLFQAI RVTDSPALRT SDLLICQKMD MNVHGKPDGL PL FRECLAP GTSISHRVVV DTSPTARGGW REGERFLETL AETAASVNQA RYAEYRAMYP GVNAIVGPIV YLGGGAGYRS KTF VTDQDD MAKVLDAQFG KVVKHVDKTR ELRVSPLVLK RTKIDNICYE MGQCELSIRR AE UniProtKB: CRISPR system Cms protein Csm5 |
-分子 #4: Csm2
| 分子 | 名称: Csm2 / タイプ: protein_or_peptide / ID: 4 / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Mycobacterium tuberculosis (結核菌) Mycobacterium tuberculosis (結核菌) |
| 分子量 | 理論値: 15.346579 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MAHMSVIQDD YVKQAEQVIR GLPKKNGDFE LTTTQLRVLL SLTAQLFDEA QLSSDQNLSP ALRDKVQYLR VRFVYQAGRE KAVRVFVER AGLLDELAQI GDSRDRLLKF CHYMEALVAY KKFLDPKETS KETE |
-分子 #3: RNA (47-MER)
| 分子 | 名称: RNA (47-MER) / タイプ: rna / ID: 3 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:   Mycobacterium tuberculosis (結核菌) Mycobacterium tuberculosis (結核菌) |
| 分子量 | 理論値: 59.360578 KDa |
| 配列 | 文字列: GUCGUCAGAC CCAAAACCCC GAGAGGGGAC GGAAACUUAA AACCGUGUUG CACUGCAACC CGGAAUUCUU GCACGUCGUC AGACCCAAA ACCCCGAGAG GGGACGGAAA CUUAAAACCG UGUUGCACUG CAACCCGGAA UUCUUGCACG UCGUCAGACC C AAAACCCC GAGAGGGGAC GGAAAC |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 119066 |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X