+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the IS621 recombinase in complex with bridge RNA, donor DNA, and target DNA in the post-strand exchange state (Holliday junction intermediate) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Holliday junction / RNA dependent recombinase / RECOMBINATION-RNA-DNA COMPLEX | |||||||||
| 機能・相同性 | Transposase, IS111A/IS1328/IS1533, N-terminal / Transposase, IS116/IS110/IS902 / : / Transposase / Transposase IS116/IS110/IS902 family / transposase activity / DNA transposition / DNA binding / IS621 transposase 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
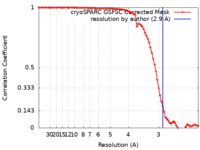
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||
 データ登録者 データ登録者 | Hiraizumi M / Yamashita K / Nishimasu H | |||||||||
| 資金援助 |  日本, 2件 日本, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2024 ジャーナル: Nature / 年: 2024タイトル: Structural mechanism of bridge RNA-guided recombination. 著者: Masahiro Hiraizumi / Nicholas T Perry / Matthew G Durrant / Teppei Soma / Naoto Nagahata / Sae Okazaki / Januka S Athukoralage / Yukari Isayama / James J Pai / April Pawluk / Silvana ...著者: Masahiro Hiraizumi / Nicholas T Perry / Matthew G Durrant / Teppei Soma / Naoto Nagahata / Sae Okazaki / Januka S Athukoralage / Yukari Isayama / James J Pai / April Pawluk / Silvana Konermann / Keitaro Yamashita / Patrick D Hsu / Hiroshi Nishimasu /   要旨: Insertion sequence (IS) elements are the simplest autonomous transposable elements found in prokaryotic genomes. We recently discovered that IS110 family elements encode a recombinase and a non- ...Insertion sequence (IS) elements are the simplest autonomous transposable elements found in prokaryotic genomes. We recently discovered that IS110 family elements encode a recombinase and a non-coding bridge RNA (bRNA) that confers modular specificity for target DNA and donor DNA through two programmable loops. Here we report the cryo-electron microscopy structures of the IS110 recombinase in complex with its bRNA, target DNA and donor DNA in three different stages of the recombination reaction cycle. The IS110 synaptic complex comprises two recombinase dimers, one of which houses the target-binding loop of the bRNA and binds to target DNA, whereas the other coordinates the bRNA donor-binding loop and donor DNA. We uncovered the formation of a composite RuvC-Tnp active site that spans the two dimers, positioning the catalytic serine residues adjacent to the recombination sites in both target and donor DNA. A comparison of the three structures revealed that (1) the top strands of target and donor DNA are cleaved at the composite active sites to form covalent 5'-phosphoserine intermediates, (2) the cleaved DNA strands are exchanged and religated to create a Holliday junction intermediate, and (3) this intermediate is subsequently resolved by cleavage of the bottom strands. Overall, this study reveals the mechanism by which a bispecific RNA confers target and donor DNA specificity to IS110 recombinases for programmable DNA recombination. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_37829.map.gz emd_37829.map.gz | 26.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-37829-v30.xml emd-37829-v30.xml emd-37829.xml emd-37829.xml | 19.7 KB 19.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_37829_fsc.xml emd_37829_fsc.xml | 7.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_37829.png emd_37829.png | 131.2 KB | ||
| マスクデータ |  emd_37829_msk_1.map emd_37829_msk_1.map | 52.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-37829.cif.gz emd-37829.cif.gz | 6.4 KB | ||
| その他 |  emd_37829_half_map_1.map.gz emd_37829_half_map_1.map.gz emd_37829_half_map_2.map.gz emd_37829_half_map_2.map.gz | 49 MB 49 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-37829 http://ftp.pdbj.org/pub/emdb/structures/EMD-37829 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37829 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37829 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_37829_validation.pdf.gz emd_37829_validation.pdf.gz | 861.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_37829_full_validation.pdf.gz emd_37829_full_validation.pdf.gz | 861.3 KB | 表示 | |
| XML形式データ |  emd_37829_validation.xml.gz emd_37829_validation.xml.gz | 15.8 KB | 表示 | |
| CIF形式データ |  emd_37829_validation.cif.gz emd_37829_validation.cif.gz | 20.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37829 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37829 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37829 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37829 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_37829.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_37829.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.10667 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_37829_msk_1.map emd_37829_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
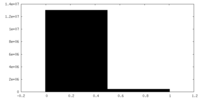
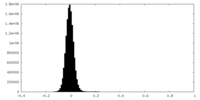
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_37829_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
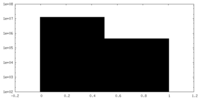
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_37829_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
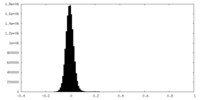
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : The IS621 recombinase in complex with bridge RNA, donor DNA, and ...
| 全体 | 名称: The IS621 recombinase in complex with bridge RNA, donor DNA, and target DNA in the post-strand exchange state (Holliday junction intermediate) |
|---|---|
| 要素 |
|
-超分子 #1: The IS621 recombinase in complex with bridge RNA, donor DNA, and ...
| 超分子 | 名称: The IS621 recombinase in complex with bridge RNA, donor DNA, and target DNA in the post-strand exchange state (Holliday junction intermediate) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#6 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: IS621 transposase
| 分子 | 名称: IS621 transposase / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 36.735355 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GPMEHELHYI GIDTAKEKLD VDVLRPDGRH RTKKFANTTK GHDELVSWLK GHKIDHAHIC IEATGTYMEP VAECLYDAGY IVSVINPAL GKAFAQSEGL RNKTDTVDAR MLAEFCRQKR PAAWEAPHPL ERALRALVVR HQALTDMHTQ ELNRTETARE V QRPSIDAH ...文字列: GPMEHELHYI GIDTAKEKLD VDVLRPDGRH RTKKFANTTK GHDELVSWLK GHKIDHAHIC IEATGTYMEP VAECLYDAGY IVSVINPAL GKAFAQSEGL RNKTDTVDAR MLAEFCRQKR PAAWEAPHPL ERALRALVVR HQALTDMHTQ ELNRTETARE V QRPSIDAH LLWLEAELKR LEKQIKDLTD DDPDMKHRRK LLESIPGIGE KTSAVLLAYI GLKDRFAHAR QFAAFAGLTP RR YESGSSV RGASRMSKAG HVSLRRALYM PAMVATSKTE WGRAFRDRLA ANGKKGKVIL GAMMRKLAQV AYGVLKSGVP FDA SRHNPV AA UniProtKB: IS621 transposase |
-分子 #2: bridge RNA
| 分子 | 名称: bridge RNA / タイプ: rna / ID: 2 / コピー数: 2 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 57.668918 KDa |
| 配列 | 文字列: GGGAGUGCAG AGAAAAUCGG CCAGUUUUCU CUGCCUGCAG UCCGCAUGCC GUAUCGGGCC UUGGGUUCUA ACCUGUUCUG UAGAUUUAU GCAGCGGACU GCCUUUCUCC CAAAGUGAUA AACCGGACAG UAUCAUGGAC CGGUUUUCCC GGUAAUCCGU A UUUACAAG GCUGGUUUCA CU |
-分子 #3: target DNA-donor DNA
| 分子 | 名称: target DNA-donor DNA / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 15.003622 KDa |
| 配列 | 文字列: (DG)(DC)(DC)(DG)(DG)(DG)(DT)(DA)(DA)(DT) (DA)(DC)(DC)(DA)(DC)(DC)(DA)(DA)(DG)(DC) (DC)(DG)(DC)(DC)(DT)(DT)(DG)(DT)(DA) (DT)(DT)(DA)(DT)(DC)(DC)(DC)(DT)(DC)(DC) (DA) (DG)(DT)(DG)(DC)(DA)(DG)(DA)(DG) (DA) |
-分子 #4: target DNA
| 分子 | 名称: target DNA / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 11.703487 KDa |
| 配列 | 文字列: (DC)(DG)(DA)(DG)(DC)(DT)(DC)(DA)(DT)(DC) (DT)(DG)(DT)(DA)(DG)(DG)(DC)(DC)(DC)(DG) (DA)(DT)(DG)(DG)(DT)(DG)(DG)(DT)(DA) (DT)(DT)(DA)(DC)(DC)(DC)(DG)(DG)(DC) |
-分子 #5: donor DNA-target DNA
| 分子 | 名称: donor DNA-target DNA / タイプ: dna / ID: 5 詳細: The linkage between 20I and 21I has been cleaved by IS621. コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 10.163561 KDa |
| 配列 | 文字列: (DT)(DG)(DC)(DA)(DG)(DG)(DC)(DC)(DA)(DT) (DA)(DA)(DG)(DT)(DC)(DA)(DA)(DT)(DC)(DT) (DA)(DC)(DA)(DG)(DA)(DT)(DG)(DA)(DG) (DC)(DT)(DC)(DG) |
-分子 #6: donor DNA
| 分子 | 名称: donor DNA / タイプ: dna / ID: 6 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 13.587737 KDa |
| 配列 | 文字列: (DT)(DC)(DT)(DC)(DT)(DG)(DC)(DA)(DC)(DT) (DG)(DG)(DA)(DG)(DG)(DG)(DA)(DT)(DA)(DA) (DT)(DA)(DC)(DA)(DA)(DG)(DA)(DT)(DA) (DC)(DT)(DG)(DT)(DT)(DA)(DT)(DG)(DG)(DC) (DC) (DT)(DG)(DC)(DA) |
-分子 #7: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 7 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 62.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z
Z Y
Y X
X