| 登録情報 | データベース: EMDB / ID: EMD-37726
|
|---|
| タイトル | Cryo-EM structure of SARS-CoV-2 receptor-binding domain (RBD) complexed with CB6 mutant,S309, and S304 antibodies |
|---|
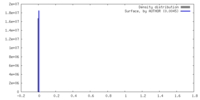
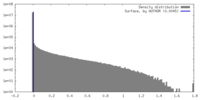
 マップデータ マップデータ | |
|---|
 試料 試料 | - 複合体: Cryo-EM structure of SARS-CoV-2 receptor-binding domain (RBD) complexed with CB6 mutant,S309, and S304 antibodies
- 複合体: fab
- タンパク質・ペプチド: S309 fab heavy chain
- タンパク質・ペプチド: S309 fab light chain
- タンパク質・ペプチド: CB6 fab mutant heavy chain
- タンパク質・ペプチド: CB6 fab mutant light chain
- タンパク質・ペプチド: S304 fab heavy chain
- タンパク質・ペプチド: S304 fab light chain
- 複合体: SARS-CoV-2 RBD
- タンパク質・ペプチド: Spike protein S1
|
|---|
 キーワード キーワード | SARS-CoV-2 / RBD / CB6 / receptor-binding domain / etesevimab / mutant / VIRAL PROTEIN / IMMUNE SYSTEM-VIRAL PROTEIN complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
symbiont-mediated disruption of host tissue / Maturation of spike protein / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / viral translation / symbiont-mediated-mediated suppression of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion ...symbiont-mediated disruption of host tissue / Maturation of spike protein / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / viral translation / symbiont-mediated-mediated suppression of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / membrane fusion / entry receptor-mediated virion attachment to host cell / Attachment and Entry / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / host cell surface receptor binding / symbiont-mediated suppression of host innate immune response / receptor ligand activity / endocytosis involved in viral entry into host cell / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / symbiont entry into host cell / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane / identical protein binding / membrane / plasma membrane類似検索 - 分子機能 Spike (S) protein S1 subunit, receptor-binding domain, SARS-CoV-2 / Spike (S) protein S1 subunit, N-terminal domain, SARS-CoV-like / Coronavirus spike glycoprotein S1, C-terminal / Coronavirus spike glycoprotein S1, C-terminal / Spike glycoprotein, N-terminal domain superfamily / Spike S1 subunit, receptor binding domain superfamily, betacoronavirus / Spike glycoprotein, betacoronavirus / Betacoronavirus spike (S) glycoprotein S1 subunit N-terminal (NTD) domain profile. / Betacoronavirus spike (S) glycoprotein S1 subunit C-terminal (CTD) domain profile. / Spike (S) protein S1 subunit, receptor-binding domain, betacoronavirus ...Spike (S) protein S1 subunit, receptor-binding domain, SARS-CoV-2 / Spike (S) protein S1 subunit, N-terminal domain, SARS-CoV-like / Coronavirus spike glycoprotein S1, C-terminal / Coronavirus spike glycoprotein S1, C-terminal / Spike glycoprotein, N-terminal domain superfamily / Spike S1 subunit, receptor binding domain superfamily, betacoronavirus / Spike glycoprotein, betacoronavirus / Betacoronavirus spike (S) glycoprotein S1 subunit N-terminal (NTD) domain profile. / Betacoronavirus spike (S) glycoprotein S1 subunit C-terminal (CTD) domain profile. / Spike (S) protein S1 subunit, receptor-binding domain, betacoronavirus / Betacoronavirus spike glycoprotein S1, receptor binding / Spike glycoprotein S1, N-terminal domain, betacoronavirus-like / Betacoronavirus-like spike glycoprotein S1, N-terminal / Spike glycoprotein S2 superfamily, coronavirus / Spike glycoprotein S2, coronavirus, heptad repeat 1 / Spike glycoprotein S2, coronavirus, heptad repeat 2 / Coronavirus spike (S) glycoprotein S2 subunit heptad repeat 1 (HR1) region profile. / Coronavirus spike (S) glycoprotein S2 subunit heptad repeat 2 (HR2) region profile. / Spike glycoprotein S2, coronavirus / Coronavirus spike glycoprotein S2類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /   Severe acute respiratory syndrome-related coronavirus (ウイルス) / Severe acute respiratory syndrome-related coronavirus (ウイルス) /   Severe acute respiratory syndrome coronavirus 2 (ウイルス) Severe acute respiratory syndrome coronavirus 2 (ウイルス) |
|---|
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.02 Å |
|---|
 データ登録者 データ登録者 | Su C / Qi JX / Gao GF |
|---|
| 資金援助 | 1件 | Organization | Grant number | 国 |
|---|
| Not funded | | |
|
|---|
 引用 引用 |  ジャーナル: Hlife / 年: 2025 ジャーナル: Hlife / 年: 2025
タイトル: Enabling the immune escaped etesevimab fully-armed against SARS-CoV-2 Omicron subvariants including KP.2
著者: Su C / He J / Xie Y / Hu Y / Li X / Qiao S / Liu P / Huang M / Zhang R / Wang L / Chang Z / Sun W / Xu K / Zhang J / Cao L / Han P / Zhao X / Qi J / Wang Q / Yang M / Gao GF |
|---|
| 履歴 | | 登録 | 2023年10月10日 | - |
|---|
| ヘッダ(付随情報) 公開 | 2024年10月16日 | - |
|---|
| マップ公開 | 2024年10月16日 | - |
|---|
| 更新 | 2025年10月29日 | - |
|---|
| 現状 | 2025年10月29日 | 処理サイト: PDBj / 状態: 公開 |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト) /
Homo sapiens (ヒト) / 

 データ登録者
データ登録者 引用
引用 ジャーナル: Hlife / 年: 2025
ジャーナル: Hlife / 年: 2025 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_37726.map.gz
emd_37726.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-37726-v30.xml
emd-37726-v30.xml emd-37726.xml
emd-37726.xml EMDBヘッダ
EMDBヘッダ emd_37726.png
emd_37726.png emd-37726.cif.gz
emd-37726.cif.gz emd_37726_half_map_1.map.gz
emd_37726_half_map_1.map.gz emd_37726_half_map_2.map.gz
emd_37726_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-37726
http://ftp.pdbj.org/pub/emdb/structures/EMD-37726 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37726
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37726

 F&H 検索
F&H 検索 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_37726.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_37726.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)