+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 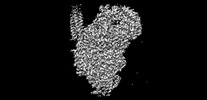 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the human parainfluenza virus hPIV3 L-P polymerase in dimeric form | |||||||||
 マップデータ マップデータ | Cryo-EM map of the human parainfluenza virus hPIV3 L-P polymerase in dimeric form, class 1 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | dimeric polymerase / parainfluenza virus / L-P polymerase / cryo-EM structure / non-segmented negative-strand RNA virus / L-L dimerization / RNA replication / RdRp active site / VIRAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報GDP polyribonucleotidyltransferase / 加水分解酵素; 酸無水物に作用; リン含有酸無水物に作用 / viral genome replication / 転移酵素; 一炭素原子の基を移すもの; メチル基を移すもの / virion component / mRNA 5'-cap (guanine-N7-)-methyltransferase activity / host cell cytoplasm / RNA-directed RNA polymerase / RNA-dependent RNA polymerase activity / GTPase activity ...GDP polyribonucleotidyltransferase / 加水分解酵素; 酸無水物に作用; リン含有酸無水物に作用 / viral genome replication / 転移酵素; 一炭素原子の基を移すもの; メチル基を移すもの / virion component / mRNA 5'-cap (guanine-N7-)-methyltransferase activity / host cell cytoplasm / RNA-directed RNA polymerase / RNA-dependent RNA polymerase activity / GTPase activity / DNA-templated transcription / RNA binding / ATP binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Human respirovirus 3 (ウイルス) Human respirovirus 3 (ウイルス) | |||||||||
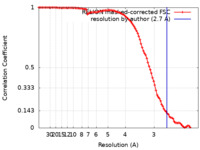
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||
 データ登録者 データ登録者 | Xie J / Wang L / Zhai G / Wu D / Lin Z / Wang M / Yan X / Gao L / Huang X / Fearns R / Chen S | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Structural basis for dimerization of a paramyxovirus polymerase complex. 著者: Jin Xie / Mohamed Ouizougun-Oubari / Li Wang / Guanglei Zhai / Daitze Wu / Zhaohu Lin / Manfu Wang / Barbara Ludeke / Xiaodong Yan / Tobias Nilsson / Lu Gao / Xinyi Huang / Rachel Fearns / Shuai Chen /    要旨: The transcription and replication processes of non-segmented, negative-strand RNA viruses (nsNSVs) are catalyzed by a multi-functional polymerase complex composed of the large protein (L) and a ...The transcription and replication processes of non-segmented, negative-strand RNA viruses (nsNSVs) are catalyzed by a multi-functional polymerase complex composed of the large protein (L) and a cofactor protein, such as phosphoprotein (P). Previous studies have shown that the nsNSV polymerase can adopt a dimeric form, however, the structure of the dimer and its function are poorly understood. Here we determine a 2.7 Å cryo-EM structure of human parainfluenza virus type 3 (hPIV3) L-P complex with the connector domain (CD') of a second L built, while reconstruction of the rest of the second L-P obtains a low-resolution map of the ring-like L core region. This study reveals detailed atomic features of nsNSV polymerase active site and distinct conformation of hPIV3 L with a unique β-strand latch. Furthermore, we report the structural basis of L-L dimerization, with CD' located at the putative template entry of the adjoining L. Disruption of the L-L interface causes a defect in RNA replication that can be overcome by complementation, demonstrating that L dimerization is necessary for hPIV3 genome replication. These findings provide further insight into how nsNSV polymerases perform their functions, and suggest a new avenue for rational drug design. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_37130.map.gz emd_37130.map.gz | 42.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-37130-v30.xml emd-37130-v30.xml emd-37130.xml emd-37130.xml | 19.5 KB 19.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_37130_fsc.xml emd_37130_fsc.xml | 9.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_37130.png emd_37130.png | 43.4 KB | ||
| マスクデータ |  emd_37130_msk_1.map emd_37130_msk_1.map | 178 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-37130.cif.gz emd-37130.cif.gz | 7.5 KB | ||
| その他 |  emd_37130_half_map_1.map.gz emd_37130_half_map_1.map.gz emd_37130_half_map_2.map.gz emd_37130_half_map_2.map.gz | 77.3 MB 77.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-37130 http://ftp.pdbj.org/pub/emdb/structures/EMD-37130 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37130 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37130 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8kdbMC  8kdcC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_37130.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_37130.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM map of the human parainfluenza virus hPIV3 L-P polymerase in dimeric form, class 1 | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.087 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_37130_msk_1.map emd_37130_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half1 map
| ファイル | emd_37130_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half1 map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half2 map
| ファイル | emd_37130_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half2 map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human parainfluenza virus hPIV3 L-P polymerase in dimeric form
| 全体 | 名称: Human parainfluenza virus hPIV3 L-P polymerase in dimeric form |
|---|---|
| 要素 |
|
-超分子 #1: Human parainfluenza virus hPIV3 L-P polymerase in dimeric form
| 超分子 | 名称: Human parainfluenza virus hPIV3 L-P polymerase in dimeric form タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Human respirovirus 3 (ウイルス) Human respirovirus 3 (ウイルス) |
-分子 #1: RNA-directed RNA polymerase L
| 分子 | 名称: RNA-directed RNA polymerase L / タイプ: protein_or_peptide / ID: 1 / 詳細: NP_067153.2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human respirovirus 3 (ウイルス) Human respirovirus 3 (ウイルス) |
| 分子量 | 理論値: 260.103703 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MISNQQSDNG QKENIKNLGA KRARKMDTES NNGTVSDILY PECHLNSPIV KGKIAQLHTI MSLPQPYDMD DDSILVITRQ KIKLNKLDK RQRSIRRLKL ILTEKVNDLG KYTFIRYPEM SKEMFKLYIP GINSKVTELL LKADRTYSQM TDGLRDLWIN V LSKLASKN ...文字列: MISNQQSDNG QKENIKNLGA KRARKMDTES NNGTVSDILY PECHLNSPIV KGKIAQLHTI MSLPQPYDMD DDSILVITRQ KIKLNKLDK RQRSIRRLKL ILTEKVNDLG KYTFIRYPEM SKEMFKLYIP GINSKVTELL LKADRTYSQM TDGLRDLWIN V LSKLASKN DGSNYDLNEE INNISKVHTT YKSDKWYNPF KTWFTIKYDM RRLQKARNEI TFNVGKDYNL LEDQKNFLLI HP ELVLILD KQNYNGYLIT PELVLMYCDV VEGRWNISAC AKLDPKLQSM YQKGNNLWEV IDKLFPIMGE KTFDVISLLE PLA LSLIQT HDPVKQLRGA FLNHVLSEME LIFESGESIR EFLSVDYIDK ILDIFNESTI DEIAEIFSFF RTFGHPPLEA SIAA EKVRK YMYIEKQLKF DTVNKCHAIF CTIIINGYRE RHGGQWPPVT LPDHAHEFII NAYGSNSAIS YENAVDYYQS FIGIK FNKF IEPQLDEDLT IYMKDKALSP KKSNWDTVYP ASNLLYRTNA SNESRRLVEV FIADSKFDPH QILDYVESGD WLDDPE FNI SYSLKEKEIK QEGRLFAKMT YKMRATQVLS ETLLANNIGK FFQENGMVKG EIELLKRLTT ISISGVPRYN EVYNNSK SH TDDLKTYNKI SNLNLSSNQK SKKFEFKSTD IYNDGYETVS CFLTTDLKKY CLNWRYESTA LFGETCNQIF GLNKLFNW L HPRLEGSTIY VGDPYCPPSD KEHISLEDHP DSGFYVHNPR GGIEGFCQKL WTLISISAIH LAAVRIGVRV TAMVQGDNQ AIAVTTRVPN NYDYRIKKEI VYKDVVRFFD SLREVMDDLG HELKLNETII SSKMFIYSKR IYYDGRILPQ ALKALSRCVF WSETVIDET RSASSNLATS FAKAIENGYS PVLGYACSIF KNIQQLYIAL GMNINPTITQ NIKDQYFKNS NWMQYASLIP A SVGGFNYM AMSRCFVRNI GDPSVAALAD IKRFIKANLL DRSVLYRIMN QEPGESSFLD WASDPYSCNL PQSQNITTMI KN ITARNVL QDSPNPLLSG LFTNTMIEED EELAEFLMDR KVILPRVAHD ILDNSLTGIR NAIAGMLDTT KSLIRVGINR GGL TYSLLR KISNYDLVQY ETLSRTLRLI VSDKIRYEDM CSVDLAIALR QKMWIHLSGG RMISGLETPD PLELLSGVVI TGSE HCKIC YSSDGTNPYT WMYLPGNIKI GSAETGVSSL RVPYFGSVTD ERSEAQLGYI KNLSKPAKAA IRIAMIYTWA FGNDE ISWM EASQIAQTRA NFTLDSLKIL TPVATSTNLS HRLKDTATQM KFSSTSLIRV SRFITMSNDN MSIKEANETK DTNLIY QQI MLTGLSVFEY LFRLKETTGH NPIVMHLHIE DECCIKESFN DEHINPESTL ELIRYPESNE FIYDKDPLKD VDLSKLM VI KDHSYTIDMN YWDDTDIIHA ISICTAITIA DTMSQLDRDN LKEIIVIAND DDINSLITEF LTLDILVFLK TFGGLLVN Q FAYTLYSLKI EGRDLIWDYI MRTLRDTSHS ILKVLSNALS HPKVFKRFWD CGVLNPIYGP NTASQDQIKL ALSICEYAL DLFMREWLNG VSLEIYICDS DMEVANDRKQ AFISRHLSFV CCLAEIASFG PNLLNLTYLE RLDLLKQYLE LNIKEDPTLK YVQISGLLI KSFPSTVTYV RKTAIKYLRI RGISPPEVID DWDPIEDENM LDNIVKTIND NCNKDNKGNK INNFWGLALK N YQVLKIRS ITSDSDDNDR LDASTSGLTL PQGGNYLSHQ LRLFGINSTS CLKALELSQI LMKEVNKDKD RLFLGEGAGA ML ACYDATL GPAINYYNSG LNITDVIGQR ELKIFPSEVS LVGKKLGNVT QILNRVKVLF NGNPNSTWIG NMECESLIWS ELN DKSIGL VHCDMEGAIG KSEETVLHEH YSVIRITYLI GDDDVVLVSK IIPTITPNWS RILYLYKLYW KDVSIISLKT SNPA STELY LISKDAYCTI MEPSEVVLSK LKRLSLLEEN NLLKWIILSK KRNNEWLHHE IKEGERDYGV MRPYHMALQI FGFQI NLNH LAKEFLSTPD LTNINNIIQS FQRTIKDVLF EWINITHDDK RHKLGGRYNI FPLKNKGKLR LLSRRLVLSW ISLSLS TRL LTGRFPDEKF EHRAQTGYVS LADTDLESLK LLSKNIIKNY RECIGSISYW FLTKEVKILM KLIGGAKLLG IPRQYKE PE EQLLENYNQH DEFDIDDYKD DDDK UniProtKB: RNA-directed RNA polymerase L |
-分子 #2: Phosphoprotein
| 分子 | 名称: Phosphoprotein / タイプ: protein_or_peptide / ID: 2 / 詳細: NP_067149.1 / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human respirovirus 3 (ウイルス) Human respirovirus 3 (ウイルス) |
| 分子量 | 理論値: 68.471312 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MESDAKNYQI MDSWEEESRD KSTNISSALN IIEFILSTDP QEDLSENDTI NTRTQQLSAT IYQPKIKPTE TSEKDSGSTD KNRQSGSSH ECTTEAKDRT IDQETVQRGP GRRSSSDSRA ETVVSGGISR SITNSKNGTQ NTEDIDLNEI RKMDKDSIEG K VRQSADVP ...文字列: MESDAKNYQI MDSWEEESRD KSTNISSALN IIEFILSTDP QEDLSENDTI NTRTQQLSAT IYQPKIKPTE TSEKDSGSTD KNRQSGSSH ECTTEAKDRT IDQETVQRGP GRRSSSDSRA ETVVSGGISR SITNSKNGTQ NTEDIDLNEI RKMDKDSIEG K VRQSADVP SEISGSDVIF TTEQSRNSDH GRSLESISTP DTRSISVVTA ATPDDEEEIL MKNSRTKKSS SIHQEDDKRI KK GGKGKDW FKKSKDTDNQ IPTSDYRSTS KGQKKISKTT TINTDTKGQT EIQTESSGTQ SSSWNLTIDN NTDRTEQTNT TPP TTTSGS TYTKESIRTN SGSKPKTQKT NGKERKDTEE SNRFTERAIT LLQNLGVIQS TSKLDLYQDK RVVCVANVLN NVDT ASKID FLAGLVIGVS MDNDTKLTQI QNEMLNLKAD LKKMDESHRR LIENQREQLS LITSLISNLK IMTERGGKKD QNESN ERVS MIKTKLKEEK IKKTRFDPLM ETQGIDKNIP DLYRHAGNTL ENDVQVKSEI LSSYNESNAT RLIPKKVSST MRSLVA VIS NSNLSQSTKQ SYINELKHCK NDEEVSELMD MFNEDVNNCQ HHHHHH UniProtKB: Phosphoprotein |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #4: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 4 / コピー数: 2 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X