+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human SIDT1 bound to cholesterol | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Membrane protein | ||||||||||||
| 機能・相同性 | RNA transmembrane transporter activity / SID1 transmembrane family / dsRNA-gated channel SID-1 / RNA transport / cholesterol binding / double-stranded RNA binding / lysosome / plasma membrane / SID1 transmembrane family member 1 機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.77 Å | ||||||||||||
 データ登録者 データ登録者 | Hirano Y / Ohto U / Shimizu T | ||||||||||||
| 資金援助 |  日本, 3件 日本, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2024 ジャーナル: Commun Biol / 年: 2024タイトル: Cryo-EM analysis reveals human SID-1 transmembrane family member 1 dynamics underlying lipid hydrolytic activity. 著者: Yoshinori Hirano / Umeharu Ohto / Ikuyo Ichi / Ryota Sato / Kensuke Miyake / Toshiyuki Shimizu /  要旨: Two mammalian homologs of systemic RNA interference defective protein 1 (SID-1) (SIDT1/2) are suggested to function as double-stranded RNA (dsRNA) transporters for extracellular dsRNA uptake or for ...Two mammalian homologs of systemic RNA interference defective protein 1 (SID-1) (SIDT1/2) are suggested to function as double-stranded RNA (dsRNA) transporters for extracellular dsRNA uptake or for release of incorporated dsRNA from lysosome to cytoplasm. SIDT1/2 is also suggested to be involved in cholesterol transport and lipid metabolism. Here, we determine the cryo-electron microscopy structures of human SIDT1, homodimer in a side-by-side arrangement, with two distinct conformations, the cholesterol-bound form and the unbound form. Our structures reveal that the membrane-spanning region of SIDT1 harbors conserved histidine and aspartate residues coordinating to putative zinc ion, in a structurally similar manner to alkaline ceramidases or adiponectin receptors that require zinc for ceramidase activity. We identify that SIDT1 has a ceramidase activity that is attenuated by cholesterol binding. Observations from two structures suggest that cholesterol molecules serve as allosteric regulator that binds the transmembrane region of SIDT1 and induces the conformation change and the reorientation of the catalytic residues. This study represents a contribution to the elucidation of the cholesterol-mediated mechanisms of lipid hydrolytic activity and RNA transport in the SID-1 family proteins. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
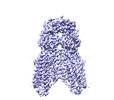
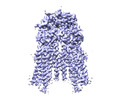
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_37112.map.gz emd_37112.map.gz | 22.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-37112-v30.xml emd-37112-v30.xml emd-37112.xml emd-37112.xml | 14.7 KB 14.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_37112.png emd_37112.png | 98.2 KB | ||
| Filedesc metadata |  emd-37112.cif.gz emd-37112.cif.gz | 6 KB | ||
| その他 |  emd_37112_half_map_1.map.gz emd_37112_half_map_1.map.gz emd_37112_half_map_2.map.gz emd_37112_half_map_2.map.gz | 22.5 MB 22.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-37112 http://ftp.pdbj.org/pub/emdb/structures/EMD-37112 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37112 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37112 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_37112_validation.pdf.gz emd_37112_validation.pdf.gz | 817.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_37112_full_validation.pdf.gz emd_37112_full_validation.pdf.gz | 817.2 KB | 表示 | |
| XML形式データ |  emd_37112_validation.xml.gz emd_37112_validation.xml.gz | 10.5 KB | 表示 | |
| CIF形式データ |  emd_37112_validation.cif.gz emd_37112_validation.cif.gz | 12.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37112 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37112 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37112 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37112 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8kcwMC  8kcxC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_37112.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_37112.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.245 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_37112_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
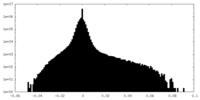
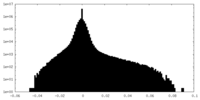
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_37112_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
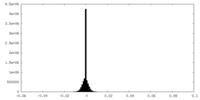
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : homo-dimer of SIDT1
| 全体 | 名称: homo-dimer of SIDT1 |
|---|---|
| 要素 |
|
-超分子 #1: homo-dimer of SIDT1
| 超分子 | 名称: homo-dimer of SIDT1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: SID1 transmembrane family member 1
| 分子 | 名称: SID1 transmembrane family member 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 96.877312 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MRGCLRLALL CALPWLLLAA SPGHPAKSPR QPPAPRRDPF DAARGADFDH VYSGVVNLST ENIYSFNYTS QPDQVTAVRV YVNSSSENL NYPVLVVVRQ QKEVLSWQVP LLFQGLYQRS YNYQEVSRTL CPSEATNETG PLQQLIFVDV ASMAPLGAQY K LLVTKLKH ...文字列: MRGCLRLALL CALPWLLLAA SPGHPAKSPR QPPAPRRDPF DAARGADFDH VYSGVVNLST ENIYSFNYTS QPDQVTAVRV YVNSSSENL NYPVLVVVRQ QKEVLSWQVP LLFQGLYQRS YNYQEVSRTL CPSEATNETG PLQQLIFVDV ASMAPLGAQY K LLVTKLKH FQLRTNVAFH FTASPSQPQY FLYKFPKDVD SVIIKVVSEM AYPCSVVSVQ NIMCPVYDLD HNVEFNGVYQ SM TKKAAIT LQKKDFPGEQ FFVVFVIKPE DYACGGSFFI QEKENQTWNL QRKKNLEVTI VPSIKESVYV KSSLFSVFIF LSF YLGCLL VGFVHYLRFQ RKSIDGSFGS NDGSGNMVAS HPIAASTPEG SNYGTIDESS SSPGRQMSSS DGGPPGQSDT DSSV EESDF DTMPDIESDK NIIRTKMFLY LSDLSRKDRR IVSKKYKIYF WNIITIAVFY ALPVIQLVIT YQTVVNVTGN QDICY YNFL CAHPLGVLSA FNNILSNLGH VLLGFLFLLI VLRRDILHRR ALEAKDIFAV EYGIPKHFGL FYAMGIALMM EGVLSA CYH VCPNYSNFQF DTSFMYMIAG LCMLKLYQTR HPDINASAYS AYASFAVVIM VTVLGVVFGK NDVWFWVIFS AIHVLAS LA LSTQIYYMGR FKIDLGIFRR AAMVFYTDCI QQCSRPLYMD RMVLLVVGNL VNWSFALFGL IYRPRDFASY MLGIFICN L LLYLAFYIIM KLRSSEKVLP VPLFCIVATA VMWAAALYFF FQNLSSWEGT PAESREKNRE CILLDFFDDH DIWHFLSAT ALFFSFLVLL TLDDDLDVVR RDQIPVFENL YFQGDYKDDD DKHHHHHHHH UniProtKB: SID1 transmembrane family member 1 |
-分子 #4: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 4 / コピー数: 2 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-分子 #5: CHOLESTEROL
| 分子 | 名称: CHOLESTEROL / タイプ: ligand / ID: 5 / コピー数: 4 / 式: CLR |
|---|---|
| 分子量 | 理論値: 386.654 Da |
| Chemical component information |  ChemComp-CLR: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL |
|---|---|
| 緩衝液 | pH: 6 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 65.7 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.77 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 59745 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー



 Z
Z Y
Y X
X