+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The cryo-EM map of close TIEA-TEC complex | |||||||||
 マップデータ マップデータ | The cryo-EM map of close TIEA-TIC complex | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | bacteriophage T4 / RNA polymerase / transcription / inhibitor / elongation accelerator / complex / TRANSCRIPTION/DNA/RNA / TRANSCRIPTION-DNA-RNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報RNA polymerase complex / submerged biofilm formation / cellular response to cell envelope stress / bacterial-type RNA polymerase core enzyme binding / cytosolic DNA-directed RNA polymerase complex / regulation of DNA-templated transcription initiation / bacterial-type flagellum assembly / bacterial-type flagellum-dependent cell motility / nitrate assimilation / transcription elongation factor complex ...RNA polymerase complex / submerged biofilm formation / cellular response to cell envelope stress / bacterial-type RNA polymerase core enzyme binding / cytosolic DNA-directed RNA polymerase complex / regulation of DNA-templated transcription initiation / bacterial-type flagellum assembly / bacterial-type flagellum-dependent cell motility / nitrate assimilation / transcription elongation factor complex / regulation of DNA-templated transcription elongation / DNA-templated transcription initiation / transcription antitermination / cell motility / ribonucleoside binding / DNA-directed 5'-3' RNA polymerase activity / DNA-directed RNA polymerase / response to heat / protein-containing complex assembly / intracellular iron ion homeostasis / DNA replication / protein dimerization activity / response to antibiotic / magnesium ion binding / DNA binding / zinc ion binding / membrane / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |   Escherichia phage T4 (ファージ) Escherichia phage T4 (ファージ) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.15 Å | |||||||||
 データ登録者 データ登録者 | Zhang KN / Liu Y / Chen M / Wang Y / Lin W / Li M / Zhang X / Gao Y / Gong Q / Chen H ...Zhang KN / Liu Y / Chen M / Wang Y / Lin W / Li M / Zhang X / Gao Y / Gong Q / Chen H / Steve M / Li S / Zhang K / Liu B | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: TIEA inhibits Sigma70-dependent transcriptions, accelerates elongation speed and elevates transcription error 著者: Zhang KN / Liu Y / Chen M / Wang Y / Lin W / Li M / Zhang X / Gao Y / Gong Q / Chen H / Steve M / Li S / Zhang K / Liu B | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_36897.map.gz emd_36897.map.gz | 136.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-36897-v30.xml emd-36897-v30.xml emd-36897.xml emd-36897.xml | 26.8 KB 26.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_36897.png emd_36897.png | 78.3 KB | ||
| Filedesc metadata |  emd-36897.cif.gz emd-36897.cif.gz | 8.4 KB | ||
| その他 |  emd_36897_additional_1.map.gz emd_36897_additional_1.map.gz emd_36897_half_map_1.map.gz emd_36897_half_map_1.map.gz emd_36897_half_map_2.map.gz emd_36897_half_map_2.map.gz | 72.2 MB 134.5 MB 134.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-36897 http://ftp.pdbj.org/pub/emdb/structures/EMD-36897 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36897 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36897 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_36897_validation.pdf.gz emd_36897_validation.pdf.gz | 1002.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_36897_full_validation.pdf.gz emd_36897_full_validation.pdf.gz | 1002.4 KB | 表示 | |
| XML形式データ |  emd_36897_validation.xml.gz emd_36897_validation.xml.gz | 14.3 KB | 表示 | |
| CIF形式データ |  emd_36897_validation.cif.gz emd_36897_validation.cif.gz | 17 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36897 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36897 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36897 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36897 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8k58MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_36897.map.gz / 形式: CCP4 / 大きさ: 144.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_36897.map.gz / 形式: CCP4 / 大きさ: 144.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The cryo-EM map of close TIEA-TIC complex | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||||||||||||||||||
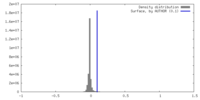
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: The cryo-EM map of close TIEA-TIC complex
| ファイル | emd_36897_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The cryo-EM map of close TIEA-TIC complex | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
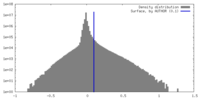
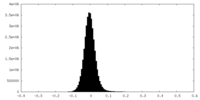
| 密度ヒストグラム |
-ハーフマップ: The cryo-EM map of close TIEA-TIC complex
| ファイル | emd_36897_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The cryo-EM map of close TIEA-TIC complex | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
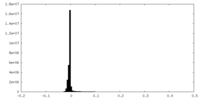
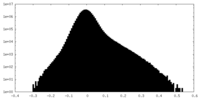
| 密度ヒストグラム |
-ハーフマップ: The cryo-EM map of close TIEA-TIC complex
| ファイル | emd_36897_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The cryo-EM map of close TIEA-TIC complex | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
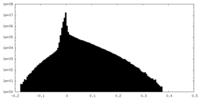
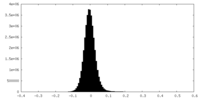
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : The cryo-EM map of close TIEA-TEC complex
| 全体 | 名称: The cryo-EM map of close TIEA-TEC complex |
|---|---|
| 要素 |
|
-超分子 #1: The cryo-EM map of close TIEA-TEC complex
| 超分子 | 名称: The cryo-EM map of close TIEA-TEC complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 400 KDa |
-分子 #1: DNA-directed RNA polymerase subunit beta
| 分子 | 名称: DNA-directed RNA polymerase subunit beta / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: DNA-directed RNA polymerase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 150.590547 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: YSYTEKKRIR KDFGKRPQVL DVPYLLSIQL DSFQKFIEQD PEGQYGLEAA FRSVFPIQSY SGNSELQYVS YRLGEPVFDV QECQIRGVT YSAPLRVKLR LVIYEREAPE GTVKDIKEQE VYMGEIPLMT DNGTFVINGT ERVIVSQLHR SPGVFFDSDK G KTHSSGKV ...文字列: YSYTEKKRIR KDFGKRPQVL DVPYLLSIQL DSFQKFIEQD PEGQYGLEAA FRSVFPIQSY SGNSELQYVS YRLGEPVFDV QECQIRGVT YSAPLRVKLR LVIYEREAPE GTVKDIKEQE VYMGEIPLMT DNGTFVINGT ERVIVSQLHR SPGVFFDSDK G KTHSSGKV LYNARIIPYR GSWLDFEFDP KDNLFVRIDR RRKLPATIIL RALNYTTEQI LDLFFEKVIF EIRDNKLQME LV PERLRGE TASFDIEANG KVYVEKGRRI TARHIRQLEK DDVKLIEVPV EYIAGKVVAK DYIDESTGEL ICAANMELSL DLL AKLSQS GHKRIETLFT NDLDHGPYIS ETLRVDPTND RLSALVEIYR MMRPGEPPTR EAAESLFENL FFSEDRYDLS AVGR MKFNR SLLREEIEGS GILSKDDIID VMKKLIDIRN GKGEVDDIDH LGNRRIRSVG EMAENQFRVG LVRVERAVKE RLSLG DLDT LMPQDMINAK PISAAVKEFF GSSQLSQFMD QNNPLSEITH KRRISALGPG GLTRERAGFE VRDVHPTHYG RVCPIE TPE GPNIGLINSL SVYAQTNEYG FLETPYRKVT DGVVTDEIHY LSAIEEGNYV IAQANSNLDE EGHFVEDLVT CRSKGES SL FSRDQVDYMD VSTQQVVSVG ASLIPFLEHD DANRALMGAN MQRQAVPTLR ADKPLVGTGM ERAVAVDSGV TAVAKRGG V VQYVDASRIV IKVNEDEMYP GEAGIDIYNL TKYTRSNQNT CINQMPCVSL GEPVERGDVL ADGPSTDLGE LALGQNMRV AFMPWNGYNF EDSILVSERV VQEDRFTTIH IQELACVSRD TKLGPEEITA DIPNVGEAAL SKLDESGIVY IGAEVTGGDI LVGKVTPKG ETQLTPEEKL LRAIFGEKAS DVKDSSLRVP NGVSGTVIDV QVFTRDGVEK DKRALEIEEM QLKQAKKDLS E ELQILEAG LFSRIRAVLV AGGVEAEKLD KLPRDRWLEL GLTDEEKQNQ LEQLAEQYDE LKHEFEKKLE AKRRKITQGD DL APGVLKI VKVYLAVKRR IQPGDKMAGR HGNKGVISKI NPIEDMPYDE NGTPVDIVLN PLGVPSRMNI GQILETHLGM AAK GIGDKI NAMLKQQQEV AKLREFIQRA YDLGADVRQK VDLSTFSDEE VMRLAENLRK GMPIATPVFD GAKEAEIKEL LKLG DLPTS GQIRLYDGRT GEQFERPVTV GYMYMLKLNH LVDDKMHARS TGSYSLVTQQ PLGGKAQFGG QRFGEMEVWA LEAYG AAYT LQEMLTVKSD DVNGRTKMYK NIVDGNHQME PGMPESFNVL LKEIRSLGIN IELEDE UniProtKB: DNA-directed RNA polymerase subunit beta |
-分子 #2: DNA-directed RNA polymerase subunit beta'
| 分子 | 名称: DNA-directed RNA polymerase subunit beta' / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号: DNA-directed RNA polymerase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 150.865766 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: TEEFDAIKIA LASPDMIRSW SFGEVKKPET INYRTFKPER DGLFCARIFG PVKDYECLCG KYKRLKHRGV ICEKCGVEVT QTKVRRERM GHIELASPTA HIWFLKSLPS RIGLLLDMPL RDIERVLYFE SYVVIEGGMT NLERQQILTE EQYLDALEEF G DEFDAKMG ...文字列: TEEFDAIKIA LASPDMIRSW SFGEVKKPET INYRTFKPER DGLFCARIFG PVKDYECLCG KYKRLKHRGV ICEKCGVEVT QTKVRRERM GHIELASPTA HIWFLKSLPS RIGLLLDMPL RDIERVLYFE SYVVIEGGMT NLERQQILTE EQYLDALEEF G DEFDAKMG AEAIQALLKS MDLEQECEQL REELNETNSE TKRKKLTKRI KLLEAFVQSG NKPEWMILTV LPVLPPDLRP LV PLDGGRF ATSDLNDLYR RVINRNNRLK RLLDLAAPDI IVRNEKRMLQ EAVDALLDNG RRGRAITGSN KRPLKSLADM IKG KQGRFR QNLLGKRVDY SGRSVITVGP YLRLHQCGLP KKMALELFKP FIYGKLELRG LATTIKAAKK MVEREEAVVW DILD EVIRE HPVLLNRAPT LHRLGIQAFE PVLIEGKAIQ LHPLVCAAYN ADFDGDQMAV HVPLTLEAQL EARALMMSTN NILSP ANGE PIIVPSQDVV LGLYYMTRDC VNAKGEGMVL TGPKEAERLY RSGLASLHAR VKVRITEYEK DANGELVAKT SLKDTT VGR AILWMIVPKG LPYSIVNQAL GKKAISKMLN TCYRILGLKP TVIFADQIMY TGFAYAARSG ASVGIDDMVI PEKKHEI IS EAEAEVAEIQ EQFQSGLVTA GERYNKVIDI WAAANDRVSK AMMDNLQTET VINRDGQEEK QVSFNSIYMM ADSGARGS A AQIRQLAGMR GLMAKPDGSI IETPITANFR EGLNVLQYFI STHGARKGLA DTALKTANSG YLTRRLVDVA QDLVVTEDD CGTHEGIMMT PVIEGGDVKE PLRDRVLGRV TAEDVLKPGT ADILVPRNTL LHEQWCDLLE ENSVDAVKVR SVVSCDTDFG VCAHCYGRD LARGHIINKG EAIGVIAAQS IGEPGTQLTM RTFHIGGAAS RAAAESSIQV KNKGSIKLSN VKSVVNSSGK L VITSRNTE LKLIDEFGRT KESYKVPYGA VLAKGDGEQV AGGETVANWD PHTMPVITEV SGFVRFTDMI DGQTITRQTD EL TGLSSLV VLDSAERTAG GKDLRPALKI VDAQGNDVLI PGTDMPAQYF LPGKAIVQLE DGVQISSGDT LARIPQESGG TKD ITGGLP RVADLFEARR PKEPAILAEI SGIVSFGKET KGKRRLVITP VDGSDPYEEM IPKWRQLNVF EGERVERGDV ISDG PEAPH DILRLRGVHA VTRYIVNEVQ DVYRLQGVKI NDKHIEVIVR QMLRKATIVN AGSSDFLEGE QVEYSRVKIA NRELE ANGK VGATYSRDLL GITKASLATE SFISAASFQE TTRVLTEAAV AGKRDELRGL KENVIVGRLI PAGTGYAYHQ DRMRRR AAG UniProtKB: DNA-directed RNA polymerase subunit beta' |
-分子 #3: DNA-directed RNA polymerase subunit omega
| 分子 | 名称: DNA-directed RNA polymerase subunit omega / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO / EC番号: DNA-directed RNA polymerase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 8.649695 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: ARVTVQDAVE KIGNRFDLVL VAARRARQMQ VGGKDPLVPE ENDKTTVIAL REIEEGLINN QILDVRERQE QQEQEA UniProtKB: DNA-directed RNA polymerase subunit omega |
-分子 #4: DNA-directed RNA polymerase subunit alpha
| 分子 | 名称: DNA-directed RNA polymerase subunit alpha / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO / EC番号: DNA-directed RNA polymerase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 25.612111 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: TEFLKPRLVD IEQVSSTHAK VTLEPLERGF GHTLGNALRR ILLSSMPGCA VTEVEIDGVL HEYSTKEGVQ EDILEILLNL KGLAVRVQG KDEVILTLNK SGIGPVTAAD ITHDGDVEIV KPQHVICHLT DENASISMRI KVQRGRGYVP ASTRIHSEED E RPIGRLLV ...文字列: TEFLKPRLVD IEQVSSTHAK VTLEPLERGF GHTLGNALRR ILLSSMPGCA VTEVEIDGVL HEYSTKEGVQ EDILEILLNL KGLAVRVQG KDEVILTLNK SGIGPVTAAD ITHDGDVEIV KPQHVICHLT DENASISMRI KVQRGRGYVP ASTRIHSEED E RPIGRLLV DACYSPVERI AYNVEAARVE QRTDLDKLVI EMETNGTIDP EEAIRRAATI LAEQLEAFVD LRD UniProtKB: DNA-directed RNA polymerase subunit alpha |
-分子 #5: 15 kDa RNA polymerase-binding protein
| 分子 | 名称: 15 kDa RNA polymerase-binding protein / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage T4 (ファージ) Escherichia phage T4 (ファージ) |
| 分子量 | 理論値: 14.731656 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTKITVNYTV DVKDIQPKHV RSESNPQNQN KIRRAWVLSL SDNAMEVIQN KIKSAPARHA YYEAIDREVS NKWIELMRKH TTESLNAGA KFIMTSCGER LEDDYCGNAD ERLIVAAQIV AETIAADFNR UniProtKB: 15 kDa RNA polymerase-binding protein |
-分子 #6: DNA (29-MER)
| 分子 | 名称: DNA (29-MER) / タイプ: dna / ID: 6 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 8.915744 KDa |
| 配列 | 文字列: (DG)(DG)(DG)(DC)(DT)(DA)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DA)(DT)(DG)(DA)(DC)(DG)(DG) (DC)(DG)(DA)(DA)(DT)(DA)(DC)(DC)(DC) |
-分子 #7: DNA (29-MER)
| 分子 | 名称: DNA (29-MER) / タイプ: dna / ID: 7 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 8.813646 KDa |
| 配列 | 文字列: (DG)(DG)(DG)(DT)(DA)(DT)(DT)(DC)(DG)(DC) (DC)(DG)(DT)(DG)(DT)(DA)(DC)(DC)(DT)(DC) (DT)(DC)(DC)(DT)(DA)(DG)(DC)(DC)(DC) |
-分子 #8: RNA (5'-R(*CP*GP*GP*AP*GP*AP*GP*GP*UP*A)-3')
| 分子 | 名称: RNA (5'-R(*CP*GP*GP*AP*GP*AP*GP*GP*UP*A)-3') / タイプ: rna / ID: 8 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 3.280036 KDa |
| 配列 | 文字列: CGGAGAGGUA |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / デジタル化 - サイズ - 横: 4092 pixel / デジタル化 - サイズ - 縦: 5760 pixel / 実像数: 8645 / 平均露光時間: 3.0 sec. / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 105000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)