+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the panda P2X7 receptor in complex with PPADS | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Channel / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報purinergic nucleotide receptor activity / extracellularly ATP-gated monoatomic cation channel activity / response to ATP / calcium ion transmembrane transport / postsynapse / ATP binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
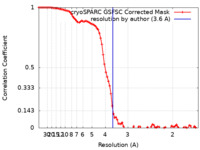
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||
 データ登録者 データ登録者 | Sheng D / Hattori M | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2024 ジャーナル: Elife / 年: 2024タイトル: Structural insights into the orthosteric inhibition of P2X receptors by non-ATP analog antagonists. 著者: Danqi Sheng / Chen-Xi Yue / Fei Jin / Yao Wang / Muneyoshi Ichikawa / Ye Yu / Chang-Run Guo / Motoyuki Hattori /  要旨: P2X receptors are extracellular ATP-gated ion channels that form homo- or heterotrimers and consist of seven subtypes. They are expressed in various tissues, including neuronal and nonneuronal cells, ...P2X receptors are extracellular ATP-gated ion channels that form homo- or heterotrimers and consist of seven subtypes. They are expressed in various tissues, including neuronal and nonneuronal cells, and play critical roles in physiological processes such as neurotransmission, inflammation, pain, and cancer. As a result, P2X receptors have attracted considerable interest as drug targets, and various competitive inhibitors have been developed. However, although several P2X receptor structures from different subtypes have been reported, the limited structural information of P2X receptors in complex with competitive antagonists hampers the understanding of orthosteric inhibition, hindering the further design and optimization of those antagonists for drug discovery. We determined the cryogenic electron microscopy (cryo-EM) structures of the mammalian P2X7 receptor in complex with two classical competitive antagonists of pyridoxal-5'-phosphate derivatives, pyridoxal-5'-phosphate-6-(2'-naphthylazo-6'-nitro-4',8'-disulfonate) (PPNDS) and pyridoxal phosphate-6-azophenyl-2',5'-disulfonic acid (PPADS), and performed structure-based mutational analysis by patch-clamp recording as well as molecular dynamics (MD) simulations. Our structures revealed the orthosteric site for PPADS/PPNDS, and structural comparison with the previously reported apo- and ATP-bound structures showed how PPADS/PPNDS binding inhibits the conformational changes associated with channel activation. In addition, structure-based mutational analysis identified key residues involved in the PPNDS sensitivity of P2X1 and P2X3, which are known to have higher affinity for PPADS/PPNDS than other P2X subtypes. #1:  ジャーナル: Elife / 年: 2023 ジャーナル: Elife / 年: 2023タイトル: Structural insights into the orthosteric inhibition of P2X receptors by non-ATP-analog antagonists 著者: Sheng D / Yue C / Jin F / Wang Y / Ichikawa M / Yu Y / Guo CR / Hattori M | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_36670.map.gz emd_36670.map.gz | 32.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-36670-v30.xml emd-36670-v30.xml emd-36670.xml emd-36670.xml | 15.2 KB 15.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_36670_fsc.xml emd_36670_fsc.xml | 9.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_36670.png emd_36670.png | 26.7 KB | ||
| Filedesc metadata |  emd-36670.cif.gz emd-36670.cif.gz | 5.6 KB | ||
| その他 |  emd_36670_half_map_1.map.gz emd_36670_half_map_1.map.gz emd_36670_half_map_2.map.gz emd_36670_half_map_2.map.gz | 59.5 MB 59.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-36670 http://ftp.pdbj.org/pub/emdb/structures/EMD-36670 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36670 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36670 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_36670_validation.pdf.gz emd_36670_validation.pdf.gz | 831.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_36670_full_validation.pdf.gz emd_36670_full_validation.pdf.gz | 830.9 KB | 表示 | |
| XML形式データ |  emd_36670_validation.xml.gz emd_36670_validation.xml.gz | 16.4 KB | 表示 | |
| CIF形式データ |  emd_36670_validation.cif.gz emd_36670_validation.cif.gz | 21 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36670 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36670 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36670 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36670 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8jv7MC  8jv8C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_36670.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_36670.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_36670_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_36670_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : P2X7 trimer
| 全体 | 名称: P2X7 trimer |
|---|---|
| 要素 |
|
-超分子 #1: P2X7 trimer
| 超分子 | 名称: P2X7 trimer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: P2X purinoceptor
| 分子 | 名称: P2X purinoceptor / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 38.716234 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSSTNYGTIK WIFHALVFSY ISFALISDKR YQKKEPLISS VHTKVKGIAE VKAEILENGM KKMVSGVFDT ADYTFPLQGN SFFVMTNFI KTEGQQQGLC PDFPTARTIC SSDRGCKKGR MDPQSKGIQT GRCVVYKERL KTCEVSAWCP IEEVKDAPRP A LLNSAENF ...文字列: GSSTNYGTIK WIFHALVFSY ISFALISDKR YQKKEPLISS VHTKVKGIAE VKAEILENGM KKMVSGVFDT ADYTFPLQGN SFFVMTNFI KTEGQQQGLC PDFPTARTIC SSDRGCKKGR MDPQSKGIQT GRCVVYKERL KTCEVSAWCP IEEVKDAPRP A LLNSAENF TVLIKNNIDF PGHNYTTRNI LPGVNITCTF HKTQNPQCPI FRLGDIFQET GDSFSDVAIQ GGIMGIEIYW DC NLDGWFH HCRPKYSFRR LDDKTTSESL YPGYNFRYAK YYKENNVEKR TLIKVFGIRF DILVFGTGGK FNVIQLAVYI GSV ISYFGL ATVFIDILIN TYSASS UniProtKB: P2X purinoceptor |
-分子 #2: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 2 / コピー数: 6 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #3: 4-[(E)-[4-methanoyl-6-methyl-5-oxidanyl-3-(phosphonooxymethyl)pyr...
| 分子 | 名称: 4-[(E)-[4-methanoyl-6-methyl-5-oxidanyl-3-(phosphonooxymethyl)pyridin-2-yl]diazenyl]benzene-1,3-disulfonic acid タイプ: ligand / ID: 3 / コピー数: 3 / 式: UO6 |
|---|---|
| 分子量 | 理論値: 511.378 Da |
| Chemical component information | 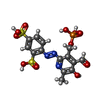 ChemComp-UO6: |
-分子 #4: water
| 分子 | 名称: water / タイプ: ligand / ID: 4 / コピー数: 3 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.3 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X