+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Mi3 fused with FKBP | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | protein cage Scaffold / LYASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報macrolide binding / activin receptor binding / cytoplasmic side of membrane / signaling receptor inhibitor activity / transforming growth factor beta receptor binding / TGFBR1 LBD Mutants in Cancer / type I transforming growth factor beta receptor binding / negative regulation of activin receptor signaling pathway / heart trabecula formation / terminal cisterna ...macrolide binding / activin receptor binding / cytoplasmic side of membrane / signaling receptor inhibitor activity / transforming growth factor beta receptor binding / TGFBR1 LBD Mutants in Cancer / type I transforming growth factor beta receptor binding / negative regulation of activin receptor signaling pathway / heart trabecula formation / terminal cisterna / ryanodine receptor complex / I-SMAD binding / regulation of amyloid precursor protein catabolic process / protein maturation by protein folding / ventricular cardiac muscle tissue morphogenesis / 'de novo' protein folding / negative regulation of phosphoprotein phosphatase activity / FK506 binding / mTORC1-mediated signalling / TGF-beta receptor signaling activates SMADs / Calcineurin activates NFAT / regulation of immune response / protein peptidyl-prolyl isomerization / supramolecular fiber organization / heart morphogenesis / regulation of ryanodine-sensitive calcium-release channel activity / sarcoplasmic reticulum membrane / T cell activation / TGF-beta receptor signaling in EMT (epithelial to mesenchymal transition) / peptidylprolyl isomerase / sarcoplasmic reticulum / peptidyl-prolyl cis-trans isomerase activity / calcium ion transmembrane transport / negative regulation of transforming growth factor beta receptor signaling pathway / Z disc / SARS-CoV-1 activates/modulates innate immune responses / regulation of protein localization / protein folding / positive regulation of protein binding / protein refolding / positive regulation of canonical NF-kappaB signal transduction / transmembrane transporter binding / amyloid fibril formation / Potential therapeutics for SARS / lyase activity / membrane / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |   Thermotoga maritima (バクテリア) / Thermotoga maritima (バクテリア) /   Thermotoga maritima (strain ATCC 43589 / DSM 3109 / JCM 10099 / NBRC 100826 / MSB8) (バクテリア) Thermotoga maritima (strain ATCC 43589 / DSM 3109 / JCM 10099 / NBRC 100826 / MSB8) (バクテリア) | |||||||||
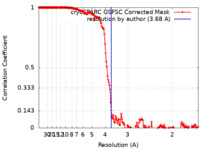
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.68 Å | |||||||||
 データ登録者 データ登録者 | Zhang HW / Kang W / Xue C | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Am Chem Soc / 年: 2024 ジャーナル: J Am Chem Soc / 年: 2024タイトル: Dynamic Metabolons Using Stimuli-Responsive Protein Cages. 著者: Wei Kang / Xiao Ma / Huawei Zhang / Juncai Ma / Chunxue Liu / Jiani Li / Hanhan Guo / Daping Wang / Rui Wang / Bo Li / Chuang Xue /   要旨: Naturally evolved metabolons have the ability to assemble and disassemble in response to environmental stimuli, allowing for the rapid reorganization of chemical reactions in living cells to meet ...Naturally evolved metabolons have the ability to assemble and disassemble in response to environmental stimuli, allowing for the rapid reorganization of chemical reactions in living cells to meet changing cellular needs. However, replicating such capability in synthetic metabolons remains a challenge due to our limited understanding of the mechanisms by which the assembly and disassembly of such naturally occurring multienzyme complexes are controlled. Here, we report the synthesis of chemical- and light-responsive protein cages for assembling synthetic metabolons, enabling the dynamic regulation of enzymatic reactions in living cells. Particularly, a chemically responsive domain was fused to a self-assembled protein cage subunit, generating engineered protein cages capable of displaying proteins containing cognate interaction domains on their surfaces in response to small molecular cues. Chemical-induced colocalization of sequential enzymes on protein cages enhances the specificity of the branched deoxyviolacein biosynthetic reactions by 2.6-fold. Further, by replacing the chemical-inducible domain with a light-inducible dimerization domain, we created an optogenetic protein cage capable of reversibly recruiting and releasing targeted proteins onto and from the exterior of the protein cages in tens of seconds by on-off of blue light. Tethering the optogenetic protein cages to membranes enables the formation of light-switchable, membrane-bound metabolons, which can repeatably recruit-release enzymes, leading to the manipulation of substrate utilization across membranes on demand. Our work demonstrates a powerful and versatile strategy for constructing dynamic metabolons in engineered living cells for efficient and controllable biocatalysis. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_36228.map.gz emd_36228.map.gz | 483.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-36228-v30.xml emd-36228-v30.xml emd-36228.xml emd-36228.xml | 13.6 KB 13.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_36228_fsc.xml emd_36228_fsc.xml | 17 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_36228.png emd_36228.png | 244.5 KB | ||
| Filedesc metadata |  emd-36228.cif.gz emd-36228.cif.gz | 5.3 KB | ||
| その他 |  emd_36228_half_map_1.map.gz emd_36228_half_map_1.map.gz emd_36228_half_map_2.map.gz emd_36228_half_map_2.map.gz | 474.7 MB 474.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-36228 http://ftp.pdbj.org/pub/emdb/structures/EMD-36228 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36228 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36228 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_36228_validation.pdf.gz emd_36228_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_36228_full_validation.pdf.gz emd_36228_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_36228_validation.xml.gz emd_36228_validation.xml.gz | 25.8 KB | 表示 | |
| CIF形式データ |  emd_36228_validation.cif.gz emd_36228_validation.cif.gz | 33.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36228 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36228 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36228 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36228 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8jgaMC  8jgcC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_36228.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_36228.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_36228_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_36228_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : FKBP-Mi3
| 全体 | 名称: FKBP-Mi3 |
|---|---|
| 要素 |
|
-超分子 #1: FKBP-Mi3
| 超分子 | 名称: FKBP-Mi3 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Thermotoga maritima (バクテリア) Thermotoga maritima (バクテリア) |
-分子 #1: Peptidyl-prolyl cis-trans isomerase FKBP1A,2-dehydro-3-deoxyphosp...
| 分子 | 名称: Peptidyl-prolyl cis-trans isomerase FKBP1A,2-dehydro-3-deoxyphosphogluconate aldolase/4-hydroxy-2-oxoglutarate aldolase タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: peptidylprolyl isomerase |
|---|---|
| 由来(天然) | 生物種:   Thermotoga maritima (strain ATCC 43589 / DSM 3109 / JCM 10099 / NBRC 100826 / MSB8) (バクテリア) Thermotoga maritima (strain ATCC 43589 / DSM 3109 / JCM 10099 / NBRC 100826 / MSB8) (バクテリア) |
| 分子量 | 理論値: 36.730309 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH GGSGVQVETI SPGDGRTFPK RGQTCVVHYT GMLEDGKKFD SSRDRNKPFK FMLGKQEVIR GWEEGVAQMS VGQRAKLTI SPDYAYGATG HPGIIPPHAT LVFDVELLKL EGGSGGSGGS GGSMKMEELF KKHKIVAVLR ANSVEEAKKK A LAVFLGGV ...文字列: MGSSHHHHHH GGSGVQVETI SPGDGRTFPK RGQTCVVHYT GMLEDGKKFD SSRDRNKPFK FMLGKQEVIR GWEEGVAQMS VGQRAKLTI SPDYAYGATG HPGIIPPHAT LVFDVELLKL EGGSGGSGGS GGSMKMEELF KKHKIVAVLR ANSVEEAKKK A LAVFLGGV HLIEITFTVP DADTVIKELS FLKEMGAIIG AGTVTSVEQA RKAVESGAEF IVSPHLDEEI SQFAKEKGVF YM PGVMTPT ELVKAMKLGH TILKLFPGEV VGPQFVKAMK GPFPNVKFVP TGGVNLDNVC EWFKAGVLAV GVGSALVKGT PVE VAEKAK AFVEKIRGCT EGSGEPEA UniProtKB: Peptidyl-prolyl cis-trans isomerase FKBP1A, 2-dehydro-3-deoxyphosphogluconate aldolase/4-hydroxy-2-oxoglutarate aldolase |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X