+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 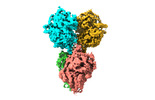 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The cryo-EM structure of the TwOSC1 tetramer | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  oxidosqualene cyclase / Tripterygium wilfordii Hook. f. / TwOSC1 / friedelin / cryo-EM structure / oxidosqualene cyclase / Tripterygium wilfordii Hook. f. / TwOSC1 / friedelin / cryo-EM structure /  PLANT PROTEIN (植物) PLANT PROTEIN (植物) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 oxidosqualene cyclase activity / triterpenoid biosynthetic process / oxidosqualene cyclase activity / triterpenoid biosynthetic process /  異性化酵素; 分子内転位酵素; ムターゼ; その他の基を移すもの / 異性化酵素; 分子内転位酵素; ムターゼ; その他の基を移すもの /  lipid droplet lipid droplet類似検索 - 分子機能 | |||||||||
| 生物種 |  Tripterygium wilfordii (植物) Tripterygium wilfordii (植物) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.75 Å クライオ電子顕微鏡法 / 解像度: 4.75 Å | |||||||||
 データ登録者 データ登録者 | Ma X / Yuru T / Yunfeng L / Jiang T | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Angew Chem Int Ed Engl / 年: 2023 ジャーナル: Angew Chem Int Ed Engl / 年: 2023タイトル: Structural and Catalytic Insight into the Unique Pentacyclic Triterpene Synthase TwOSC. 著者: Yunfeng Luo / Xiaoli Ma / Yufan Qiu / Yun Lu / Siyu Shen / Yang Li / Haiyun Gao / Kang Chen / Jiawei Zhou / Tianyuan Hu / Lichan Tu / Huan Zhao / Dan Li / Faqiang Leng / Wei Gao / Tao Jiang / ...著者: Yunfeng Luo / Xiaoli Ma / Yufan Qiu / Yun Lu / Siyu Shen / Yang Li / Haiyun Gao / Kang Chen / Jiawei Zhou / Tianyuan Hu / Lichan Tu / Huan Zhao / Dan Li / Faqiang Leng / Wei Gao / Tao Jiang / Changli Liu / Luqi Huang / Ruibo Wu / Yuru Tong /  要旨: The oxidosqualene cyclase (OSC) catalyzed cyclization of the linear substrate (3S)-2,3-oxidosqualene to form diverse pentacyclic triterpenoid (PT) skeletons is one of the most complex reactions in ...The oxidosqualene cyclase (OSC) catalyzed cyclization of the linear substrate (3S)-2,3-oxidosqualene to form diverse pentacyclic triterpenoid (PT) skeletons is one of the most complex reactions in nature. Friedelin has a unique PT skeleton involving a fascinating nine-step cation shuttle run (CSR) cascade rearrangement reaction, in which the carbocation formed at C2 moves to the other side of the skeleton, runs back to C3 to yield a friedelin cation, which is finally deprotonated. However, as crystal structure data of plant OSCs are lacking, it remains unknown why the CSR cascade reactions occur in friedelin biosynthesis, as does the exact catalytic mechanism of the CSR. In this study, we determined the first cryogenic electron microscopy structure of a plant OSC, friedelin synthase, from Tripterygium wilfordii Hook. f (TwOSC). We also performed quantum mechanics/molecular mechanics simulations to reveal the energy profile for the CSR cascade reaction and identify key residues crucial for PT skeleton formation. Furthermore, we semirationally designed two TwOSC mutants, which significantly improved the yields of friedelin and β-amyrin, respectively. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
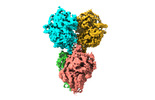
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_35996.map.gz emd_35996.map.gz | 49.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-35996-v30.xml emd-35996-v30.xml emd-35996.xml emd-35996.xml | 14.8 KB 14.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_35996.png emd_35996.png | 52.9 KB | ||
| Filedesc metadata |  emd-35996.cif.gz emd-35996.cif.gz | 5.7 KB | ||
| その他 |  emd_35996_half_map_1.map.gz emd_35996_half_map_1.map.gz emd_35996_half_map_2.map.gz emd_35996_half_map_2.map.gz | 39.7 MB 39.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-35996 http://ftp.pdbj.org/pub/emdb/structures/EMD-35996 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35996 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35996 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8j5zMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_35996.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_35996.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_35996_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
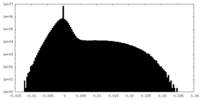
| 投影像・断面図 |
| ||||||||||||
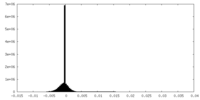
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_35996_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
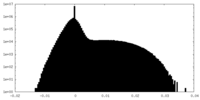
| 投影像・断面図 |
| ||||||||||||
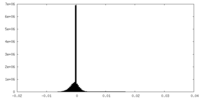
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Tetramer of TwOSC1
| 全体 | 名称: Tetramer of TwOSC1 |
|---|---|
| 要素 |
|
-超分子 #1: Tetramer of TwOSC1
| 超分子 | 名称: Tetramer of TwOSC1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Tripterygium wilfordii (植物) Tripterygium wilfordii (植物) |
-分子 #1: Terpene cyclase/mutase family member
| 分子 | 名称: Terpene cyclase/mutase family member / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Tripterygium wilfordii (植物) Tripterygium wilfordii (植物) |
| 分子量 | 理論値: 88.2125 KDa |
| 組換発現 | 生物種:   Komagataella pastoris (菌類) Komagataella pastoris (菌類) |
| 配列 | 文字列: MWKLKVAERG NAPYSEYLYT TNDFSGRQTW EFDPNAGTPQ ELAKVEEARR KFTEDRHTVK PASDLLWMMQ FMREKNFKQT IPPVRLGEE EQVTYEDLTT ALTRTTNFFT ALQASDGHWP AENGGVSFFL PPFIFSLYIT GHLNSIITPE YRKEILRFIY N HQNEDGGW ...文字列: MWKLKVAERG NAPYSEYLYT TNDFSGRQTW EFDPNAGTPQ ELAKVEEARR KFTEDRHTVK PASDLLWMMQ FMREKNFKQT IPPVRLGEE EQVTYEDLTT ALTRTTNFFT ALQASDGHWP AENGGVSFFL PPFIFSLYIT GHLNSIITPE YRKEILRFIY N HQNEDGGW GIHIEGHSTM FGTAFSYVCL RILGIEVDGG KDNACARARK WILDHGGITY MPSWGKTWLS ILGVYDWYGC NP MPPEFWL LPSYLPIHPA KIWCYCRMVY MPMSYLYGKR FVGPITPLIL QLREELHTQP FHEIQWRQTR HRCAKEDLYY PHS LIQDFI WDSLYVASEP LLTRWPLNKI REKALAKAME HIHYEDENSR YITIGCVEKA LCMLCCWVED PNSDYFKKHL ARIP DYLWV AEDGMKVQSF GSQLWDATFG FQALVASNLT EDEVGPALAK AYDFIKKSQV KDNPSGDFES MHRHISKGSW TFSDQ DHGW QLSDCTAEAL KCCLLAATMP QEVVGEKMKP EWVYEAINII LSLQSKSGGL AGWEPVRAGE WMEILNPMEF LENIVI EHT YVECTGSSII AFVSLKKLYP GHRTKDIDNF IRNAIRYLED VQYPDGSWYG NWGICFIYST MFALGGLAAT GRTYDNC QA VRRGVDFILK NQSDDGGWGE SYLSCPRKVY TPLDGRRSNV VQTAWAMLGL LYAGQAERDP TPLHRGAKVL INYQMEDG G YPQQEITGVF KMNCMLHYPI YRNAFPIWAL GEYRKRVPLP SKGY UniProtKB: Terpene cyclase/mutase family member |
-分子 #2: octyl beta-D-glucopyranoside
| 分子 | 名称: octyl beta-D-glucopyranoside / タイプ: ligand / ID: 2 / コピー数: 4 / 式: BOG |
|---|---|
| 分子量 | 理論値: 292.369 Da |
| Chemical component information |  ChemComp-BOG: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 詳細: 50 mM Tris-HCl (pH 8.0), 300 mM NaCl, 2 mM dithiothreitol, and 0.2% OG |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy最大 デフォーカス(公称値): 2.3000000000000003 µm 最小 デフォーカス(公称値): 1.5 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 4.75 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 32698 |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X