+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the CnCas12f1-sgRNA-DNA complex | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード |  Complex / Complex /  CRISPR-Cas (CRISPR) / RNA BINDING PROTEIN/RNA/DNA / RNA BINDING PROTEIN-RNA-DNA complex CRISPR-Cas (CRISPR) / RNA BINDING PROTEIN/RNA/DNA / RNA BINDING PROTEIN-RNA-DNA complex | ||||||||||||
| 機能・相同性 | Transposase IS605, OrfB, C-terminal / Putative transposase DNA-binding domain / transposition / DNA recombination /  DNA binding / DNA binding /  Transposase Transposase 機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |   Clostridium novyi (ノビイ菌) / synthetic construct (人工物) Clostridium novyi (ノビイ菌) / synthetic construct (人工物) | ||||||||||||
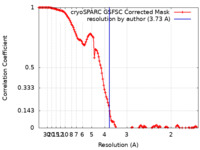
| 手法 |  単粒子再構成法 / 解像度: 3.73 Å 単粒子再構成法 / 解像度: 3.73 Å | ||||||||||||
 データ登録者 データ登録者 | Li F / Ji Q | ||||||||||||
| 資金援助 |  中国, 3件 中国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Structure and engineering of miniature Clostridium novyi CRISPR-Cas12f1 with rare C-rich PAM specificity 著者: Li F / Ji Q | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34958.map.gz emd_34958.map.gz | 203.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34958-v30.xml emd-34958-v30.xml emd-34958.xml emd-34958.xml | 15.4 KB 15.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_34958_fsc.xml emd_34958_fsc.xml | 14.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_34958.png emd_34958.png | 111.3 KB | ||
| その他 |  emd_34958_half_map_1.map.gz emd_34958_half_map_1.map.gz emd_34958_half_map_2.map.gz emd_34958_half_map_2.map.gz | 200.7 MB 200.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34958 http://ftp.pdbj.org/pub/emdb/structures/EMD-34958 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34958 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34958 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8hr5MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34958.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34958.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_34958_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_34958_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ternary complex of CnCas12f1-sgRNA-DNA
| 全体 | 名称: Ternary complex of CnCas12f1-sgRNA-DNA |
|---|---|
| 要素 |
|
-超分子 #1: Ternary complex of CnCas12f1-sgRNA-DNA
| 超分子 | 名称: Ternary complex of CnCas12f1-sgRNA-DNA / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Clostridium novyi (ノビイ菌) Clostridium novyi (ノビイ菌) |
-分子 #1: Transposase
| 分子 | 名称: Transposase / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Clostridium novyi (ノビイ菌) Clostridium novyi (ノビイ菌) |
| 分子量 | 理論値: 58.60968 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MITVRKIKLT IMGDKDTRNS QYKWIRDEQY NQYRALNMGM TYLAVNDILY MNESGLEIRT IKDLKDCEKD IDKNKKEIEK LTARLEKEQ NKKNSSSEKL DEIKYKISLV ENKIEDYKLK IVELNKILEE TQKERMDIQK EFKEKYVDDL YQVLDKIPFK H LDNKSLVT ...文字列: MITVRKIKLT IMGDKDTRNS QYKWIRDEQY NQYRALNMGM TYLAVNDILY MNESGLEIRT IKDLKDCEKD IDKNKKEIEK LTARLEKEQ NKKNSSSEKL DEIKYKISLV ENKIEDYKLK IVELNKILEE TQKERMDIQK EFKEKYVDDL YQVLDKIPFK H LDNKSLVT QRIKADIKSD KSNGLLKGER SIRNYKRNFP LMTRGRDLKF KYDDNDDIEI KWMEGIKFKV ILGNRIKNSL EL RHTLHKV IEGKYKICDS SLQFDKNNNL ILNLTLDIPI DIVNKKVSGR VVGVDLGLKI PAYCALNDVE YIKKSIGRID DFL KVRTQM QSRRRRLQIA IQSAKGGKGR VNKLQALERF AEKEKNFAKT YNHFLSSNIV KFAVSNQAEQ INMELLSLKE TQNK SILRN WSYYQLQTMI EYKAQREGIK VKYIDPYHTS QTCSKCGNYE EGQRESQADF ICKKCGYKVN ADYNAARNIA MSNKY ITKK EESKYYKIKE SMV UniProtKB:  Transposase Transposase |
-分子 #2: sgRNA
| 分子 | 名称: sgRNA / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 75.498406 KDa |
| 配列 | 文字列: AACAUAGUUA AACUAAUAAA AACAGGGCGA UUUAACGUCC UAAGGCUGAG AGAAGUUUUU UCUACUCGGC AAGGGUUAAU CUCGAUUGU UGUGUUACCG AUCGAGCGUU UCACAAAAUG CGAGAGAAAU CUCGCAUUUU UAAUUUUGCA GUAAGGCUAG U UUUUAUAU ...文字列: AACAUAGUUA AACUAAUAAA AACAGGGCGA UUUAACGUCC UAAGGCUGAG AGAAGUUUUU UCUACUCGGC AAGGGUUAAU CUCGAUUGU UGUGUUACCG AUCGAGCGUU UCACAAAAUG CGAGAGAAAU CUCGCAUUUU UAAUUUUGCA GUAAGGCUAG U UUUUAUAU AAAUAUGCUA UAACUAGGGU UUUAGUUUAA CUAUGUGAAA UGUAAAUAAU AGAUGUGAAG UCGCUUU |
-分子 #3: DNA (28-MER)
| 分子 | 名称: DNA (28-MER) / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 8.612591 KDa |
| 配列 | 文字列: (DG)(DA)(DA)(DA)(DA)(DG)(DC)(DG)(DA)(DC) (DT)(DT)(DC)(DA)(DC)(DA)(DT)(DC)(DT)(DA) (DT)(DT)(DA)(DG)(DG)(DT)(DT)(DA) |
-分子 #4: DNA (5'-D(*TP*AP*AP*CP*CP*TP*AP*AP*TP*AP*GP*AP*TP*GP*TP*GP*AP*A)-3')
| 分子 | 名称: DNA (5'-D(*TP*AP*AP*CP*CP*TP*AP*AP*TP*AP*GP*AP*TP*GP*TP*GP*AP*A)-3') タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 5.547643 KDa |
| 配列 | 文字列: (DT)(DA)(DA)(DC)(DC)(DT)(DA)(DA)(DT)(DA) (DG)(DA)(DT)(DG)(DT)(DG)(DA)(DA) |
-実験情報
-構造解析
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
|---|---|
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 1.2 µm Bright-field microscopy / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X