+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | cryo-EM structure of cellodextrin phosphorylase from Clostridium thermocellum | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | cellulose / cellodextrin / phosphorolysis / synthesis / CARBOHYDRATE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Acetivibrio thermocellus (バクテリア) Acetivibrio thermocellus (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.3 Å | |||||||||
 データ登録者 データ登録者 | Kuga T / Sunagawa N / Igarashi K | |||||||||
| 資金援助 |  日本, 2件 日本, 2件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: structure and dynamics of cellodextrin phosphorylase from Clostridium thermocellum determine chain length and crystalline packing of highly ordered cellulose II synthesized in vitro 著者: Kuga T / Sunagawa N / Igarashi K | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34504.map.gz emd_34504.map.gz | 217.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34504-v30.xml emd-34504-v30.xml emd-34504.xml emd-34504.xml | 16.7 KB 16.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_34504_fsc.xml emd_34504_fsc.xml | 21.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_34504.png emd_34504.png | 78.5 KB | ||
| Filedesc metadata |  emd-34504.cif.gz emd-34504.cif.gz | 6.2 KB | ||
| その他 |  emd_34504_half_map_1.map.gz emd_34504_half_map_1.map.gz emd_34504_half_map_2.map.gz emd_34504_half_map_2.map.gz | 391.7 MB 391.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34504 http://ftp.pdbj.org/pub/emdb/structures/EMD-34504 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34504 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34504 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_34504_validation.pdf.gz emd_34504_validation.pdf.gz | 800.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_34504_full_validation.pdf.gz emd_34504_full_validation.pdf.gz | 800 KB | 表示 | |
| XML形式データ |  emd_34504_validation.xml.gz emd_34504_validation.xml.gz | 24.1 KB | 表示 | |
| CIF形式データ |  emd_34504_validation.cif.gz emd_34504_validation.cif.gz | 31.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34504 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34504 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34504 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34504 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8h6hMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34504.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34504.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_34504_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
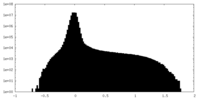
| 投影像・断面図 |
| ||||||||||||
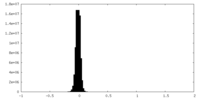
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_34504_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
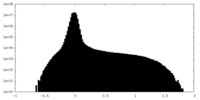
| 投影像・断面図 |
| ||||||||||||
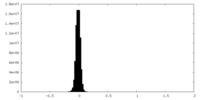
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Homodimeric structure of cellodextrin phosphorylase from Clostrid...
| 全体 | 名称: Homodimeric structure of cellodextrin phosphorylase from Clostridium thermocellum (CtCDP) |
|---|---|
| 要素 |
|
-超分子 #1: Homodimeric structure of cellodextrin phosphorylase from Clostrid...
| 超分子 | 名称: Homodimeric structure of cellodextrin phosphorylase from Clostridium thermocellum (CtCDP) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Acetivibrio thermocellus (バクテリア) / 株: YM4 Acetivibrio thermocellus (バクテリア) / 株: YM4 |
| 分子量 | 理論値: 240 KDa |
-分子 #1: Cellodextrin phosphorylase
| 分子 | 名称: Cellodextrin phosphorylase / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Acetivibrio thermocellus (バクテリア) Acetivibrio thermocellus (バクテリア) |
| 分子量 | 理論値: 112.599523 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MVTKVTARNN KITPVELLNQ KFGNKINLGN FADAVFTDAA FKNVAGIANL PMKAPVMQVL MENCIVSKYL KQFVPDRSVC FVEEGQKFY IVLEDGQKIE VPEDVNKALK ATVSDVKHWA GYLTEDGEHV IDLLKPAPGP HFYVNLLIGN RLGFKRTLQT T PKSVVDRF ...文字列: MVTKVTARNN KITPVELLNQ KFGNKINLGN FADAVFTDAA FKNVAGIANL PMKAPVMQVL MENCIVSKYL KQFVPDRSVC FVEEGQKFY IVLEDGQKIE VPEDVNKALK ATVSDVKHWA GYLTEDGEHV IDLLKPAPGP HFYVNLLIGN RLGFKRTLQT T PKSVVDRF GRGSFRSHAA TQVLATRFDM RQEENGFPAN RQFYLYEDGK QIFYSALIDD NIVEATCKHS CNRTVIKYKT AC NLEITRT IFLVPHKKGF PLATELQRIE IKNASDKARN LSITYTGMFG TGAVHAIFED VTYTNVIMQS AALYNDKGEF IGI TPDYYP EEFKQDTRFV TMIVRNGDEK SFPQSFCTDY NDFVGTGTLE HPAGGCNLNN KLNRKGPGFF ALGAPFTVEP GKTV IIDTF TGLSSSKDNE NYSDAVMLRE LDNLLRYFEK SESVEETLNE IINFHENYGK YFQFNTGNKL FDSGFNRNLA FQVLY QTFM SRSFGQTQKG YREIGFREIQ DLFASMYYFI NIGYQDFVKE LLFEWTANVY KMGYANHNFY WVGKQPGLYS DDSLWL LQA YYRYIIYTKD TSVLNEEVPV ADGNNEKRAV RETLKAIIQY SACISVGDHG LPLLDLADWN DCLKIDSNSI DGATKEK LY YEQLKKTNGK YGDRFMSDYS ESVMNAFLLK LAIDHLAEIA TLDNDTQLAQ QMSELSKEVT DRIQKHAWKE NFFARVLI N RYKDGSYTYL GAKGDKLSAD PNIDGVYFLN SFAWSVLSDV ATDEQIAIMV DVIKKHLLTP YGLRLVTPAD LNKIANDTA TGHYFFGDRE NGAVFKHASM MAVAALIKAA KKVKDNELAK EMARIAYFMI DLVLPYKNLE NPFQVAGNPR ICTQYINTDT GENIGPLLS GTATWLNLNL ISLAGIEYTR DGISFNPILR EEETQLNFTL KAPKCSYKFS ITKPVGFARM ESSEYELFVD G QKIDNTVI PMYTDEKEHI VTLKFKHHHH HH UniProtKB: Cellodextrin phosphorylase |
-分子 #2: CHLORIDE ION
| 分子 | 名称: CHLORIDE ION / タイプ: ligand / ID: 2 / コピー数: 4 / 式: CL |
|---|---|
| 分子量 | 理論値: 35.453 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.0 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: 20 mM Tris-HCl, 120 mM NaCl, 0.1 mM DTT | ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 280 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 49.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー








 Z
Z Y
Y X
X