登録情報 データベース : EMDB / ID : EMD-34398タイトル Cryo-EM structure of Synechocystis sp. PCC 6803 CTP-bound RPitc 複合体 : The complex of Synechocystis sp. PCC 6803 CTP-bound RPitcタンパク質・ペプチド : x 6種DNA : x 2種RNA : x 1種リガンド : x 3種 / / / / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
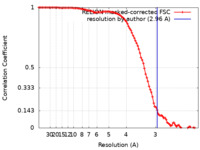
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Synechocystis sp. PCC 6803 (バクテリア) / synthetic construct (人工物) 手法 / / 解像度 : 2.96 Å You LL / Shen LQ / Zhang Y 資金援助 Organization Grant number 国 Chinese Academy of Sciences
ジャーナル : Proc Natl Acad Sci U S A / 年 : 2023タイトル : An SI3-σ arch stabilizes cyanobacteria transcription initiation complex.著者 : Liqiang Shen / Giorgio Lai / Linlin You / Jing Shi / Xiaoxian Wu / Maria Puiu / Zhanxi Gu / Yu Feng / Yulia Yuzenkova / Yu Zhang / 要旨 : Multisubunit RNA polymerases (RNAPs) associate with initiation factors (σ in bacteria) to start transcription. The σ factors are responsible for recognizing and unwinding promoter DNA in all ... Multisubunit RNA polymerases (RNAPs) associate with initiation factors (σ in bacteria) to start transcription. The σ factors are responsible for recognizing and unwinding promoter DNA in all bacterial RNAPs. Here, we report two cryo-EM structures of cyanobacterial transcription initiation complexes at near-atomic resolutions. The structures show that cyanobacterial RNAP forms an "SI3-σ" arch interaction between domain 2 of σ (σ) and sequence insertion 3 (SI3) in the mobile catalytic domain Trigger Loop (TL). The "SI3-σ" arch facilitates transcription initiation from promoters of different classes through sealing the main cleft and thereby stabilizing the RNAP-promoter DNA open complex. Disruption of the "SI3-σ" arch disturbs cyanobacteria growth and stress response. Our study reports the structure of cyanobacterial RNAP and a unique mechanism for its transcription initiation. Our data suggest functional plasticity of SI3 and provide the foundation for further research into cyanobacterial and chloroplast transcription. 履歴 登録 2022年9月27日 - ヘッダ(付随情報) 公開 2023年4月19日 - マップ公開 2023年4月19日 - 更新 2024年10月30日 - 現状 2024年10月30日 処理サイト : RCSB / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 データ登録者
データ登録者 中国, 1件
中国, 1件  引用
引用 ジャーナル: Proc Natl Acad Sci U S A / 年: 2023
ジャーナル: Proc Natl Acad Sci U S A / 年: 2023

 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_34398.map.gz
emd_34398.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-34398-v30.xml
emd-34398-v30.xml emd-34398.xml
emd-34398.xml EMDBヘッダ
EMDBヘッダ emd_34398_fsc.xml
emd_34398_fsc.xml FSCデータファイル
FSCデータファイル emd_34398.png
emd_34398.png emd_34398_msk_1.map
emd_34398_msk_1.map マスクマップ
マスクマップ emd-34398.cif.gz
emd-34398.cif.gz emd_34398_additional_1.map.gz
emd_34398_additional_1.map.gz emd_34398_half_map_1.map.gz
emd_34398_half_map_1.map.gz emd_34398_half_map_2.map.gz
emd_34398_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-34398
http://ftp.pdbj.org/pub/emdb/structures/EMD-34398 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34398
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34398 emd_34398_validation.pdf.gz
emd_34398_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_34398_full_validation.pdf.gz
emd_34398_full_validation.pdf.gz emd_34398_validation.xml.gz
emd_34398_validation.xml.gz emd_34398_validation.cif.gz
emd_34398_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34398
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34398 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34398
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34398 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_34398.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_34398.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) emd_34398_msk_1.map
emd_34398_msk_1.map 試料の構成要素
試料の構成要素 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 画像解析
画像解析
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)