+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of baculovirus LEF-3 in complex with ssDNA | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ssDNA / SSB / Baculovirus / DNA BINDING PROTEIN | |||||||||
| 機能・相同性 | Nucleopolyhedrovirus late expression factor 3 / Nucleopolyhedrovirus late expression factor 3 (LEF-3) / regulation of DNA-templated transcription / DNA binding / Lef3 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Helicoverpa armigera nucleopolyhedrovirus (ウイルス) / synthetic construct (人工物) Helicoverpa armigera nucleopolyhedrovirus (ウイルス) / synthetic construct (人工物) | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Yin J / Fu Y / Rao G / Li Z / Cao S | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2022 ジャーナル: Nucleic Acids Res / 年: 2022タイトル: Structural transitions during the cooperative assembly of baculovirus single-stranded DNA-binding protein on ssDNA. 著者: Jiayi Yin / Yan Fu / Guibo Rao / Zhiqiang Li / Kexing Tian / Tingting Chong / Kai Kuang / Manli Wang / Zhihong Hu / Sheng Cao /  要旨: Single-stranded DNA-binding proteins (SSBs) interact with single-stranded DNA (ssDNA) to form filamentous structures with various degrees of cooperativity, as a result of intermolecular interactions ...Single-stranded DNA-binding proteins (SSBs) interact with single-stranded DNA (ssDNA) to form filamentous structures with various degrees of cooperativity, as a result of intermolecular interactions between neighboring SSB subunits on ssDNA. However, it is still challenging to perform structural studies on SSB-ssDNA filaments at high resolution using the most studied SSB models, largely due to the intrinsic flexibility of these nucleoprotein complexes. In this study, HaLEF-3, an SSB protein from Helicoverpa armigera nucleopolyhedrovirus, was used for in vitro assembly of SSB-ssDNA filaments, which were structurally studied at atomic resolution using cryo-electron microscopy. Combined with the crystal structure of ssDNA-free HaLEF-3 octamers, our results revealed that the three-dimensional rearrangement of HaLEF-3 induced by an internal hinge-bending movement is essential for the formation of helical SSB-ssDNA complexes, while the contacting interface between adjacent HaLEF-3 subunits remains basically intact. We proposed a local cooperative SSB-ssDNA binding model, in which, triggered by exposure to oligonucleotides, HaLEF-3 molecules undergo ring-to-helix transition to initiate continuous SSB-SSB interactions along ssDNA. Unique structural features revealed by the assembly of HaLEF-3 on ssDNA suggest that HaLEF-3 may represent a new class of SSB. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34010.map.gz emd_34010.map.gz | 49.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34010-v30.xml emd-34010-v30.xml emd-34010.xml emd-34010.xml | 15.3 KB 15.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_34010.png emd_34010.png | 159.3 KB | ||
| Filedesc metadata |  emd-34010.cif.gz emd-34010.cif.gz | 5.5 KB | ||
| その他 |  emd_34010_half_map_1.map.gz emd_34010_half_map_1.map.gz emd_34010_half_map_2.map.gz emd_34010_half_map_2.map.gz | 48.9 MB 48.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34010 http://ftp.pdbj.org/pub/emdb/structures/EMD-34010 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34010 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34010 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_34010_validation.pdf.gz emd_34010_validation.pdf.gz | 878.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_34010_full_validation.pdf.gz emd_34010_full_validation.pdf.gz | 878.5 KB | 表示 | |
| XML形式データ |  emd_34010_validation.xml.gz emd_34010_validation.xml.gz | 11.8 KB | 表示 | |
| CIF形式データ |  emd_34010_validation.cif.gz emd_34010_validation.cif.gz | 13.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34010 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34010 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34010 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34010 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34010.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34010.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.95 Å | ||||||||||||||||||||||||||||||||||||
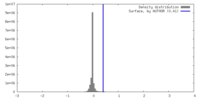
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_34010_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
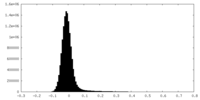
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_34010_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
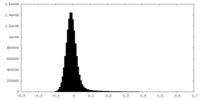
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Baculovirus LEF-3 in complex with ssDNA
| 全体 | 名称: Baculovirus LEF-3 in complex with ssDNA |
|---|---|
| 要素 |
|
-超分子 #1: Baculovirus LEF-3 in complex with ssDNA
| 超分子 | 名称: Baculovirus LEF-3 in complex with ssDNA / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|
-超分子 #2: LEF-3
| 超分子 | 名称: LEF-3 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Helicoverpa armigera nucleopolyhedrovirus (ウイルス) Helicoverpa armigera nucleopolyhedrovirus (ウイルス) |
-超分子 #3: ssDNA
| 超分子 | 名称: ssDNA / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|
-分子 #1: Lef3
| 分子 | 名称: Lef3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Helicoverpa armigera nucleopolyhedrovirus (ウイルス) Helicoverpa armigera nucleopolyhedrovirus (ウイルス) |
| 分子量 | 理論値: 47.637707 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH SSGLVPRGSH MASMTGGQQM GRGSMSNMDI SPVKQLIDIE NDDAMNTPEK GMKRPLMRTM SSVEEPQAKM AKLRTLNVK GQLLTKTTMS INNEDYYLFK FLVNNKSIDY YGTQTQFFSL INNKTYELVL QYSRKKLLIK SYEQCEDEDL L MTVCKSVT ...文字列: MGSSHHHHHH SSGLVPRGSH MASMTGGQQM GRGSMSNMDI SPVKQLIDIE NDDAMNTPEK GMKRPLMRTM SSVEEPQAKM AKLRTLNVK GQLLTKTTMS INNEDYYLFK FLVNNKSIDY YGTQTQFFSL INNKTYELVL QYSRKKLLIK SYEQCEDEDL L MTVCKSVT FQEFCANEIK SLLAKFLYGF KIYGSSNVYK LVFVILLEDN NGTINGVQVE MMSDFKRLSG AFKNHVIENE ND LFDCMYK SEEKYFNLYR IKCNHNANNY KSLSLSSNSQ LERLETDDSM FEYEFQYDYT VNISRSNKII QKHRVTGNFT SER NIYQNS DRFVISYDTA NEKIKTSIYN RMENAESKTD YDTSITLKDV TLSQLNSLIE SNLVQVDVYL VTDPNNVKNN VIAG ITKIE IDGTYEPL UniProtKB: Lef3 |
-分子 #2: DNA (28-MER)
| 分子 | 名称: DNA (28-MER) / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 8.724834 KDa |
| 配列 | 文字列: (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 緩衝液 | pH: 6.3 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 15.105 Å 想定した対称性 - らせんパラメータ - ΔΦ: -108.281 ° 想定した対称性 - らせんパラメータ - 軸対称性: C1 (非対称) 解像度のタイプ: BY AUTHOR / 解像度: 3.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 530580 |
|---|---|
| 初期モデル | モデルのタイプ: NONE |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)