+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human Slo1-LRRC26 complex with C1 symmetry | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of voltage-gated potassium channel activity / monoatomic ion-gated channel activity / potassium channel activator activity / ligand-gated monoatomic cation channel activity / voltage-gated potassium channel activity / potassium channel activity / potassium channel regulator activity / voltage-gated potassium channel complex / potassium ion transmembrane transport / transmembrane transporter binding ...positive regulation of voltage-gated potassium channel activity / monoatomic ion-gated channel activity / potassium channel activator activity / ligand-gated monoatomic cation channel activity / voltage-gated potassium channel activity / potassium channel activity / potassium channel regulator activity / voltage-gated potassium channel complex / potassium ion transmembrane transport / transmembrane transporter binding / cytoskeleton / extracellular exosome / membrane / plasma membrane / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
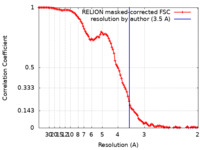
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Yamanouchi D / Nureki O | |||||||||
| 資金援助 |  日本, 1件 日本, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Structural basis for dual allosteric gating modulation of Slo1-LRRC channel complex 著者: Yamanouchi D | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33976.map.gz emd_33976.map.gz | 15.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33976-v30.xml emd-33976-v30.xml emd-33976.xml emd-33976.xml | 15.4 KB 15.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_33976_fsc.xml emd_33976_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_33976.png emd_33976.png | 106.1 KB | ||
| マスクデータ |  emd_33976_msk_1.map emd_33976_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-33976.cif.gz emd-33976.cif.gz | 6.2 KB | ||
| その他 |  emd_33976_half_map_1.map.gz emd_33976_half_map_1.map.gz emd_33976_half_map_2.map.gz emd_33976_half_map_2.map.gz | 80.9 MB 80.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33976 http://ftp.pdbj.org/pub/emdb/structures/EMD-33976 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33976 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33976 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_33976_validation.pdf.gz emd_33976_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_33976_full_validation.pdf.gz emd_33976_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_33976_validation.xml.gz emd_33976_validation.xml.gz | 17.6 KB | 表示 | |
| CIF形式データ |  emd_33976_validation.cif.gz emd_33976_validation.cif.gz | 23 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33976 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33976 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33976 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33976 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7ynzMC  7yo0C  7yo1C  7yo2C  7yo3C  7yo4C  7yo5C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33976.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33976.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.996 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_33976_msk_1.map emd_33976_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_33976_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_33976_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : human Slo1-LRRC26 complex
| 全体 | 名称: human Slo1-LRRC26 complex |
|---|---|
| 要素 |
|
-超分子 #1: human Slo1-LRRC26 complex
| 超分子 | 名称: human Slo1-LRRC26 complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 616 kDa/nm |
-分子 #1: Calcium-activated potassium channel subunit alpha-1
| 分子 | 名称: Calcium-activated potassium channel subunit alpha-1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 119.4585 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDALIIPVTM EVPCDSRGQR MWWAFLASSM VTFFGGLFII LLWRTLKYLW TVCCHCGGKT KEAQKINNGS SQADGTLKPV DEKEEAVAA EVGWMTSVKD WAGVMISAQT LTGRVLVVLV FALSIGALVI YFIDSSNPIE SCQNFYKDFT LQIDMAFNVF F LLYFGLRF ...文字列: MDALIIPVTM EVPCDSRGQR MWWAFLASSM VTFFGGLFII LLWRTLKYLW TVCCHCGGKT KEAQKINNGS SQADGTLKPV DEKEEAVAA EVGWMTSVKD WAGVMISAQT LTGRVLVVLV FALSIGALVI YFIDSSNPIE SCQNFYKDFT LQIDMAFNVF F LLYFGLRF IAANDKLWFW LEVNSVVDFF TVPPVFVSVY LNRSWLGLRF LRALRLIQFS EILQFLNILK TSNSIKLVNL LS IFISTWL TAAGFIHLVE NSGDPWENFQ NNQALTYWEC VYLLMVTMST VGYGDVYAKT TLGRLFMVFF ILGGLAMFAS YVP EIIELI GNRKKYGGSY SAVSGRKHIV VCGHITLESV SNFLKDFLHK DRDDVNVEIV FLHNISPNLE LEALFKRHFT QVEF YQGSV LNPHDLARVK IESADACLIL ANKYCADPDA EDASNIMRVI SIKNYHPKIR IITQMLQYHN KAHLLNIPSW NWKEG DDAI CLAELKLGFI AQSCLAQGLS TMLANLFSMR SFIKIEEDTW QKYYLEGVSN EMYTEYLSSA FVGLSFPTVC ELCFVK LKL LMIAIEYKSA NRESRSRSRI LINPGNHLKI QEGTLGFFIA SDAKEVKRAF FYCKACHDDI TDPKRIKKCG CKRLEDE QP STLSPKKKQR NGGMRNSPNT SPKLMRHDPL LIPGNDQIDN MDSNVKKYDS TGMFHWCAPK EIEKVILTRS EAAMTVLS G HVVVCIFGDV SSALIGLRNL VMPLRASNFH YHELKHIVFV GSIEYLKREW ETLHNFPKVS ILPGTPLSRA DLRAVNINL CDMCVILSAN QNNIDDTSLQ DKECILASLN IKSMQFDDSI GVLQANSQGF TPPGMDRSSP DNSPVHGMLR QPSITTGVNI PIITELVND TNVQFLDQDD DDDPDTELYL TQPFACGTAF AVSVLDSLMS ATYFNDNILT LIRTLVTGGA TPELEALIAE E NALRGGYS TPQTLANRDR CRVAQLALLD GPFADLGDGG CYGDLFCKAL KTYNMLCFGI YRLRDAHLST PSQCTKRYVI TN PPYEFEL VPTDLIFCLM QFD UniProtKB: Calcium-activated potassium channel subunit alpha-1 |
-分子 #2: Leucine-rich repeat-containing protein 26
| 分子 | 名称: Leucine-rich repeat-containing protein 26 / タイプ: protein_or_peptide / ID: 2 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 34.893629 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MRGPSWSRPR PLLLLLLLLS PWPVWAQVSA TASPSGSLGA PDCPEVCTCV PGGLASCSAL SLPAVPPGLS LRLRALLLDH NRVRALPPG AFAGAGALQR LDLRENGLHS VHVRAFWGLG ALQLLDLSAN QLEALAPGTF APLRALRNLS LAGNRLARLE P AALGALPL ...文字列: MRGPSWSRPR PLLLLLLLLS PWPVWAQVSA TASPSGSLGA PDCPEVCTCV PGGLASCSAL SLPAVPPGLS LRLRALLLDH NRVRALPPG AFAGAGALQR LDLRENGLHS VHVRAFWGLG ALQLLDLSAN QLEALAPGTF APLRALRNLS LAGNRLARLE P AALGALPL LRSLSLQDNE LAALAPGLLG RLPALDALHL RGNPWGCGCA LRPLCAWLRR HPLPASEAET VLCVWPGRLT LS PLTAFSD AAFSHCAQPL ALRDLAVVYT LGPASFLVSL ASCLALGSGL TACRARRRRL RTAALRPPRP PDPNPDPDPH GCA SPADPG SPAAAAQA UniProtKB: Leucine-rich repeat-containing protein 26 |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 4 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #4: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 4 / コピー数: 8 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.0 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 48.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)