登録情報 データベース : EMDB / ID : EMD-33515タイトル The cryo-EM structure of an AlpA-loading complex 複合体 : AlpA-loading complexタンパク質・ペプチド : DNA-directed RNA polymerase subunit alphaタンパク質・ペプチド : DNA-directed RNA polymerase subunit betaタンパク質・ペプチド : DNA-directed RNA polymerase subunit beta'タンパク質・ペプチド : DNA-directed RNA polymerase subunit omegaタンパク質・ペプチド : RNA polymerase sigma factor RpoDDNA : nontemplate strand DNARNA : RNADNA : template strand DNAタンパク質・ペプチド : AlpAリガンド : MAGNESIUM ION機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Pseudomonas aeruginosa (緑膿菌)手法 / / 解像度 : 3.3 Å Wen A / Feng Y 資金援助 Organization Grant number 国 National Natural Science Foundation of China (NSFC) 31970040
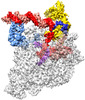
ジャーナル : Nucleic Acids Res / 年 : 2022タイトル : Structural basis of AlpA-dependent transcription antitermination.著者 : Aijia Wen / Minxing Zhao / Sha Jin / Yuan-Qiang Lu / Yu Feng / 要旨 : AlpA positively regulates a programmed cell death pathway linked to the virulence of Pseudomonas aeruginosa by recognizing an AlpA binding element within the promoter, then binding RNA polymerase ... AlpA positively regulates a programmed cell death pathway linked to the virulence of Pseudomonas aeruginosa by recognizing an AlpA binding element within the promoter, then binding RNA polymerase directly and allowing it to bypass an intrinsic terminator positioned downstream. Here, we report the single-particle cryo-electron microscopy structures of both an AlpA-loading complex and an AlpA-loaded complex. These structures indicate that the C-terminal helix-turn-helix motif of AlpA binds to the AlpA binding element and that the N-terminal segment of AlpA forms a narrow ring inside the RNA exit channel. AlpA was also revealed to render RNAP resistant to termination signals by prohibiting RNA hairpin formation in the RNA exit channel. Structural analysis predicted that AlpA, 21Q, λQ and 82Q share the same mechanism of transcription antitermination. 履歴 登録 2022年6月1日 - ヘッダ(付随情報) 公開 2022年7月20日 - マップ公開 2022年7月20日 - 更新 2022年8月24日 - 現状 2022年8月24日 処理サイト : PDBj / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 機能・相同性情報
機能・相同性情報
 データ登録者
データ登録者 中国, 1件
中国, 1件  引用
引用 ジャーナル: Nucleic Acids Res / 年: 2022
ジャーナル: Nucleic Acids Res / 年: 2022
 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_33515.map.gz
emd_33515.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-33515-v30.xml
emd-33515-v30.xml emd-33515.xml
emd-33515.xml EMDBヘッダ
EMDBヘッダ emd_33515.png
emd_33515.png emd_33515_msk_1.map
emd_33515_msk_1.map マスクマップ
マスクマップ emd_33515_half_map_1.map.gz
emd_33515_half_map_1.map.gz emd_33515_half_map_2.map.gz
emd_33515_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-33515
http://ftp.pdbj.org/pub/emdb/structures/EMD-33515 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33515
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33515 emd_33515_validation.pdf.gz
emd_33515_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_33515_full_validation.pdf.gz
emd_33515_full_validation.pdf.gz emd_33515_validation.xml.gz
emd_33515_validation.xml.gz emd_33515_validation.cif.gz
emd_33515_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33515
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33515 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33515
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33515

 F&H 検索
F&H 検索 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_33515.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_33515.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) emd_33515_msk_1.map
emd_33515_msk_1.map 試料の構成要素
試料の構成要素 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 画像解析
画像解析 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)