+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
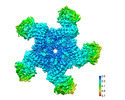
| タイトル | Cryo EM structure of oligomeric complex formed by wheat CNL Sr35 and the effector AvrSr35 of the wheat stem rust pathogen | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | LRR / CC / direct recognition / PLANT PROTEIN-INHIBITOR COMPLEX | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Puccinia graminis f. sp. tritici (菌類) Puccinia graminis f. sp. tritici (菌類) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Alexander F / Li ET / Aaron L / Deng YN / Sun Y / Chai JJ | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2022 ジャーナル: Nature / 年: 2022タイトル: A wheat resistosome defines common principles of immune receptor channels. 著者: Alexander Förderer / Ertong Li / Aaron W Lawson / Ya-Nan Deng / Yue Sun / Elke Logemann / Xiaoxiao Zhang / Jie Wen / Zhifu Han / Junbiao Chang / Yuhang Chen / Paul Schulze-Lefert / Jijie Chai /   要旨: Plant intracellular nucleotide-binding leucine-rich repeat receptors (NLRs) detect pathogen effectors to trigger immune responses. Indirect recognition of a pathogen effector by the dicotyledonous ...Plant intracellular nucleotide-binding leucine-rich repeat receptors (NLRs) detect pathogen effectors to trigger immune responses. Indirect recognition of a pathogen effector by the dicotyledonous Arabidopsis thaliana coiled-coil domain containing NLR (CNL) ZAR1 induces the formation of a large hetero-oligomeric protein complex, termed the ZAR1 resistosome, which functions as a calcium channel required for ZAR1-mediated immunity. Whether the resistosome and channel activities are conserved among plant CNLs remains unknown. Here we report the cryo-electron microscopy structure of the wheat CNL Sr35 in complex with the effector AvrSr35 of the wheat stem rust pathogen. Direct effector binding to the leucine-rich repeats of Sr35 results in the formation of a pentameric Sr35-AvrSr35 complex, which we term the Sr35 resistosome. Wheat Sr35 and Arabidopsis ZAR1 resistosomes bear striking structural similarities, including an arginine cluster in the leucine-rich repeats domain not previously recognized as conserved, which co-occurs and forms intramolecular interactions with the 'EDVID' motif in the coiled-coil domain. Electrophysiological measurements show that the Sr35 resistosome exhibits non-selective cation channel activity. These structural insights allowed us to generate new variants of closely related wheat and barley orphan NLRs that recognize AvrSr35. Our data support the evolutionary conservation of CNL resistosomes in plants and demonstrate proof of principle for structure-based engineering of NLRs for crop improvement. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33112.map.gz emd_33112.map.gz | 15.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33112-v30.xml emd-33112-v30.xml emd-33112.xml emd-33112.xml | 17.1 KB 17.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_33112.png emd_33112.png | 112.5 KB | ||
| Filedesc metadata |  emd-33112.cif.gz emd-33112.cif.gz | 6.2 KB | ||
| その他 |  emd_33112_half_map_1.map.gz emd_33112_half_map_1.map.gz emd_33112_half_map_2.map.gz emd_33112_half_map_2.map.gz | 10.1 MB 10.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33112 http://ftp.pdbj.org/pub/emdb/structures/EMD-33112 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33112 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33112 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_33112_validation.pdf.gz emd_33112_validation.pdf.gz | 526.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_33112_full_validation.pdf.gz emd_33112_full_validation.pdf.gz | 526.1 KB | 表示 | |
| XML形式データ |  emd_33112_validation.xml.gz emd_33112_validation.xml.gz | 14.4 KB | 表示 | |
| CIF形式データ |  emd_33112_validation.cif.gz emd_33112_validation.cif.gz | 17.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33112 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33112 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33112 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33112 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7xc2MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33112.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33112.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_33112_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
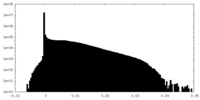
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_33112_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
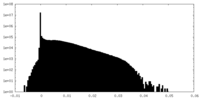
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo EM structure of oligomeric complex formed by wheat CNL Sr35 ...
| 全体 | 名称: Cryo EM structure of oligomeric complex formed by wheat CNL Sr35 and the effector AvrSr35 of the wheat stem rust pathogen |
|---|---|
| 要素 |
|
-超分子 #1: Cryo EM structure of oligomeric complex formed by wheat CNL Sr35 ...
| 超分子 | 名称: Cryo EM structure of oligomeric complex formed by wheat CNL Sr35 and the effector AvrSr35 of the wheat stem rust pathogen タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #2: CNL9
| 超分子 | 名称: CNL9 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Puccinia graminis f. sp. tritici (菌類) Puccinia graminis f. sp. tritici (菌類) |
-超分子 #3: Avirulence factor
| 超分子 | 名称: Avirulence factor / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|
-分子 #1: CNL9
| 分子 | 名称: CNL9 / タイプ: protein_or_peptide / ID: 1 / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 102.046242 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: TITLEKKVRK GIESLITELK LMQAVLSKVS KVPADQLDEG VKIWAGNVKE LSYQMEDIVD AFMVRVGDGG ESTNPKNRVK KILKKVKKL FKNGKDLHRI SAALEEVVLQ AKQLAELRQR YEQEMRDTSA QTSVDPRMMA LYTDVTELVG IEETRDKLIN M LTEGDDWS ...文字列: TITLEKKVRK GIESLITELK LMQAVLSKVS KVPADQLDEG VKIWAGNVKE LSYQMEDIVD AFMVRVGDGG ESTNPKNRVK KILKKVKKL FKNGKDLHRI SAALEEVVLQ AKQLAELRQR YEQEMRDTSA QTSVDPRMMA LYTDVTELVG IEETRDKLIN M LTEGDDWS KHPLKTISIV GFGGLGKTTL AKAAYDKIKV QFDCGAFVSV SRNPEMKKVL KDILYGLDKV KYENIHNAAR DE KYLIDDI IEFLNDKRYL IVIDDIWNEK AWELIKCAFS KKSPGSRLIT TTRNVSVSEA CCSSEDDIYR MEPLSNDVSR TLF CKRIFS QEEGCPQELL KVSEEILKKC GGVPLAIITI ASLLANKGHI KAKDEWYALL SSIGHGLTKN RSLEQMKKIL LFSY YDLPS YLKPCLLYLS IFPEDREIRR ARLVWRWISE GFVYSEKQDI SLYELGDSYF NELVNRSMIQ PIGIDDEGKV KACRV HDMV LDLICSLSSE ENFVTILDDP RRKMPNSESK VRRLSIQNSK IDVDTTRMEH MRSVTVFSDN VVGKVLDISR FKVLRV LDL EGCHVSDVGY VGNLLHLRYL GLKGTHVKDL PMEVGKLQFL LTLDLRGTKI EVLPWSVVQL RRLMCLYVDY GMKLPSG IG NLTFLEVLDD LGLSDVDLDF VKELGRLTKL RVLRLDFHGF DQSMGKALEE SISNMYKLDS LDVFVNRGLI NCLSEHWV P PPRLCRLAFP SKRSWFKTLP SWINPSSLPL LSYLDITLFE VRSEDIQLLG TLPALVYLEI WNYSVFEEAH EVEAPVLSS GAALFPCATE CRFIGIGAVP SMFPQGAAPR LKRLWFTFPA KWSSSENIGL GMRHLPSLQR VVVDVISEGA SREEADEAEA ALRAAAEDH PNRPILDIW UniProtKB: CNL9 |
-分子 #2: Avirulence factor
| 分子 | 名称: Avirulence factor / タイプ: protein_or_peptide / ID: 2 / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Puccinia graminis f. sp. tritici (菌類) Puccinia graminis f. sp. tritici (菌類) |
| 分子量 | 理論値: 50.51148 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: VNFPFPKKMI TESNSKDIRE YLASTFPFEQ QSTILDSVKS IAKVQIDDRK AFDLQLKFRQ ENLAELKDQI ILSLGANNGN QNWQKLLDY TNKLDELSNT KISPEEFIEE IQKVLYKVKL ESTSTSKLYS QFNLSIQDFA LQIIHSKYKS NQISQNDLLK L ITEDEMLK ...文字列: VNFPFPKKMI TESNSKDIRE YLASTFPFEQ QSTILDSVKS IAKVQIDDRK AFDLQLKFRQ ENLAELKDQI ILSLGANNGN QNWQKLLDY TNKLDELSNT KISPEEFIEE IQKVLYKVKL ESTSTSKLYS QFNLSIQDFA LQIIHSKYKS NQISQNDLLK L ITEDEMLK ILAKTKVLTY KMKYFDSASK MGINKYISTE MMDLDWQFSH YKTFNDALKK NKASDSSYLG WLTHGYSIKY GL SPNNERS MFFQDGRKYA ELYAFSKSPH RKIIPGEHLK DLLAKINKSK GIFLDQNALL DKRIYAFHEL NTLETHFPGI TSS FTDDLK SNYRKKMESV SLTCQVLQEI GNIHRFIESK VPYHSSTEYG LFSIPKIFSI PIDYKHGEKE NLVSYVDFLY STAH ERILQ DNSINQLCLD PLQESLNRIK SNIPVFFNL UniProtKB: Avirulence factor |
-分子 #3: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 5 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: DARK FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.5 µm |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.0 Å / 解像度の算出法: FSC 0.33 CUT-OFF / 使用した粒子像数: 230485 |
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X