+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of RNC-RAC complex in presence of Ssb from S. cerevisiae 2 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | RAC / co-translational folding / TRANSLATION / TRANSLATION-RNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報translational frameshifting / 'de novo' cotranslational protein folding / Regulation of HSF1-mediated heat shock response / protein folding chaperone complex / regulation of translational fidelity / ribosomal subunit export from nucleus / Hsp70 protein binding / rRNA processing / protein folding / ribosome binding ...translational frameshifting / 'de novo' cotranslational protein folding / Regulation of HSF1-mediated heat shock response / protein folding chaperone complex / regulation of translational fidelity / ribosomal subunit export from nucleus / Hsp70 protein binding / rRNA processing / protein folding / ribosome binding / transcription coactivator activity / ribosome / intracellular signal transduction / nucleolus / mitochondrion / DNA binding / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / ネガティブ染色法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Chen Y / Gao N | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structural remodeling of ribosome associated Hsp40-Hsp70 chaperones during co-translational folding. 著者: Yan Chen / Bin Tsai / Ningning Li / Ning Gao /  要旨: Ribosome associated complex (RAC), an obligate heterodimer of HSP40 and HSP70 (Zuo1 and Ssz1 in yeast), is conserved in eukaryotes and functions as co-chaperone for another HSP70 (Ssb1/2 in yeast) to ...Ribosome associated complex (RAC), an obligate heterodimer of HSP40 and HSP70 (Zuo1 and Ssz1 in yeast), is conserved in eukaryotes and functions as co-chaperone for another HSP70 (Ssb1/2 in yeast) to facilitate co-translational folding of nascent polypeptides. Many mechanistic details, such as the coordination of one HSP40 with two HSP70s and the dynamic interplay between RAC-Ssb and growing nascent chains, remain unclear. Here, we report three sets of structures of RAC-containing ribosomal complexes isolated from Saccharomyces cerevisiae. Structural analyses indicate that RAC on the nascent-chain-free ribosome is in an autoinhibited conformation, and in the presence of a nascent chain at the peptide tunnel exit (PTE), RAC undergoes large-scale structural remodeling to make Zuo1 J-Domain more accessible to Ssb. Our data also suggest a role of Zuo1 in orienting Ssb-SBD proximal to the PTE for easy capture of the substrate. Altogether, in accordance with previous data, our work suggests a sequence of structural remodeling events for RAC-Ssb during co-translational folding, triggered by the binding and passage of growing nascent chain from one to another. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_32978.map.gz emd_32978.map.gz | 140.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-32978-v30.xml emd-32978-v30.xml emd-32978.xml emd-32978.xml | 14 KB 14 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_32978.png emd_32978.png | 70.3 KB | ||
| Filedesc metadata |  emd-32978.cif.gz emd-32978.cif.gz | 5.1 KB | ||
| その他 |  emd_32978_half_map_1.map.gz emd_32978_half_map_1.map.gz emd_32978_half_map_2.map.gz emd_32978_half_map_2.map.gz | 140.9 MB 140.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-32978 http://ftp.pdbj.org/pub/emdb/structures/EMD-32978 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32978 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32978 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_32978_validation.pdf.gz emd_32978_validation.pdf.gz | 943.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_32978_full_validation.pdf.gz emd_32978_full_validation.pdf.gz | 943.2 KB | 表示 | |
| XML形式データ |  emd_32978_validation.xml.gz emd_32978_validation.xml.gz | 14.9 KB | 表示 | |
| CIF形式データ |  emd_32978_validation.cif.gz emd_32978_validation.cif.gz | 17.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32978 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32978 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32978 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32978 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7x34MC  7x3kC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_32978.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_32978.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.356 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_32978_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
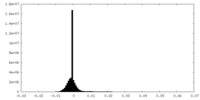
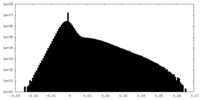
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_32978_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
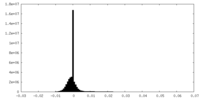
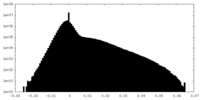
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : RNC-RAC complex in presence of Ssb from S. cerevisiae 2
| 全体 | 名称: RNC-RAC complex in presence of Ssb from S. cerevisiae 2 |
|---|---|
| 要素 |
|
-超分子 #1: RNC-RAC complex in presence of Ssb from S. cerevisiae 2
| 超分子 | 名称: RNC-RAC complex in presence of Ssb from S. cerevisiae 2 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Zuotin
| 分子 | 名称: Zuotin / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 10.619055 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: ASAKADKKKA KEAAKAAKKK NKRAIRNSAK EADYFGDADK ATTIDEQVGL IVDSLNDEEL VSTADKIKAN AAGAKEVLKE SAKTIVDSG KLPSSLLSYF V UniProtKB: Zuotin |
-分子 #2: RNA (130-mer)
| 分子 | 名称: RNA (130-mer) / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 42.010996 KDa |
| 配列 | 文字列: CGCCCGUCGC UAGUACCGAU UGAAUGGCUU AGUGAGGCCU CAGGAUCUGC UUAGAGAAGG GGGCAACUCC AUCUCAGAGC GGAGAAUUU GGACAAACUU GGUCAUUUAG AGGAACUAAA AGUCGUAACA A GENBANK: GENBANK: CP011821.1 |
-実験情報
-構造解析
| 手法 | ネガティブ染色法, クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 染色 | タイプ: NEGATIVE / 材質: Uranyl Acetate |
| 凍結 | 凍結剤: NITROGEN |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 35.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: DARK FIELD / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 65816 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-7x34: |
 ムービー
ムービー コントローラー
コントローラー










 Z
Z Y
Y X
X