+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of AtSLAC1 S59A mutant | |||||||||||||||||||||
 マップデータ マップデータ | Cryo-EM map of the AtSLAC1 S59A mutant | |||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
 キーワード キーワード | anion channel / SLAC1 / stomata / guard cell / TRANSPORT PROTEIN | |||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報response to humidity / stomatal closure / regulation of stomatal opening / inorganic anion transport / voltage-gated monoatomic anion channel activity / regulation of stomatal closure / stomatal movement / response to ozone / intracellular monoatomic ion homeostasis / organic anion transport ...response to humidity / stomatal closure / regulation of stomatal opening / inorganic anion transport / voltage-gated monoatomic anion channel activity / regulation of stomatal closure / stomatal movement / response to ozone / intracellular monoatomic ion homeostasis / organic anion transport / response to carbon dioxide / monoatomic anion transmembrane transporter activity / response to abscisic acid / multicellular organismal-level water homeostasis / monoatomic anion transport / abscisic acid-activated signaling pathway / response to light stimulus / protein phosphatase binding / protein kinase binding / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||||||||||||||
| 生物種 |  | |||||||||||||||||||||
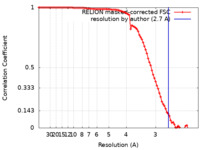
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Sun L / Liu X | |||||||||||||||||||||
| 資金援助 |  中国, 6件 中国, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structure of the Arabidopsis guard cell anion channel SLAC1 suggests activation mechanism by phosphorylation. 著者: Yawen Li / Yinan Ding / Lili Qu / Xinru Li / Qinxuan Lai / Pingxia Zhao / Yongxiang Gao / Chengbin Xiang / Chunlei Cang / Xin Liu / Linfeng Sun /  要旨: Stomata play a critical role in the regulation of gas exchange and photosynthesis in plants. Stomatal closure participates in multiple stress responses, and is regulated by a complex network ...Stomata play a critical role in the regulation of gas exchange and photosynthesis in plants. Stomatal closure participates in multiple stress responses, and is regulated by a complex network including abscisic acid (ABA) signaling and ion-flux-induced turgor changes. The slow-type anion channel SLAC1 has been identified to be a central controller of stomatal closure and phosphoactivated by several kinases. Here, we report the structure of SLAC1 in Arabidopsis thaliana (AtSLAC1) in an inactivated, closed state. The cytosolic amino (N)-terminus and carboxyl (C)-terminus of AtSLAC1 are partially resolved and form a plug-like structure which packs against the transmembrane domain (TMD). Breaking the interactions between the cytosolic plug and transmembrane domain triggers channel activation. An inhibition-release model is proposed for SLAC1 activation by phosphorylation that the cytosolic plug dissociates from the transmembrane domain upon phosphorylation, and induces conformational changes to open the pore. These findings facilitate our understanding of the regulation of SLAC1 activity and stomatal aperture in plants. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_32633.map.gz emd_32633.map.gz | 59.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-32633-v30.xml emd-32633-v30.xml emd-32633.xml emd-32633.xml | 16 KB 16 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_32633_fsc.xml emd_32633_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_32633.png emd_32633.png | 106.3 KB | ||
| Filedesc metadata |  emd-32633.cif.gz emd-32633.cif.gz | 5.7 KB | ||
| その他 |  emd_32633_half_map_1.map.gz emd_32633_half_map_1.map.gz emd_32633_half_map_2.map.gz emd_32633_half_map_2.map.gz | 59 MB 59 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-32633 http://ftp.pdbj.org/pub/emdb/structures/EMD-32633 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32633 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32633 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_32633_validation.pdf.gz emd_32633_validation.pdf.gz | 763.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_32633_full_validation.pdf.gz emd_32633_full_validation.pdf.gz | 763.1 KB | 表示 | |
| XML形式データ |  emd_32633_validation.xml.gz emd_32633_validation.xml.gz | 16.4 KB | 表示 | |
| CIF形式データ |  emd_32633_validation.cif.gz emd_32633_validation.cif.gz | 21.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32633 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32633 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32633 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32633 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7wnqMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_32633.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_32633.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM map of the AtSLAC1 S59A mutant | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half map 2
| ファイル | emd_32633_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_32633_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Trimeric structure of the anion channel SLAC1
| 全体 | 名称: Trimeric structure of the anion channel SLAC1 |
|---|---|
| 要素 |
|
-超分子 #1: Trimeric structure of the anion channel SLAC1
| 超分子 | 名称: Trimeric structure of the anion channel SLAC1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Guard cell S-type anion channel SLAC1
| 分子 | 名称: Guard cell S-type anion channel SLAC1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 63.311742 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MERKQSNAHS TFADINEVED EAEQELQQQE NNNNKRFSGN RGPNRGKQRP FRGFSRQVSL ETGFSVLNRE SRERDDKKSL PRSGRSFGG FESGGIINGG DGRKTDFSMF RTKSTLSKQK SLLPSIIRER DIENSLRTED GETKDDSINE NVSAGRYFAA L RGPELDEV ...文字列: MERKQSNAHS TFADINEVED EAEQELQQQE NNNNKRFSGN RGPNRGKQRP FRGFSRQVSL ETGFSVLNRE SRERDDKKSL PRSGRSFGG FESGGIINGG DGRKTDFSMF RTKSTLSKQK SLLPSIIRER DIENSLRTED GETKDDSINE NVSAGRYFAA L RGPELDEV KDNEDILLPK EEQWPFLLRF PIGCFGICLG LSSQAVLWLA LAKSPATNFL HITPLINLVV WLFSLVVLVS VS FTYILKC IFYFEAVKRE YFHPVRVNFF FAPWVVCMFL AISVPPMFSP NRKYLHPAIW CVFMGPYFFL ELKIYGQWLS GGK RRLCKV ANPSSHLSVV GNFVGAILAS KVGWDEVAKF LWAVGFAHYL VVFVTLYQRL PTSEALPKEL HPVYSMFIAA PSAA SIAWN TIYGQFDGCS RTCFFIALFL YISLVARINF FTGFKFSVAW WSYTFPMTTA SVATIKYAEA VPGYPSRALA LTLSF ISTA MVCVLFVSTL LHAFVWQTLF PNDLAIAITK RKLTREKKPF KRAYDLKRWT KQALAKKISA EKDFEAEEES HH UniProtKB: Guard cell S-type anion channel SLAC1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 10 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 281.0 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X