+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The structure of PldA-PA3488 complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報plasma membrane region / phospholipase D activity / phospholipid catabolic process / regulation of vesicle-mediated transport / intracellular membrane-bounded organelle / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |   Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌) Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌) | |||||||||
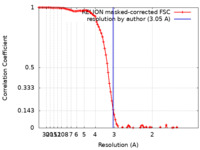
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.05 Å | |||||||||
 データ登録者 データ登録者 | Zhao L / Yang XY / Li ZQ | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structural insights into PA3488-mediated inactivation of Pseudomonas aeruginosa PldA. 著者: Xiaoyun Yang / Zongqiang Li / Liang Zhao / Zhun She / Zengqiang Gao / Sen-Fang Sui / Yuhui Dong / Yanhua Li /  要旨: PldA, a phospholipase D (PLD) effector, catalyzes hydrolysis of the phosphodiester bonds of glycerophospholipids-the main component of cell membranes-and assists the invasion of the opportunistic ...PldA, a phospholipase D (PLD) effector, catalyzes hydrolysis of the phosphodiester bonds of glycerophospholipids-the main component of cell membranes-and assists the invasion of the opportunistic pathogen Pseudomonas aeruginosa. As a cognate immunity protein, PA3488 can inhibit the activity of PldA to avoid self-toxicity. However, the precise inhibitory mechanism remains elusive. We determine the crystal structures of full-length and truncated PldA and the cryogenic electron microscopy structure of the PldA-PA3488 complex. Structural analysis reveals that there are different intermediates of PldA between the "open" and "closed" states of the catalytic pocket, accompanied by significant conformational changes in the "lid" region and the peripheral helical domain. Through structure-based mutational analysis, we identify the key residues responsible for the enzymatic activity of PldA. Together, these data provide an insight into the molecular mechanisms of PldA invasion and its neutralization by PA3488, aiding future design of PLD-targeted inhibitors and drugs. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_32438.map.gz emd_32438.map.gz | 85.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-32438-v30.xml emd-32438-v30.xml emd-32438.xml emd-32438.xml | 11.8 KB 11.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_32438_fsc.xml emd_32438_fsc.xml | 9.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_32438.png emd_32438.png | 50.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-32438 http://ftp.pdbj.org/pub/emdb/structures/EMD-32438 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32438 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32438 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_32438_validation.pdf.gz emd_32438_validation.pdf.gz | 475.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_32438_full_validation.pdf.gz emd_32438_full_validation.pdf.gz | 474.8 KB | 表示 | |
| XML形式データ |  emd_32438_validation.xml.gz emd_32438_validation.xml.gz | 11.1 KB | 表示 | |
| CIF形式データ |  emd_32438_validation.cif.gz emd_32438_validation.cif.gz | 14.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32438 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32438 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32438 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32438 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7wdkMC  7v53C  7v55C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_32438.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_32438.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.668 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : The structure of PldA-PA3488 complex
| 全体 | 名称: The structure of PldA-PA3488 complex |
|---|---|
| 要素 |
|
-超分子 #1: The structure of PldA-PA3488 complex
| 超分子 | 名称: The structure of PldA-PA3488 complex / タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
-分子 #1: Phospholipase D
| 分子 | 名称: Phospholipase D / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌) Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌)株: ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1 |
| 分子量 | 理論値: 122.478062 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MLQKKPYNGL HEKELNQINQ QDGSPCVAIS APGCFIKGSN LFSEKRAGNR VRFFTTGRDY FSDLASALDS ASSSIFITGW QVNYDVLLD GRRSLWQCLR QALERSPALK VYVMPWLSPS GSLGTYDFET MLAVFQLNAG LEGGARAFCT PAIQQSDMQG L GVAFSHHQ ...文字列: MLQKKPYNGL HEKELNQINQ QDGSPCVAIS APGCFIKGSN LFSEKRAGNR VRFFTTGRDY FSDLASALDS ASSSIFITGW QVNYDVLLD GRRSLWQCLR QALERSPALK VYVMPWLSPS GSLGTYDFET MLAVFQLNAG LEGGARAFCT PAIQQSDMQG L GVAFSHHQ KSVVIDNRIG YVGGIDLAYG RRDDNDFSLD ASGRRGNDAY NPGLPHLGWM AEDEHVSSMG LMMATLFDLS RP LASLTLH APTLRLSPFP HIAASDEPLL SIPLAPSRAR ALNGAAYLSD LFRSPMLPSL QWLGRAYNSS KEGLDEGFER LDA LRRQMV ASSIRAIANL IADNLDALPI EPELERRLRA WLEELRTAAL NLPEALRIKS LLLINQWMSE TELGQVLTLI SGKG FEDIP QNLSGKAGEL AGSLFWTLHR LMQARAGGHQ QPYRYLDEAP QPLASPDNAR LAADQPRMPW QDVHCRIEGP SVYDL ARNF IDRWNGQQAY LAKTPALQDT ALVRSALEAV MKWLNSLAAA AGLENYLDEK RNLRLELDPP TPCWINAPEQ LPQEPE VRR GGMTVQVLRS AAARMLEQEQ AGRLGAGVNL PLQVGVSTEG VQSNCKDAML LAISGAQQFI YIENQFFQSE FGKEGEV FK DLPLSGPMAS LRDVGSLRRD FVVRIRLEEA LEQRDLWLLD WAEVEKIAQE PGTEARQFLK SMLAMWGVNA QGWLTHKL G EAQHGLLNEI GEALARRIER AIQREHPFHV YLVLPVHPEG ALNVPNIMHQ VHLTQQSLVF GEQSLVKRIQ RQMALKALE GKSDPAQARE IIERKDARGR PVYEQQDWSR YLTLLNLRTW AVLGGRVVTE QIYVHSKLLI ADDRVAILGS ANINDRSLQG ERDSELAVM VRDSEPLTVR LDGKNDAIVG KAIHQLRVNL WKKHFGLSQG PGGFVKPASE LSAYLSIPAA QEAWEAIQTL A KENTRAYE RTFNFIPQNI SQTQLQLTPE PPKGFEDGFP ASIWPTWAYR KPGELRAGGQ LMEPMPYQEI FWRSSNLTSV KT FPPPNGV SGFITALPTS WTRGERNDSG LNLSILAHQD SRSLPTQVAM NGDSSAQGKH RT |
-分子 #2: Tli4_C domain-containing protein
| 分子 | 名称: Tli4_C domain-containing protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 43.088637 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKRVLMGLIL LSSSNITWAE APSSKYQECL GRMTFEIPEE MEWATYDASR VWQISKGGGH NFTAEVTAVG DNGSYDYDSM IFYVSEKVD KNEFHNASNY IKGTAEIYQD HLRENIKLDK KAISTLQKNK SEEKSIERIK KGIAEMEAKI PLAKIYEHDL G IPDSHILG ...文字列: MKRVLMGLIL LSSSNITWAE APSSKYQECL GRMTFEIPEE MEWATYDASR VWQISKGGGH NFTAEVTAVG DNGSYDYDSM IFYVSEKVD KNEFHNASNY IKGTAEIYQD HLRENIKLDK KAISTLQKNK SEEKSIERIK KGIAEMEAKI PLAKIYEHDL G IPDSHILG SKNIPFHVLL WRNQRVYYFT FSKPTENSAQ RIKDLIARFR TRELYEVPNE PGICFPYGFI ADDGKTAYEL KN SLRFTRT PNVIFSLLTA SANDPWQTRP TSGLYDSDFR PGYDRQKWKK SALLDSLHIG KRLAAFEGWR LDPRPDSGER ERA WFGLAH TGGTLDPLVA IQVQTFQKGT DDLTDYTPPP EEVLPRLKAL SQSIEQRLAR |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー