+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
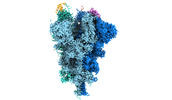
| タイトル | Cryo-EM structure of the SARS-CoV-2 spike protein (2-up RBD) bound to neutralizing nanobodies P17 | |||||||||||||||||||||||||||
 マップデータ マップデータ | ||||||||||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||||||||
 キーワード キーワード | spike / nanobody / VHH / VIRAL PROTEIN | |||||||||||||||||||||||||||
| 生物種 |   | |||||||||||||||||||||||||||
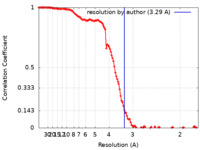
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.29 Å | |||||||||||||||||||||||||||
 データ登録者 データ登録者 | Maeda R / Fujita J / Konishi Y / Kazuma Y / Yamazaki H / Anzai I / Yamaguchi K / Kasai K / Nagata K / Yamaoka Y ...Maeda R / Fujita J / Konishi Y / Kazuma Y / Yamazaki H / Anzai I / Yamaguchi K / Kasai K / Nagata K / Yamaoka Y / Miyakawa K / Ryo A / Shirakawa K / Makino F / Matsuura Y / Inoue T / Imura A / Namba K / Takaori-Kondo A | |||||||||||||||||||||||||||
| 資金援助 |  日本, 8件 日本, 8件
| |||||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2022 ジャーナル: Commun Biol / 年: 2022タイトル: A panel of nanobodies recognizing conserved hidden clefts of all SARS-CoV-2 spike variants including Omicron. 著者: Ryota Maeda / Junso Fujita / Yoshinobu Konishi / Yasuhiro Kazuma / Hiroyuki Yamazaki / Itsuki Anzai / Tokiko Watanabe / Keishi Yamaguchi / Kazuki Kasai / Kayoko Nagata / Yutaro Yamaoka / Kei ...著者: Ryota Maeda / Junso Fujita / Yoshinobu Konishi / Yasuhiro Kazuma / Hiroyuki Yamazaki / Itsuki Anzai / Tokiko Watanabe / Keishi Yamaguchi / Kazuki Kasai / Kayoko Nagata / Yutaro Yamaoka / Kei Miyakawa / Akihide Ryo / Kotaro Shirakawa / Kei Sato / Fumiaki Makino / Yoshiharu Matsuura / Tsuyoshi Inoue / Akihiro Imura / Keiichi Namba / Akifumi Takaori-Kondo /  要旨: We are amid the historic coronavirus infectious disease 2019 (COVID-19) pandemic. Imbalances in the accessibility of vaccines, medicines, and diagnostics among countries, regions, and populations, ...We are amid the historic coronavirus infectious disease 2019 (COVID-19) pandemic. Imbalances in the accessibility of vaccines, medicines, and diagnostics among countries, regions, and populations, and those in war crises, have been problematic. Nanobodies are small, stable, customizable, and inexpensive to produce. Herein, we present a panel of nanobodies that can detect the spike proteins of five SARS-CoV-2 variants of concern (VOCs) including Omicron. Here we show via ELISA, lateral flow, kinetic, flow cytometric, microscopy, and Western blotting assays that our nanobodies can quantify the spike variants. This panel of nanobodies broadly neutralizes viral infection caused by pseudotyped and authentic SARS-CoV-2 VOCs. Structural analyses show that the P86 clone targets epitopes that are conserved yet unclassified on the receptor-binding domain (RBD) and contacts the N-terminal domain (NTD). Human antibodies rarely access both regions; consequently, the clone buries hidden crevasses of SARS-CoV-2 spike proteins that go undetected by conventional antibodies. | |||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
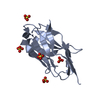
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_32081.map.gz emd_32081.map.gz | 168 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-32081-v30.xml emd-32081-v30.xml emd-32081.xml emd-32081.xml | 23.1 KB 23.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_32081_fsc.xml emd_32081_fsc.xml | 12.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_32081.png emd_32081.png | 56.2 KB | ||
| マスクデータ |  emd_32081_msk_1.map emd_32081_msk_1.map | 178 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-32081.cif.gz emd-32081.cif.gz | 6.6 KB | ||
| その他 |  emd_32081_half_map_1.map.gz emd_32081_half_map_1.map.gz emd_32081_half_map_2.map.gz emd_32081_half_map_2.map.gz | 165.3 MB 165.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-32081 http://ftp.pdbj.org/pub/emdb/structures/EMD-32081 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32081 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32081 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_32081_validation.pdf.gz emd_32081_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_32081_full_validation.pdf.gz emd_32081_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_32081_validation.xml.gz emd_32081_validation.xml.gz | 20.9 KB | 表示 | |
| CIF形式データ |  emd_32081_validation.cif.gz emd_32081_validation.cif.gz | 26.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32081 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32081 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32081 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32081 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_32081.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_32081.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.874 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_32081_msk_1.map emd_32081_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
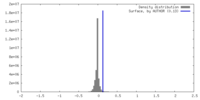
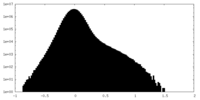
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_32081_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
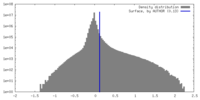
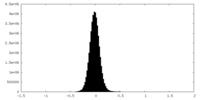
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_32081_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
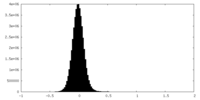
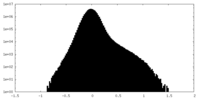
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SARS-CoV-2 spike protein (2-up RBD) bound to neutralizing nanobod...
| 全体 | 名称: SARS-CoV-2 spike protein (2-up RBD) bound to neutralizing nanobodies P17 |
|---|---|
| 要素 |
|
-超分子 #1: SARS-CoV-2 spike protein (2-up RBD) bound to neutralizing nanobod...
| 超分子 | 名称: SARS-CoV-2 spike protein (2-up RBD) bound to neutralizing nanobodies P17 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #2: SARS-CoV-2 spike protein (2-up RBD)
| 超分子 | 名称: SARS-CoV-2 spike protein (2-up RBD) / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: neutralizing nanobodies P17
| 超分子 | 名称: neutralizing nanobodies P17 / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|
-分子 #1: Spike glycoprotein
| 分子 | 名称: Spike glycoprotein / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MFVFLVLLPL VSSQCVNLTT RTQLPPAYTN SFTRGVYYPD KVFRSSVLHS TQDLFLPFFS NVTWFHAIHV SGTNGTKRFD NPVLPFNDGV YFASTEKSNI IRGWIFGTTL DSKTQSLLIV NNATNVVIKV CEFQFCNDPF LGVYYHKNNK SWMESEFRVY SSANNCTFEY ...文字列: MFVFLVLLPL VSSQCVNLTT RTQLPPAYTN SFTRGVYYPD KVFRSSVLHS TQDLFLPFFS NVTWFHAIHV SGTNGTKRFD NPVLPFNDGV YFASTEKSNI IRGWIFGTTL DSKTQSLLIV NNATNVVIKV CEFQFCNDPF LGVYYHKNNK SWMESEFRVY SSANNCTFEY VSQPFLMDLE GKQGNFKNLR EFVFKNIDGY FKIYSKHTPI NLVRDLPQGF SALEPLVDLP IGINITRFQT LLALHRSYLT PGDSSSGWTA GAAAYYVGYL QPRTFLLKYN ENGTITDAVD CALDPLSETK CTLKSFTVEK GIYQTSNFRV QPTESIVRFP NITNLCPFGE VFNATRFASV YAWNRKRISN CVADYSVLYN SASFSTFKCY GVSPTKLNDL CFTNVYADSF VIRGDEVRQI APGQTGKIAD YNYKLPDDFT GCVIAWNSNN LDSKVGGNYN YLYRLFRKSN LKPFERDIST EIYQAGSTPC NGVEGFNCYF PLQSYGFQPT NGVGYQPYRV VVLSFELLHA PATVCGPKKS TNLVKNKCVN FNFNGLTGTG VLTESNKKFL PFQQFGRDIA DTTDAVRDPQ TLEILDITPC SFGGVSVITP GTNTSNQVAV LYQGVNCTEV PVAIHADQLT PTWRVYSTGS NVFQTRAGCL IGAEHVNNSY ECDIPIGAGI CASYQTQTNS PGSASSVASQ SIIAYTMSLG AENSVAYSNN SIAIPTNFTI SVTTEILPVS MTKTSVDCTM YICGDSTECS NLLLQYGSFC TQLNRALTGI AVEQDKNTQE VFAQVKQIYK TPPIKDFGGF NFSQILPDPS KPSKRSFIED LLFNKVTLAD AGFIKQYGDC LGDIAARDLI CAQKFNGLTV LPPLLTDEMI AQYTSALLAG TITSGWTFGA GAALQIPFAM QMAYRFNGIG VTQNVLYENQ KLIANQFNSA IGKIQDSLSS TASALGKLQD VVNQNAQALN TLVKQLSSNF GAISSVLNDI LSRLDPPEAE VQIDRLITGR LQSLQTYVTQ QLIRAAEIRA SANLAATKMS ECVLGQSKRV DFCGKGYHLM SFPQSAPHGV VFLHVTYVPA QEKNFTTAPA ICHDGKAHFP REGVFVSNGT HWFVTQRNFY EPQIITTDNT FVSGNCDVVI GIVNNTVYDP LQPELDSFKE ELDKYFKNHT SPDVDLGDIS GINASVVNIQ KEIDRLNEVA KNLNESLIDL QELGKYEQGS GYIPEAPRDG QAYVRKDGEW VLLSTFLGSH HHHHHHH GENBANK: GENBANK: QHD43416.1 |
-分子 #2: Neutralizing nanobody P17
| 分子 | 名称: Neutralizing nanobody P17 / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QVQLQLQESG GGLVQAGGSL RLSCAASGRT SSVYNMAWFR QTPGKEREFV AAITGNGGTT LYADSVKGRL TISRGNAKNT VSLQMNVLKP DDTAVYYCAA GGWGKERNYA YWGQGTQVTV SSHHHHHH |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL | ||||||
|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 / 構成要素:
| ||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 200 / 支持フィルム - 材質: GRAPHENE 詳細: The graphene grid was chemically oxidized and modified. | ||||||
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV | ||||||
| 詳細 | Mixed with 5 times molar excess of P86. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 300 |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: In-column Omega Filter エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 平均露光時間: 3.0 sec. / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 60000 |
| 試料ステージ | 試料ホルダーモデル: JEOL CRYOSPECPORTER / ホルダー冷却材: NITROGEN |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)