[日本語] English
 万見
万見- EMDB-31827: Cryo-EM structure of E.coli retron-Ec86 (RT-msDNA-RNA) at 3.2 angstrom -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of E.coli retron-Ec86 (RT-msDNA-RNA) at 3.2 angstrom | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Reverse transcriptase / RNA BINDING PROTEIN / TRANSFERASE-DNA-RNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報RNA-directed DNA polymerase / RNA-directed DNA polymerase activity / defense response to virus / RNA binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.12 Å | |||||||||
 データ登録者 データ登録者 | Wang YJ / Guan ZY | |||||||||
| 資金援助 |  中国, 2件 中国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Microbiol / 年: 2022 ジャーナル: Nat Microbiol / 年: 2022タイトル: Cryo-EM structures of Escherichia coli Ec86 retron complexes reveal architecture and defence mechanism. 著者: Yanjing Wang / Zeyuan Guan / Chen Wang / Yangfan Nie / Yibei Chen / Zhaoyang Qian / Yongqing Cui / Han Xu / Qiang Wang / Fen Zhao / Delin Zhang / Pan Tao / Ming Sun / Ping Yin / Shuangxia Jin ...著者: Yanjing Wang / Zeyuan Guan / Chen Wang / Yangfan Nie / Yibei Chen / Zhaoyang Qian / Yongqing Cui / Han Xu / Qiang Wang / Fen Zhao / Delin Zhang / Pan Tao / Ming Sun / Ping Yin / Shuangxia Jin / Shan Wu / Tingting Zou /  要旨: First discovered in the 1980s, retrons are bacterial genetic elements consisting of a reverse transcriptase and a non-coding RNA (ncRNA). Retrons mediate antiphage defence in bacteria but their ...First discovered in the 1980s, retrons are bacterial genetic elements consisting of a reverse transcriptase and a non-coding RNA (ncRNA). Retrons mediate antiphage defence in bacteria but their structure and defence mechanisms are unknown. Here, we investigate the Escherichia coli Ec86 retron and use cryo-electron microscopy to determine the structures of the Ec86 (3.1 Å) and cognate effector-bound Ec86 (2.5 Å) complexes. The Ec86 reverse transcriptase exhibits a characteristic right-hand-like fold consisting of finger, palm and thumb subdomains. Ec86 reverse transcriptase reverse-transcribes part of the ncRNA into satellite, multicopy single-stranded DNA (msDNA, a DNA-RNA hybrid) that we show wraps around the reverse transcriptase electropositive surface. In msDNA, both inverted repeats are present and the 3' sides of the DNA/RNA chains are close to the reverse transcriptase active site. The Ec86 effector adopts a two-lobe fold and directly binds reverse transcriptase and msDNA. These findings offer insights into the structure-function relationship of the retron-effector unit and provide a structural basis for the optimization of retron-based genome editing systems. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
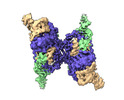
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_31827.map.gz emd_31827.map.gz | 79.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-31827-v30.xml emd-31827-v30.xml emd-31827.xml emd-31827.xml | 13.1 KB 13.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_31827.png emd_31827.png | 92 KB | ||
| Filedesc metadata |  emd-31827.cif.gz emd-31827.cif.gz | 5.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-31827 http://ftp.pdbj.org/pub/emdb/structures/EMD-31827 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31827 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31827 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_31827_validation.pdf.gz emd_31827_validation.pdf.gz | 485.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_31827_full_validation.pdf.gz emd_31827_full_validation.pdf.gz | 484.7 KB | 表示 | |
| XML形式データ |  emd_31827_validation.xml.gz emd_31827_validation.xml.gz | 6.2 KB | 表示 | |
| CIF形式データ |  emd_31827_validation.cif.gz emd_31827_validation.cif.gz | 7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31827 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31827 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31827 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31827 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7v9uMC  7xjgC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_31827.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_31827.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.85 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : retron-Ec86 (RT-msDNA-RNA)
| 全体 | 名称: retron-Ec86 (RT-msDNA-RNA) |
|---|---|
| 要素 |
|
-超分子 #1: retron-Ec86 (RT-msDNA-RNA)
| 超分子 | 名称: retron-Ec86 (RT-msDNA-RNA) / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: RNA-directed DNA polymerase from retron EC86
| 分子 | 名称: RNA-directed DNA polymerase from retron EC86 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: RNA-directed DNA polymerase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 36.484641 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKSAEYLNTF RLRNLGLPVM NNLHDMSKAT RISVETLRLL IYTADFRYRI YTVEKKGPEK RMRTIYQPSR ELKALQGWVL RNILDKLSS SPFSIGFEKH QSILNNATPH IGANFILNID LEDFFPSLTA NKVFGVFHSL GYNRLISSVL TKICCYKNLL P QGAPSSPK ...文字列: MKSAEYLNTF RLRNLGLPVM NNLHDMSKAT RISVETLRLL IYTADFRYRI YTVEKKGPEK RMRTIYQPSR ELKALQGWVL RNILDKLSS SPFSIGFEKH QSILNNATPH IGANFILNID LEDFFPSLTA NKVFGVFHSL GYNRLISSVL TKICCYKNLL P QGAPSSPK LANLICSKLD YRIQGYAGSR GLIYTRYADD LTLSAQSMKK VVKARDFLFS IIPSEGLVIN SKKTCISGPR SQ RKVTGLV ISQEKVGIGR EKYKEIRAKI HHIFCGKSSE IEHVRGWLSF ILSVDSKSHR RLITYISKLE KKYGKNPLNK AKT UniProtKB: Retron Ec86 reverse transcriptase |
-分子 #2: DNA (105-MER)
| 分子 | 名称: DNA (105-MER) / タイプ: dna / ID: 2 / コピー数: 2 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 32.468803 KDa |
| 配列 | 文字列: (DG)(DA)(DA)(DA)(DG)(DT)(DT)(DG)(DC)(DG) (DC)(DA)(DC)(DC)(DC)(DT)(DT)(DA)(DC)(DG) (DT)(DC)(DA)(DG)(DA)(DA)(DA)(DA)(DA) (DA)(DC)(DG)(DG)(DG)(DT)(DT)(DT)(DC)(DC) (DT) (DG)(DG)(DT)(DT)(DG) ...文字列: (DG)(DA)(DA)(DA)(DG)(DT)(DT)(DG)(DC)(DG) (DC)(DA)(DC)(DC)(DC)(DT)(DT)(DA)(DC)(DG) (DT)(DC)(DA)(DG)(DA)(DA)(DA)(DA)(DA) (DA)(DC)(DG)(DG)(DG)(DT)(DT)(DT)(DC)(DC) (DT) (DG)(DG)(DT)(DT)(DG)(DG)(DC)(DT) (DC)(DG)(DG)(DA)(DG)(DA)(DG)(DC)(DA)(DT) (DC)(DA) (DG)(DG)(DC)(DG)(DA)(DT)(DG) (DC)(DT)(DC)(DT)(DC)(DC)(DG)(DT)(DT)(DC) (DC)(DA)(DA) (DC)(DA)(DA)(DG)(DG)(DA) (DA)(DA)(DA)(DC)(DA)(DG)(DA)(DC)(DA)(DG) (DT)(DA)(DA)(DC) (DT)(DC)(DA)(DG)(DA) GENBANK: GENBANK: M24408.1 |
-分子 #3: RNA (81-MER)
| 分子 | 名称: RNA (81-MER) / タイプ: rna / ID: 3 / コピー数: 2 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 25.870203 KDa |
| 配列 | 文字列: UGCGCACCCU UAGCGAGAGG UUUAUCAUUA AGGUCAACCU CUGGAUGUUG UUUCGGCAUC CUGCAUUGAA UCUGAGUUAC U GENBANK: GENBANK: M24408.1 |
-分子 #4: RNA (5'-R(P*CP*GP*UP*AP*AP*GP*GP*G)-3')
| 分子 | 名称: RNA (5'-R(P*CP*GP*UP*AP*AP*GP*GP*G)-3') / タイプ: rna / ID: 4 / コピー数: 2 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 4.541771 KDa |
| 配列 | 文字列: CGUAAGGGUG CGCA |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.4 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 53.68 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: RANDOM CONICAL TILT |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C2 (2回回転対称) / 解像度のタイプ: BY AUTHOR / 解像度: 3.12 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 144586 |
| 初期 角度割当 | タイプ: PROJECTION MATCHING |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. 3.1) |
 ムービー
ムービー コントローラー
コントローラー



 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)




















