+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 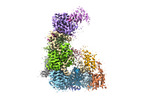 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the Dicer-2-R2D2 heterodimer | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | RNA binding protein / Ribonuclease / Double-stranded RNA-binding protein | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報follicle cell of egg chamber stalk formation / lncRNA catabolic process / : / positive regulation of Toll signaling pathway / regulatory ncRNA processing / MicroRNA (miRNA) biogenesis / Small interfering RNA (siRNA) biogenesis / PKR-mediated signaling / regulation of regulatory ncRNA processing / dsRNA transport ...follicle cell of egg chamber stalk formation / lncRNA catabolic process / : / positive regulation of Toll signaling pathway / regulatory ncRNA processing / MicroRNA (miRNA) biogenesis / Small interfering RNA (siRNA) biogenesis / PKR-mediated signaling / regulation of regulatory ncRNA processing / dsRNA transport / dosage compensation by hyperactivation of X chromosome / global gene silencing by mRNA cleavage / ribonuclease III / apoptotic DNA fragmentation / RISC-loading complex / deoxyribonuclease I activity / detection of virus / RISC complex assembly / ribonuclease III activity / regulatory ncRNA-mediated post-transcriptional gene silencing / siRNA binding / ATP-dependent activity, acting on RNA / siRNA processing / positive regulation of innate immune response / RISC complex / positive regulation of defense response to virus by host / heterochromatin formation / helicase activity / locomotory behavior / mRNA 3'-UTR binding / cellular response to virus / cytoplasmic ribonucleoprotein granule / double-stranded RNA binding / defense response to virus / perinuclear region of cytoplasm / ATP hydrolysis activity / RNA binding / ATP binding / nucleus / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
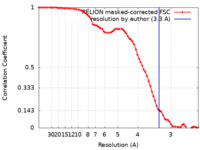
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Yamaguchi S / Nishizawa T / Kusakizako T / Yamashita K / Tomita A / Hirano H / Nishimasu H / Nureki O | |||||||||
| 資金援助 |  日本, 1件 日本, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2022 ジャーナル: Nature / 年: 2022タイトル: Structure of the Dicer-2-R2D2 heterodimer bound to a small RNA duplex. 著者: Sonomi Yamaguchi / Masahiro Naganuma / Tomohiro Nishizawa / Tsukasa Kusakizako / Yukihide Tomari / Hiroshi Nishimasu / Osamu Nureki /  要旨: In flies, Argonaute2 (Ago2) and small interfering RNA (siRNA) form an RNA-induced silencing complex to repress viral transcripts. The RNase III enzyme Dicer-2 associates with its partner protein R2D2 ...In flies, Argonaute2 (Ago2) and small interfering RNA (siRNA) form an RNA-induced silencing complex to repress viral transcripts. The RNase III enzyme Dicer-2 associates with its partner protein R2D2 and cleaves long double-stranded RNAs to produce 21-nucleotide siRNA duplexes, which are then loaded into Ago2 in a defined orientation. Here we report cryo-electron microscopy structures of the Dicer-2-R2D2 and Dicer-2-R2D2-siRNA complexes. R2D2 interacts with the helicase domain and the central linker of Dicer-2 to inhibit the promiscuous processing of microRNA precursors by Dicer-2. Notably, our structure represents the strand-selection state in the siRNA-loading process, and reveals that R2D2 asymmetrically recognizes the end of the siRNA duplex with the higher base-pairing stability, and the other end is exposed to the solvent and is accessible by Ago2. Our findings explain how R2D2 senses the thermodynamic asymmetry of the siRNA and facilitates the siRNA loading into Ago2 in a defined orientation, thereby determining which strand of the siRNA duplex is used by Ago2 as the guide strand for target silencing. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_31741.map.gz emd_31741.map.gz | 2.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-31741-v30.xml emd-31741-v30.xml emd-31741.xml emd-31741.xml | 16.1 KB 16.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_31741_fsc.xml emd_31741_fsc.xml | 7.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_31741.png emd_31741.png | 66 KB | ||
| マスクデータ |  emd_31741_msk_1.map emd_31741_msk_1.map | 20.8 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-31741.cif.gz emd-31741.cif.gz | 6.6 KB | ||
| その他 |  emd_31741_half_map_1.map.gz emd_31741_half_map_1.map.gz emd_31741_half_map_2.map.gz emd_31741_half_map_2.map.gz | 17.6 MB 17.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-31741 http://ftp.pdbj.org/pub/emdb/structures/EMD-31741 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31741 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31741 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_31741_validation.pdf.gz emd_31741_validation.pdf.gz | 793 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_31741_full_validation.pdf.gz emd_31741_full_validation.pdf.gz | 792.6 KB | 表示 | |
| XML形式データ |  emd_31741_validation.xml.gz emd_31741_validation.xml.gz | 13.3 KB | 表示 | |
| CIF形式データ |  emd_31741_validation.cif.gz emd_31741_validation.cif.gz | 16.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31741 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31741 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31741 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31741 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7v6bMC  7v6cC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
| 電子顕微鏡画像生データ |  EMPIAR-11099 (タイトル: Structure of the Dicer-2-R2D2 heterodimer bound to a small RNA duplex EMPIAR-11099 (タイトル: Structure of the Dicer-2-R2D2 heterodimer bound to a small RNA duplexData size: 1.4 TB Data #1: Structure of the Dicer-2-R2D2 heterodimer [micrographs - multiframe] Data #2: Structure of the Dicer-2-R2D2 heterodimer bound to small RNA duplex [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_31741.map.gz / 形式: CCP4 / 大きさ: 20.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_31741.map.gz / 形式: CCP4 / 大きさ: 20.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.245 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_31741_msk_1.map emd_31741_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_31741_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_31741_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Dicer-2-R2D2
| 全体 | 名称: Dicer-2-R2D2 |
|---|---|
| 要素 |
|
-超分子 #1: Dicer-2-R2D2
| 超分子 | 名称: Dicer-2-R2D2 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Dicer-2, isoform A
| 分子 | 名称: Dicer-2, isoform A / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: deoxyribonuclease I |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 198.134734 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSMEDVEIKP RGYQLRLVDH LTKSNGIVYL PTGSGKTFVA ILVLKRFSQD FDKPIESGGK RALFMCNTVE LARQQAMAVR RCTNFKVGF YVGEQGVDDW TRGMWSDEIK KNQVLVGTAQ VFLDMVTQTY VALSSLSVVI IDECHHGTGH HPFREFMRLF T IANQTKLP ...文字列: GSMEDVEIKP RGYQLRLVDH LTKSNGIVYL PTGSGKTFVA ILVLKRFSQD FDKPIESGGK RALFMCNTVE LARQQAMAVR RCTNFKVGF YVGEQGVDDW TRGMWSDEIK KNQVLVGTAQ VFLDMVTQTY VALSSLSVVI IDECHHGTGH HPFREFMRLF T IANQTKLP RVVGLTGVLI KGNEITNVAT KLKELEITYR GNIITVSDTK ELENVMLYAT KPTEVMVSFP HQEQVLTVTR LI SAEIEKF YVSLDLMNIG VQPIRRSKSL QCLRDPSKKS FVKQLFNDFL YQMKEYGIYA ASIAIISLIV EFDIKRRQAE TLS VKLMHR TALTLCEKIR HLLVQKLQDM TYDDDDDNVN TEEVIMNFST PKVQRFLMSL KVSFADKDPK DICCLVFVER RYTC KCIYG LLLNYIQSTP ELRNVLTPQF MVGRNNISPD FESVLERKWQ KSAIQQFRDG NANLMICSSV LEEGIDVQAC NHVFI LDPV KTFNMYVQSK GRARTTEAKF VLFTADKERE KTIQQIYQYR KAHNDIAEYL KDRVLEKTEP ELYEIKGHFQ DDIDPF TNE NGAVLLPNNA LAILHRYCQT IPTDAFGFVI PWFHVLQEDE RDRIFGVSAK GKHVISINMP VNCMLRDTIY SDPMDNV KT AKISAAFKAC KVLYSLGELN ERFVPKTLKE RVASIADVHF EHWNKYGDSV TATVNKADKS KDRTYKTECP LEFYDALP R VGEICYAYEI FLEPQFESCE YTEHMYLNLQ TPRNYAILLR NKLPRLAEMP LFSNQGKLHV RVANAPLEVI IQNSEQLEL LHQFHGMVFR DILKIWHPFF VLDRRSKENS YLVVPLILGA GEQKCFDWEL MTNFRRLPQS HGSNVQQREQ QPAPRPEDFE GKIVTQWYA NYDKPMLVTK VHRELTPLSY MEKNQQDKTY YEFTMSKYGN RIGDVVHKDK FMIEVRDLTE QLTFYVHNRG K FNAKSKAK MKVILIPELC FNFNFPGDLW LKLIFLPSIL NRMYFLLHAE ALRKRFNTYL NLHLLPFNGT DYMPRPLEID YS LKRNVDP LGNVIPTEDI EEPKSLLEPM PTKSIEASVA NLEITEFENP WQKYMEPVDL SRNLLSTYPV ELDYYYHFSV GNV CEMNEM DFEDKEYWAK NQFHMPTGNI YGNRTPAKTN ANVPALMPSK PTVRGKVKPL LILQKTVSKE HITPAEQGEF LAAI TASSA ADVFDMERLE ILGDSFLKLS ATLYLASKYS DWNEGTLTEV KSKLVSNRNL LFCLIDADIP KTLNTIQFTP RYTWL PPGI SLPHNVLALW RENPEFAKII GPHNLRDLAL GDEESLVKGN CSDINYNRFV EGCRANGQSF YAGADFSSEV NFCVGL VTI PNKVIADTLE ALLGVIVKNY GLQHAFKMLE YFKICRADID KPLTQLLNLE LGGKKMRANV NTTEIDGFLI NHYYLEK NL GYTFKDRRYL LQALTHPSYP TNRITGSYQE LEFIGDAILD FLISAYIFEN NTKMNPGALT DLRSALVNNT TLACICVR H RLHFFILAEN AKLSEIISKF VNFQESQGHR VTNYVRILLE EADVQPTPLD LDDELDMTEL PHANKCISQE AEKGVPPKG EFNMSTNVDV PKALGDVLEA LIAAVYLDCR DLQRTWEVIF NLFEPELQEF TRKVPINHIR QLVEHKHAKP VFSSPIVEGE TVMVSCQFT CMEKTIKVYG FGSNKDQAKL SAAKHALQQL SKCDA UniProtKB: Endoribonuclease Dcr-2 |
-分子 #2: R2D2
| 分子 | 名称: R2D2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 35.517699 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GPFTMDNKSA VSALQEFCAR TQINLPTYSF IPGEDGGYVC KVELLEIEAL GNGRSKRDAK HLAASNILRK IQLLPGIHGL MKDSTVGDL DEELTNLNRD MVKELRDYCV RREMPLPCIE VVQQSGTPSA PEFVACCSVA SIVRYGKSDK KKDARQRAAI E MLALISSN ...文字列: GPFTMDNKSA VSALQEFCAR TQINLPTYSF IPGEDGGYVC KVELLEIEAL GNGRSKRDAK HLAASNILRK IQLLPGIHGL MKDSTVGDL DEELTNLNRD MVKELRDYCV RREMPLPCIE VVQQSGTPSA PEFVACCSVA SIVRYGKSDK KKDARQRAAI E MLALISSN SDNLRPDQMQ VASTSKLKVV DMEESMEELE ALRRKKFTTY WELKEAGSVD HTGMRLCDRH NYFKNFYPTL KK EAIEAIN SDEYESSKDK AMDVMSSLKI TPKISEVESS SLVPLLSVEL NCAFDVVLMA KETDIYDHII DYFRTMLI UniProtKB: LD06392p |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: NITROGEN |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 53.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
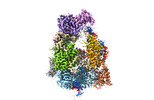
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X