+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of TELO2-TTI1-TTI2 complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | adaptor / CHAPERONE / TELO2 / TTI1 / TTI2 | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of DNA damage checkpoint / TTT Hsp90 cochaperone complex / 'de novo' cotranslational protein folding / TORC2 complex / TORC1 complex / regulation of TOR signaling / telomeric DNA binding / Hsp90 protein binding / chromosome, telomeric region / molecular adaptor activity ...positive regulation of DNA damage checkpoint / TTT Hsp90 cochaperone complex / 'de novo' cotranslational protein folding / TORC2 complex / TORC1 complex / regulation of TOR signaling / telomeric DNA binding / Hsp90 protein binding / chromosome, telomeric region / molecular adaptor activity / nuclear body / protein stabilization / protein-containing complex binding / protein kinase binding / membrane / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.2 Å | |||||||||
 データ登録者 データ登録者 | Cho Y / Kim Y | |||||||||
| 資金援助 |  韓国, 1件 韓国, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2022 ジャーナル: J Mol Biol / 年: 2022タイトル: Structure of the Human TELO2-TTI1-TTI2 Complex. 著者: Youngran Kim / Junhyeon Park / So Young Joo / Byung-Gyu Kim / Aera Jo / Hyunsook Lee / Yunje Cho /  要旨: Phosphatidylinositol 3-kinase-related protein kinases (PIKKs) play critical roles in various metabolic pathways related to cell proliferation and survival. The TELO2-TTI1-TTI2 (TTT) complex has been ...Phosphatidylinositol 3-kinase-related protein kinases (PIKKs) play critical roles in various metabolic pathways related to cell proliferation and survival. The TELO2-TTI1-TTI2 (TTT) complex has been proposed to recognize newly synthesized PIKKs and to deliver them to the R2TP complex (RUVBL1-RUVBL2-RPAP3-PIH1D1) and the heat shock protein 90 chaperone, thereby supporting their folding and assembly. Here, we determined the cryo-EM structure of the TTT complex at an average resolution of 4.2 Å. We describe the full-length structures of TTI1 and TELO2, and a partial structure of TTI2. All three proteins form elongated helical repeat structures. TTI1 provides a platform on which TELO2 and TTI2 bind to its central region and C-terminal end, respectively. The TELO2 C-terminal domain (CTD) is required for the interaction with TTI1 and recruitment of Ataxia-telangiectasia mutated (ATM). The N- and C-terminal segments of TTI1 recognize the FRAP-ATM-TRRAP (FAT) domain and the N-terminal HEAT repeats of ATM, respectively. The TELO2 CTD and TTI1 N- and C-terminal segments are required for cell survival in response to ionizing radiation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_31454.map.gz emd_31454.map.gz | 145.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-31454-v30.xml emd-31454-v30.xml emd-31454.xml emd-31454.xml | 14.3 KB 14.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_31454.png emd_31454.png | 67 KB | ||
| Filedesc metadata |  emd-31454.cif.gz emd-31454.cif.gz | 6.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-31454 http://ftp.pdbj.org/pub/emdb/structures/EMD-31454 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31454 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31454 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_31454_validation.pdf.gz emd_31454_validation.pdf.gz | 337.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_31454_full_validation.pdf.gz emd_31454_full_validation.pdf.gz | 336.7 KB | 表示 | |
| XML形式データ |  emd_31454_validation.xml.gz emd_31454_validation.xml.gz | 6.8 KB | 表示 | |
| CIF形式データ |  emd_31454_validation.cif.gz emd_31454_validation.cif.gz | 7.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31454 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31454 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31454 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31454 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7f4uMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_31454.map.gz / 形式: CCP4 / 大きさ: 163.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_31454.map.gz / 形式: CCP4 / 大きさ: 163.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||
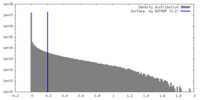
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : chaperone adaptor
| 全体 | 名称: chaperone adaptor |
|---|---|
| 要素 |
|
-超分子 #1: chaperone adaptor
| 超分子 | 名称: chaperone adaptor / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Telomere length regulation protein TEL2 homolog
| 分子 | 名称: Telomere length regulation protein TEL2 homolog / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 91.846883 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MEPAPSEVRL AVREAIHALS SSEDGGHIFC TLESLKRYLG EMEPPALPRE KEEFASAHFS PVLRCLASRL SPAWLELLPH GRLEELWAS FFLEGPADQA FLVLMETIEG AAGPSFRLMK MARLLARFLR EGRLAVLMEA QCRQQTQPGF ILLRETLLGK V VALPDHLG ...文字列: MEPAPSEVRL AVREAIHALS SSEDGGHIFC TLESLKRYLG EMEPPALPRE KEEFASAHFS PVLRCLASRL SPAWLELLPH GRLEELWAS FFLEGPADQA FLVLMETIEG AAGPSFRLMK MARLLARFLR EGRLAVLMEA QCRQQTQPGF ILLRETLLGK V VALPDHLG NRLQQENLAE FFPQNYFRLL GEEVVRVLQA VVDSLQGGLD SSVSFVSQVL GKACVHGRQQ EILGVLVPRL AA LTQGSYL HQRVCWRLVE QVPDRAMEAV LTGLVEAALG PEVLSRLLGN LVVKNKKAQF VMTQKLLFLQ SRLTTPMLQS LLG HLAMDS QRRPLLLQVL KELLETWGSS SAIRHTPLPQ QRHVSKAVLI CLAQLGEPEL RDSRDELLAS MMAGVKCRLD SSLP PVRRL GMIVAEVVSA RIHPEGPPLK FQYEEDELSL ELLALASPQP AGDGASEAGT SLVPATAEPP AETPAEIVDG GVPQA QLAG SDSDLDSDDE FVPYDMSGDR ELKSSKAPAY VRDCVEALTT SEDIERWEAA LRALEGLVYR SPTATREVSV ELAKVL LHL EEKTCVVGFA GLRQRALVAV TVTDPAPVAD YLTSQFYALN YSLRQRMDIL DVLTLAAQEL SRPGCLGRTP QPGSPSP NT PCLPEAAVSQ PGSAVASDWR VVVEERIRSK TQRLSKGGPR QGPAGSPSRF NSVAGHFFFP LLQRFDRPLV TFDLLGED Q LVLGRLAHTL GALMCLAVNT TVAVAMGKAL LEFVWALRFH IDAYVRQGLL SAVSSVLLSL PAARLLEDLM DELLEARSW LADVAEKDPD EDCRTLALRA LLLLQRLKNR LLPPASP UniProtKB: Telomere length regulation protein TEL2 homolog |
-分子 #2: TELO2-interacting protein 1 homolog
| 分子 | 名称: TELO2-interacting protein 1 homolog / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 117.124562 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MHHHHHMAVF DTPEEAFGVL RPVCVQLTKT QTVENVEHLQ TRLQAVSDSA LQELQQYILF PLRFTLKTPG PKRERLIQSV VECLTFVLS STCVKEQELL QELFSELSAC LYSPSSQKPA AVSEELKLAV IQGLSTLMHS AYGDIILTFY EPSILPRLGF A VSLLLGLA ...文字列: MHHHHHMAVF DTPEEAFGVL RPVCVQLTKT QTVENVEHLQ TRLQAVSDSA LQELQQYILF PLRFTLKTPG PKRERLIQSV VECLTFVLS STCVKEQELL QELFSELSAC LYSPSSQKPA AVSEELKLAV IQGLSTLMHS AYGDIILTFY EPSILPRLGF A VSLLLGLA EQE(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)DCQDHP R(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)GDFKQGH SIVVSSLKIF YKTVSFIMAD EQLKRISKVQ AKPAVEHR V AELMVYREAD WVKKTGDKLT ILIKKIIECV SVHPHWKVRL ELVELVEDLL LKCSQSLVEC AGPLLKALVG LVNDESPEI QAQCNKVLRH FADQKVVVGN KALADILSES LHSLATSLPR LMNSQDDQGK FSTLSLLLGY LKLLGPKINF VLNSVAHLQR LSKALIQVL ELDVADIKIV EERRWNSDDL NASPKTSATQ PWNRIQRRYF RFFTDERIFM LLRQVCQLLG YYGNLYLLVD H FMELYHQS VVYRKQAAMI LNELVTGAAG LEVEDLHEKH IKTNPEELRE IVTSILEEYT SQENWYLVTC LETEEMGEEL MM EHPGLQA ITSGEHTCQV TSFLAFSKPS PTICSMNSNI WQICIQLEGI GQFAYALGKD FCLLLMSALY PVLEKAGDQT LLI SQVATS TMMDVCRACG YDSLQHLINQ NSDYLVNGIS LNLRHLALHP HTPKVLEVML RNSDANLLPL VADVVQDVLA TLDQ FYDKR AASFVSVLHA LMAALAQWFP DTGNLGHLQE QSLGEEGSHL NQRPAALEKS TTTAEDIEQF LLNYLKEKDV ADGNV SDFD NEEEEQSVPP KVDENDTRPD VEPPL(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)R LTRD(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)LVTQAP ISARAGPVYS H(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) LNKVADACLI YLSVKQP(UNK)(UNK)(UNK) (UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)GQQNP YTT NVLQLLKELQ UniProtKB: TELO2-interacting protein 1 homolog |
-分子 #3: TELO2-interacting protein 2
| 分子 | 名称: TELO2-interacting protein 2 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 54.944137 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MELDSALEAP SQEDSNLSEE LSHSAFGQAF SKILHCLARP (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) D AVLKDLGDLI EATEF(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK) (UNK)(UNK) ...文字列: MELDSALEAP SQEDSNLSEE LSHSAFGQAF SKILHCLARP (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) D AVLKDLGDLI EATEF(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)VAKA LEKYAA(UNK)(UNK)(UNK)(UNK) (UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)QVG LLF (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)LVG PAWQTGL(UNK) (UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) TPRSREVARE (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)FLH GENEDEKGRL SVILGLLKPD LYKESWKNNP AIKHVFSWTL QQVTRPW LS QHLERVLPAS LVISDDYQTE NKILGVHCLH HIVLNVPAAD LLQYNRAQVL YHAISNHLYT PEHHLIQAVL LCLLDLFP I LEKTLHWKGD GARPTTHCDE VLRLILTHME PEHRLLLRRT YARNLPAFVN RLGILTVRHL KRLERVIIGY LEVYDGPEE EARLKILETL KLLMQHTWPR VSCRLVVLLK ALLKLICDVA RDPNLTPESV KSALLQEATD CLILLDRCSQ GRVKGLLAKI PQSCEDRKV VNYIRKVQQV SEGAPYNGT |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 42.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: DIFFRACTION / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 4.2 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 323679 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)