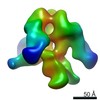
登録情報 データベース : EMDB / ID : EMD-3096タイトル cryo-ET subtomogram averaging of BG505 SOSIP.664 in complex with sCD4, 17b, and 8ANC195 subtomogram-averaging of 1745 sub-volumes. 試料 : soluble HIV-1 Env trimer BG505 SOSIP.664 in complex with soluble CD4s (D1-D2), broadly neutralizing antibody 17b Fabs, and broadly neutralizing antibody 8ANC195 variant G32K5 Fabsタンパク質・ペプチド : Soluble HIV-1 Env trimer BG505 SOSIP.664タンパク質・ペプチド : Fab of broadly neutralizing antibody 17bタンパク質・ペプチド : soluble CD4 (D1-D2 domains)タンパク質・ペプチド : Fab of broadly neutralizing antibody 8ANC195 variant G52K5機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 / Homo sapiens (ヒト)手法 / / 解像度 : 23.0 Å Scharf L / Wang H / Gao H / Chen S / McDowall A / Bjorkman P ジャーナル : Cell / 年 : 2015タイトル : Broadly Neutralizing Antibody 8ANC195 Recognizes Closed and Open States of HIV-1 Env.著者 : Louise Scharf / Haoqing Wang / Han Gao / Songye Chen / Alasdair W McDowall / Pamela J Bjorkman / 要旨 : The HIV-1 envelope (Env) spike contains limited epitopes for broadly neutralizing antibodies (bNAbs); thus, most neutralizing antibodies are strain specific. The 8ANC195 epitope, defined by crystal ... The HIV-1 envelope (Env) spike contains limited epitopes for broadly neutralizing antibodies (bNAbs); thus, most neutralizing antibodies are strain specific. The 8ANC195 epitope, defined by crystal and electron microscopy (EM) structures of bNAb 8ANC195 complexed with monomeric gp120 and trimeric Env, respectively, spans the gp120 and gp41 Env subunits. To investigate 8ANC195's gp41 epitope at higher resolution, we solved a 3.58 Å crystal structure of 8ANC195 complexed with fully glycosylated Env trimer, revealing 8ANC195 insertion into a glycan shield gap to contact gp120 and gp41 glycans and protein residues. To determine whether 8ANC195 recognizes the CD4-bound open Env conformation that leads to co-receptor binding and fusion, one of several known conformations of virion-associated Env, we solved EM structures of an Env/CD4/CD4-induced antibody/8ANC195 complex. 8ANC195 binding partially closed the CD4-bound trimer, confirming structural plasticity of Env by revealing a previously unseen conformation. 8ANC195's ability to bind different Env conformations suggests advantages for potential therapeutic applications. 履歴 登録 2015年7月14日 - ヘッダ(付随情報) 公開 2015年7月29日 - マップ公開 2015年9月23日 - 更新 2015年9月23日 - 現状 2015年9月23日 処理サイト : PDBe / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 機能・相同性情報
機能・相同性情報
 Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /
Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 引用
引用 ジャーナル: Cell / 年: 2015
ジャーナル: Cell / 年: 2015
 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_3096.map.gz
emd_3096.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-3096-v30.xml
emd-3096-v30.xml emd-3096.xml
emd-3096.xml EMDBヘッダ
EMDBヘッダ emd_3096_fsc.xml
emd_3096_fsc.xml FSCデータファイル
FSCデータファイル emd-3096.jpg
emd-3096.jpg http://ftp.pdbj.org/pub/emdb/structures/EMD-3096
http://ftp.pdbj.org/pub/emdb/structures/EMD-3096 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3096
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3096 emd_3096_validation.pdf.gz
emd_3096_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_3096_full_validation.pdf.gz
emd_3096_full_validation.pdf.gz emd_3096_validation.xml.gz
emd_3096_validation.xml.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3096
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3096 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3096
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3096 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_3096.map.gz / 形式: CCP4 / 大きさ: 478.5 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_3096.map.gz / 形式: CCP4 / 大きさ: 478.5 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素
 Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)
Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Homo sapiens (ヒト) / 組換細胞: HEK293-6E / 組換プラスミド: pTT5
Homo sapiens (ヒト) / 組換細胞: HEK293-6E / 組換プラスミド: pTT5 Homo sapiens (ヒト) / 別称: Human
Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 組換細胞: HEK293-6E / 組換プラスミド: pTT5
Homo sapiens (ヒト) / 組換細胞: HEK293-6E / 組換プラスミド: pTT5 Homo sapiens (ヒト) / 別称: Human
Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 組換細胞: HEK293-6E / 組換プラスミド: pTT5
Homo sapiens (ヒト) / 組換細胞: HEK293-6E / 組換プラスミド: pTT5 Homo sapiens (ヒト) / 別称: Human
Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 組換細胞: HEK293-6E / 組換プラスミド: pTT5
Homo sapiens (ヒト) / 組換細胞: HEK293-6E / 組換プラスミド: pTT5 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 画像解析
画像解析 Chimera
Chimera
 ムービー
ムービー コントローラー
コントローラー






























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)
























