+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
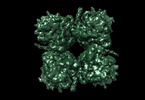
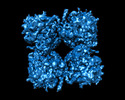
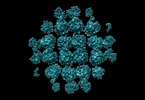
| タイトル | phiPA3 PhuN Tetramer, p2 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Pseudomonas phage PhiPA3 (ファージ) Pseudomonas phage PhiPA3 (ファージ) | |||||||||
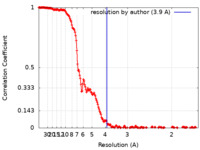
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.9 Å | |||||||||
 データ登録者 データ登録者 | Nieweglowska ES / Brilot AF / Mendez-Moran M / Kokontis C / Baek M / Li J / Cheng Y / Baker D / Bondy-Denomy J / Agard DA | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: The ϕPA3 phage nucleus is enclosed by a self-assembling 2D crystalline lattice. 著者: Eliza S Nieweglowska / Axel F Brilot / Melissa Méndez-Moran / Claire Kokontis / Minkyung Baek / Junrui Li / Yifan Cheng / David Baker / Joseph Bondy-Denomy / David A Agard /  要旨: To protect themselves from host attack, numerous jumbo bacteriophages establish a phage nucleus-a micron-scale, proteinaceous structure encompassing the replicating phage DNA. Bacteriophage and host ...To protect themselves from host attack, numerous jumbo bacteriophages establish a phage nucleus-a micron-scale, proteinaceous structure encompassing the replicating phage DNA. Bacteriophage and host proteins associated with replication and transcription are concentrated inside the phage nucleus while other phage and host proteins are excluded, including CRISPR-Cas and restriction endonuclease host defense systems. Here, we show that nucleus fragments isolated from ϕPA3 infected Pseudomonas aeruginosa form a 2-dimensional lattice, having p2 or p4 symmetry. We further demonstrate that recombinantly purified primary Phage Nuclear Enclosure (PhuN) protein spontaneously assembles into similar 2D sheets with p2 and p4 symmetry. We resolve the dominant p2 symmetric state to 3.9 Å by cryo-EM. Our structure reveals a two-domain core, organized into quasi-symmetric tetramers. Flexible loops and termini mediate adaptable inter-tetramer contacts that drive subunit assembly into a lattice and enable the adoption of different symmetric states. While the interfaces between subunits are mostly well packed, two are open, forming channels that likely have functional implications for the transport of proteins, mRNA, and small molecules. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29550.map.gz emd_29550.map.gz | 473.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29550-v30.xml emd-29550-v30.xml emd-29550.xml emd-29550.xml | 18 KB 18 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_29550_fsc.xml emd_29550_fsc.xml | 23.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_29550.png emd_29550.png | 89.2 KB | ||
| マスクデータ |  emd_29550_msk_1.map emd_29550_msk_1.map | 512 MB |  マスクマップ マスクマップ | |
| その他 |  emd_29550_half_map_1.map.gz emd_29550_half_map_1.map.gz emd_29550_half_map_2.map.gz emd_29550_half_map_2.map.gz | 23 MB 23 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29550 http://ftp.pdbj.org/pub/emdb/structures/EMD-29550 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29550 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29550 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_29550_validation.pdf.gz emd_29550_validation.pdf.gz | 672.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_29550_full_validation.pdf.gz emd_29550_full_validation.pdf.gz | 671.7 KB | 表示 | |
| XML形式データ |  emd_29550_validation.xml.gz emd_29550_validation.xml.gz | 26.2 KB | 表示 | |
| CIF形式データ |  emd_29550_validation.cif.gz emd_29550_validation.cif.gz | 34.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29550 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29550 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29550 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29550 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29550.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29550.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.834 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_29550_msk_1.map emd_29550_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_29550_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_29550_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Core tetramer assembly (p2) of the phiPA3 bacteriophage PhuN protein
| 全体 | 名称: Core tetramer assembly (p2) of the phiPA3 bacteriophage PhuN protein |
|---|---|
| 要素 |
|
-超分子 #1: Core tetramer assembly (p2) of the phiPA3 bacteriophage PhuN protein
| 超分子 | 名称: Core tetramer assembly (p2) of the phiPA3 bacteriophage PhuN protein タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas phage PhiPA3 (ファージ) Pseudomonas phage PhiPA3 (ファージ) |
-分子 #1: Maltose/maltodextrin-binding periplasmic protein, phiPA3 PhuN
| 分子 | 名称: Maltose/maltodextrin-binding periplasmic protein, phiPA3 PhuN タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas phage PhiPA3 (ファージ) Pseudomonas phage PhiPA3 (ファージ) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: HHHHHHMKIE EGKLVIWING DKGYNGLAEV GKKFEKDTGI KVTVEHPDKL EEKFPQVAAT GDGPDIIFWA HDRFGGYAQS GLLAEITPD KAFQDKLYPF TWDAVRYNGK LIAYPIAVEA LSLIYNKDLL PNPPKTWEEI PALDKELKAK GKSALMFNLQ E PYFTWPLI ...文字列: HHHHHHMKIE EGKLVIWING DKGYNGLAEV GKKFEKDTGI KVTVEHPDKL EEKFPQVAAT GDGPDIIFWA HDRFGGYAQS GLLAEITPD KAFQDKLYPF TWDAVRYNGK LIAYPIAVEA LSLIYNKDLL PNPPKTWEEI PALDKELKAK GKSALMFNLQ E PYFTWPLI AADGGYAFKY ENGKYDIKDV GVDNAGAKAG LTFLVDLIKN KHMNADTDYS IAEAAFNKGE TAMTINGPWA WS NIDTSKV NYGVTVLPTF KGQPSKPFVG VLSAGINAAS PNKELAKEFL ENYLLTDEGL EAVNKDKPLG AVALKSYEEE LAK DPRIAA TMENAQKGEI MPNIPQMSAF WYAVRTAVIN AASGRQTVDE ALKDAQTGKP IPNPLLGLDS TENLYFQGMQ QTQQ GPKVQ TQTLQGGAGN LNSIFQRSGR TDGGDARASE ALAVFNKLKE EAIAQQDLHD DFLVFRFDRD QNRVGYSALL VVKRA AING QQVIVTRPLV MPNDQITLPT KKLTIQNGMH QETIEAEADV QDVFTTQYWN RICDSIRQQT GKHDAMVINA GPTVIP ADF DLKDELVLKQ LLIKSVNLCD DMLAKRSGEQ PFSVAMLKGT DETLAARLNF TGKPMHDSLG YPIRSDILVS LNRVKKP GQ QENEFYEAED KLNQVSCFVN LEYTPQPQQA IYGAPQQTQQ LPPLTPAIVI TDVRQAEWLK ANTMELYLFA LSNAFRVT A NQSWARSLLP QLGKVKDMRD IGAIGYLSRL AARVETKTET FTDQNFAELL YNMVRPSPVF MSDLNRFGDN AAIENVFID ALGGVNQQRA VAAIIAGVNN LIGGGFEKFF DHNTMPIIQP YGTDIQLGYY LDGEGEKQDR RDLDVLGALN ASDGNIQEWM SWYGTQCNV AVHPELRARQ SKNFDRQYLG NSVTYTTRAH RGIWNPKFIE ALDKAIASVG LTVAMDNVAQ VFGAQRFSGN L AIADYAVT GTAQVSSGLV SNGGYNPQFG VGQGSGFY |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | 2D array |
- 試料調製
試料調製
| 緩衝液 | pH: 6.5 構成要素:
詳細: 0.25 cOmplete Protease Inhibitor Tablet also included | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 67.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X